
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 6,264,515 – 6,264,697 |
| Length | 182 |
| Max. P | 0.999844 |

| Location | 6,264,515 – 6,264,632 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 94.62 |
| Mean single sequence MFE | -45.00 |
| Consensus MFE | -37.34 |
| Energy contribution | -38.18 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.47 |
| Structure conservation index | 0.83 |
| SVM decision value | 2.35 |
| SVM RNA-class probability | 0.992751 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
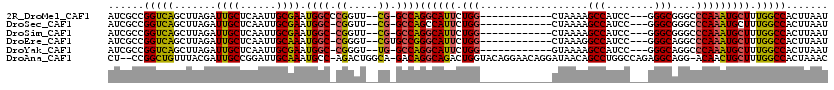
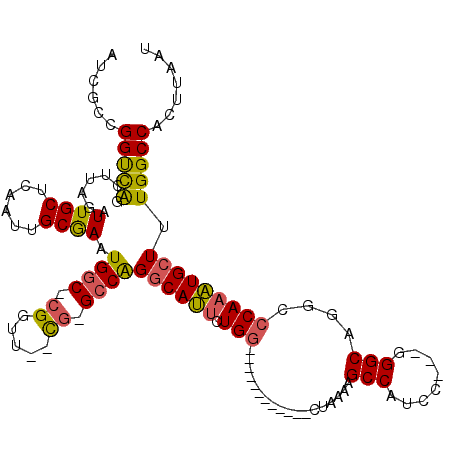
>2R_DroMel_CAF1 6264515 117 + 20766785 AUCACGUCAGUUUAAAGCCCAUUGGCCUGGCCAACUGCCAUACACAAAUGACUGCGAGUGCCAGCGACCACAGGCCACUCAUUAAGUGGCCAAAGCAUUUGGGCCCGCCCGGAUGGC .....(((........(((....)))(((((..((((((((......)))...)).)))))))).)))....(((((((.....)))))))....((((((((....)))))))).. ( -39.60) >DroSec_CAF1 7685 117 + 1 AUCACGGCAGUUUAAAGCCCAUUGAACUGGCCAACUGCCAUACGCAAAUGACUGCGAGUGCCAGCGACCACAGGCCACUCAUUAAGUGGCCAAAGCAUUUGGGCCCGCCCGGAUGGC .((..((((((((((......))))))).)))..(((.(((.((((......)))).))).))).)).....(((((((.....)))))))....((((((((....)))))))).. ( -44.70) >DroSim_CAF1 7901 117 + 1 AUCACGGCAGUUUAAAGCCCGUUGGACUGGCCAACUGCCAUACGCAAAUGACUGCGAGUGCCAGCGACCACAGGCCACUCAUUAAGUGGCCAAAGCAUUUGGGCCCGCCCGGAUGGC .....(((........)))((((((((((((.....)))...((((......))))))).))))))......(((((((.....)))))))....((((((((....)))))))).. ( -47.30) >DroEre_CAF1 7407 117 + 1 AUCACGGCAGUUUGGCGCCCGUUGGCCUGGCCAACUGCCAUACGCAAAUGACUGCGAGUGCCAGCGACCACAGGCCACUCAUUAAGUGGCCAAAGCAUUUGGGCCUGCCCGGAUGGC ..((((((((((.((((((....)))...))))))))))...((((......)))).)))...((.......(((((((.....)))))))....((((((((....)))))))))) ( -51.30) >DroYak_CAF1 8466 117 + 1 AUCAAGACAGUUUAAAGCCCGUUGGUCUGUCCAACUGCCAUACGCAAAUGGCUGCGAGUGCCAGUGACCACAGGCCACUCAUUAAGUGGCCAAAGCAUUUGGGCCUGCCCGGAUGGC .....(((((.((((......)))).)))))..((((.(((.((((......)))).))).)))).......(((((((.....)))))))....((((((((....)))))))).. ( -42.10) >consensus AUCACGGCAGUUUAAAGCCCGUUGGACUGGCCAACUGCCAUACGCAAAUGACUGCGAGUGCCAGCGACCACAGGCCACUCAUUAAGUGGCCAAAGCAUUUGGGCCCGCCCGGAUGGC ..((((((((((....(((.........))).)))))))...((((......)))).)))...((.......(((((((.....)))))))....((((((((....)))))))))) (-37.34 = -38.18 + 0.84)

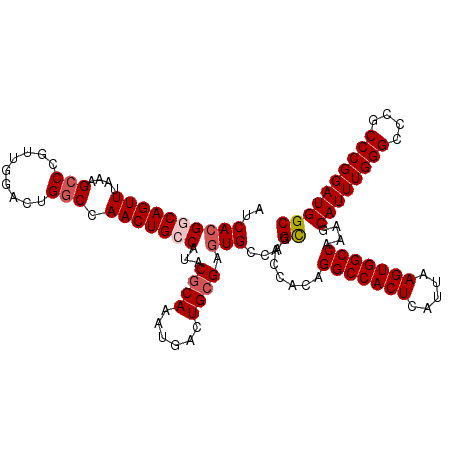
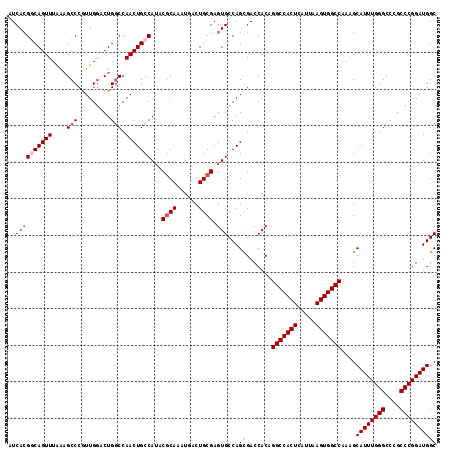
| Location | 6,264,515 – 6,264,632 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 94.62 |
| Mean single sequence MFE | -49.76 |
| Consensus MFE | -46.34 |
| Energy contribution | -46.06 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.47 |
| Structure conservation index | 0.93 |
| SVM decision value | 4.23 |
| SVM RNA-class probability | 0.999844 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 6264515 117 - 20766785 GCCAUCCGGGCGGGCCCAAAUGCUUUGGCCACUUAAUGAGUGGCCUGUGGUCGCUGGCACUCGCAGUCAUUUGUGUAUGGCAGUUGGCCAGGCCAAUGGGCUUUAAACUGACGUGAU (((.....)))(((((((...(((..((((((((...))))))))..(((((((((.((..(((((....)))))..)).))).)))))))))...))))))).............. ( -49.20) >DroSec_CAF1 7685 117 - 1 GCCAUCCGGGCGGGCCCAAAUGCUUUGGCCACUUAAUGAGUGGCCUGUGGUCGCUGGCACUCGCAGUCAUUUGCGUAUGGCAGUUGGCCAGUUCAAUGGGCUUUAAACUGCCGUGAU ........((((((((((........((((((((...))))))))..(((((((((.((..(((((....)))))..)).))).))))))......)))))......)))))..... ( -49.80) >DroSim_CAF1 7901 117 - 1 GCCAUCCGGGCGGGCCCAAAUGCUUUGGCCACUUAAUGAGUGGCCUGUGGUCGCUGGCACUCGCAGUCAUUUGCGUAUGGCAGUUGGCCAGUCCAACGGGCUUUAAACUGCCGUGAU ((((.(((((....)))....((...((((((((...)))))))).))))....))))...(((((....)))))((((((((((....(((((...)))))...)))))))))).. ( -49.80) >DroEre_CAF1 7407 117 - 1 GCCAUCCGGGCAGGCCCAAAUGCUUUGGCCACUUAAUGAGUGGCCUGUGGUCGCUGGCACUCGCAGUCAUUUGCGUAUGGCAGUUGGCCAGGCCAACGGGCGCCAAACUGCCGUGAU ((((.(((((....)))....((...((((((((...)))))))).))))....))))...(((((....)))))(((((((((((((...(((....)))))).)))))))))).. ( -53.40) >DroYak_CAF1 8466 117 - 1 GCCAUCCGGGCAGGCCCAAAUGCUUUGGCCACUUAAUGAGUGGCCUGUGGUCACUGGCACUCGCAGCCAUUUGCGUAUGGCAGUUGGACAGACCAACGGGCUUUAAACUGUCUUGAU ...(((.((((((((((.........((((((((...))))))))..(((((((((.((..(((((....)))))..)).))))......)))))..))))......)))))).))) ( -46.60) >consensus GCCAUCCGGGCGGGCCCAAAUGCUUUGGCCACUUAAUGAGUGGCCUGUGGUCGCUGGCACUCGCAGUCAUUUGCGUAUGGCAGUUGGCCAGGCCAACGGGCUUUAAACUGCCGUGAU ...(((..(((((((((.........((((((((...))))))))..(((((((((.((..(((((....)))))..)).))).)))))).......))))......)))))..))) (-46.34 = -46.06 + -0.28)

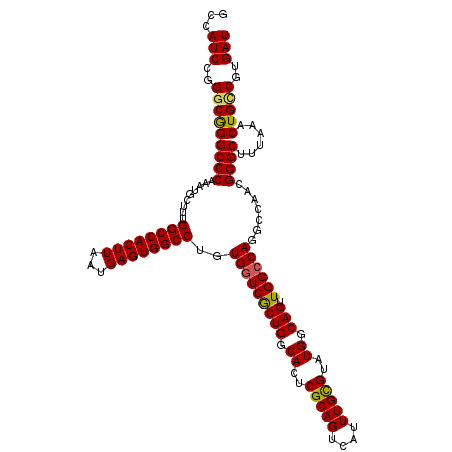
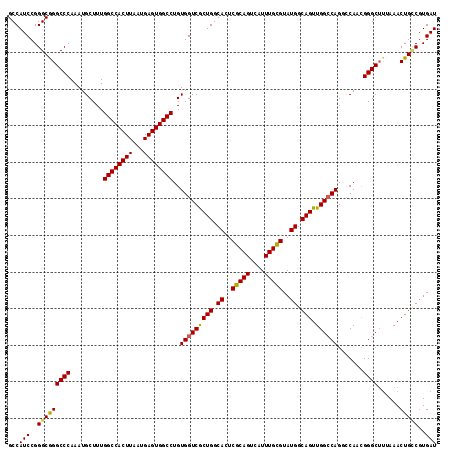
| Location | 6,264,555 – 6,264,657 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.67 |
| Mean single sequence MFE | -40.35 |
| Consensus MFE | -25.48 |
| Energy contribution | -27.28 |
| Covariance contribution | 1.81 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.56 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.77 |
| SVM RNA-class probability | 0.846385 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 6264555 102 + 20766785 UACACAAAUGACUGCGAGUGCCAGCGACCACAGGCCACUCAUUAAGUGGCCAAAGCAUUUGGGCCCGCCC---GGAUGGCUUUUAG------------CCAGAAUGCCUGGC-CG--AAC .................(.(((((........(((((((.....)))))))...(((((((((....)))---...((((.....)------------))))))))))))))-).--... ( -37.60) >DroSec_CAF1 7725 102 + 1 UACGCAAAUGACUGCGAGUGCCAGCGACCACAGGCCACUCAUUAAGUGGCCAAAGCAUUUGGGCCCGCCC---GGAUGGCUUUUAG------------CCAGAAUGGCUGGC-CG--AAC ..((((......)))).(.((((((.......(((((((.....)))))))....((((((((....)))---...((((.....)------------))))))))))))))-).--... ( -43.70) >DroSim_CAF1 7941 102 + 1 UACGCAAAUGACUGCGAGUGCCAGCGACCACAGGCCACUCAUUAAGUGGCCAAAGCAUUUGGGCCCGCCC---GGAUGGCUUUUAG------------CCAGAAUGCCUGGC-CG--AAC ..((((......)))).(.(((((........(((((((.....)))))))...(((((((((....)))---...((((.....)------------))))))))))))))-).--... ( -42.20) >DroEre_CAF1 7447 103 + 1 UACGCAAAUGACUGCGAGUGCCAGCGACCACAGGCCACUCAUUAAGUGGCCAAAGCAUUUGGGCCUGCCC---GGAUGGCCUUUAG------------CCAGAAUGCCCGGCACG--ACC ..((((......)))).(((((..........(((((((.....)))))))...(((((((((....)))---...((((.....)------------)))))))))..))))).--... ( -43.40) >DroYak_CAF1 8506 102 + 1 UACGCAAAUGGCUGCGAGUGCCAGUGACCACAGGCCACUCAUUAAGUGGCCAAAGCAUUUGGGCCUGCCC---GGAUGGCUUUUAC------------CCAGAAUGCCUGGC-CA--ACC ..((((......)))).(.(((((........(((((((.....)))))))...(((((((((...(((.---....))).....)------------)))).)))))))))-).--... ( -39.80) >DroAna_CAF1 6596 110 + 1 GA--------CCUGCUUGGGCCAGCGACCACAUGCCACUCGUUUAGUGGCCAAAGCAGUUGU-CCUGCCUCUGGCCAGGCUGUUAUCCUGUUCCUGUACCAGUCUGCCUGUC-UGCCAGU ((--------(..((...((((((.(.......((((((.....))))))....((((....-.))))).))))))((((((.(((.........))).))))))))..)))-....... ( -35.40) >consensus UACGCAAAUGACUGCGAGUGCCAGCGACCACAGGCCACUCAUUAAGUGGCCAAAGCAUUUGGGCCCGCCC___GGAUGGCUUUUAG____________CCAGAAUGCCUGGC_CG__AAC ..((((......)))).(.(((((........(((((((.....)))))))...(((((((((((..((....))..)))))...................))))))))))).)...... (-25.48 = -27.28 + 1.81)

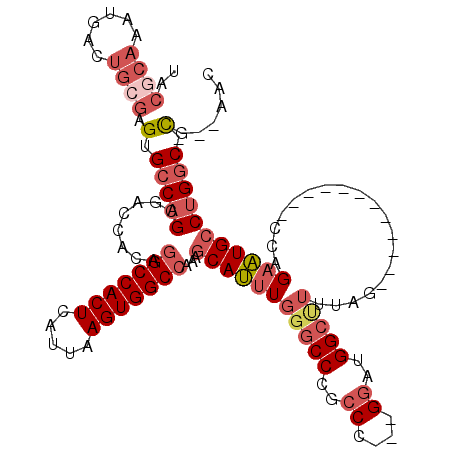
| Location | 6,264,555 – 6,264,657 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.67 |
| Mean single sequence MFE | -43.42 |
| Consensus MFE | -28.98 |
| Energy contribution | -30.23 |
| Covariance contribution | 1.25 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.47 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.82 |
| SVM RNA-class probability | 0.858614 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 6264555 102 - 20766785 GUU--CG-GCCAGGCAUUCUGG------------CUAAAAGCCAUCC---GGGCGGGCCCAAAUGCUUUGGCCACUUAAUGAGUGGCCUGUGGUCGCUGGCACUCGCAGUCAUUUGUGUA (((--((-((((((((((..((------------((....(((....---.))).))))..))))))..((((((((...))))))))..))))))..)))...(((((....))))).. ( -42.00) >DroSec_CAF1 7725 102 - 1 GUU--CG-GCCAGCCAUUCUGG------------CUAAAAGCCAUCC---GGGCGGGCCCAAAUGCUUUGGCCACUUAAUGAGUGGCCUGUGGUCGCUGGCACUCGCAGUCAUUUGCGUA ...--..-((((((((((..((------------((....(((....---.))).))))..))))((..((((((((...))))))))...))..))))))...(((((....))))).. ( -42.70) >DroSim_CAF1 7941 102 - 1 GUU--CG-GCCAGGCAUUCUGG------------CUAAAAGCCAUCC---GGGCGGGCCCAAAUGCUUUGGCCACUUAAUGAGUGGCCUGUGGUCGCUGGCACUCGCAGUCAUUUGCGUA (((--((-((((((((((..((------------((....(((....---.))).))))..))))))..((((((((...))))))))..))))))..)))...(((((....))))).. ( -43.90) >DroEre_CAF1 7447 103 - 1 GGU--CGUGCCGGGCAUUCUGG------------CUAAAGGCCAUCC---GGGCAGGCCCAAAUGCUUUGGCCACUUAAUGAGUGGCCUGUGGUCGCUGGCACUCGCAGUCAUUUGCGUA ...--.((((((((((((..((------------((....(((....---.))).))))..)))))...((((((((...))))))))........))))))).(((((....))))).. ( -45.80) >DroYak_CAF1 8506 102 - 1 GGU--UG-GCCAGGCAUUCUGG------------GUAAAAGCCAUCC---GGGCAGGCCCAAAUGCUUUGGCCACUUAAUGAGUGGCCUGUGGUCACUGGCACUCGCAGCCAUUUGCGUA (((--((-((((((((((.(((------------((....(((....---.)))..))))))))))...((((((((...))))))))........))))).....)))))......... ( -43.90) >DroAna_CAF1 6596 110 - 1 ACUGGCA-GACAGGCAGACUGGUACAGGAACAGGAUAACAGCCUGGCCAGAGGCAGG-ACAACUGCUUUGGCCACUAAACGAGUGGCAUGUGGUCGCUGGCCCAAGCAGG--------UC (((.((.-..(((.....))).....((..((((((.((((((.((((((((.(((.-....)))))))))))(((.....)))))).))).))).)))..))..)).))--------). ( -42.20) >consensus GUU__CG_GCCAGGCAUUCUGG____________CUAAAAGCCAUCC___GGGCAGGCCCAAAUGCUUUGGCCACUUAAUGAGUGGCCUGUGGUCGCUGGCACUCGCAGUCAUUUGCGUA ........(((((...........................(((........))).((((.....((...(((((((.....))))))).)))))).)))))...(((((....))))).. (-28.98 = -30.23 + 1.25)

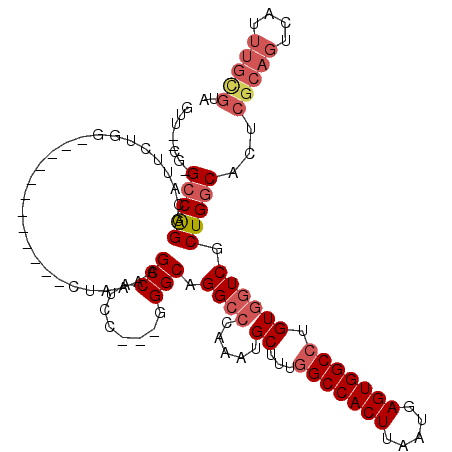

| Location | 6,264,595 – 6,264,697 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.24 |
| Mean single sequence MFE | -41.80 |
| Consensus MFE | -21.46 |
| Energy contribution | -21.10 |
| Covariance contribution | -0.36 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.85 |
| Structure conservation index | 0.51 |
| SVM decision value | 0.69 |
| SVM RNA-class probability | 0.822793 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 6264595 102 - 20766785 AUCGCCGGUCAGCUUAGAUUGCUCAAUUGCGAAUGGCCCGGUU--CG-GCCAGGCAUUCUGG------------CUAAAAGCCAUCC---GGGCGGGCCCAAAUGCUUUGGCCACUUAAU ...(((((...(((....((((......))))..)))))))).--.(-((((((((((..((------------((....(((....---.))).))))..))))).))))))....... ( -41.10) >DroSec_CAF1 7765 101 - 1 AUCGCCGGUCAGCUUAGAUUGCUCAAUUGCGAAUGGC-CGGUU--CG-GCCAGCCAUUCUGG------------CUAAAAGCCAUCC---GGGCGGGCCCAAAUGCUUUGGCCACUUAAU ...((((((((.....((((....)))).....))))-)))).--.(-(((((.((((..((------------((....(((....---.))).))))..))))..))))))....... ( -38.40) >DroSim_CAF1 7981 101 - 1 AUCGCCGGUCAGCUUAGAUUGCUCAAUUGCGAAUGGC-CGGUU--CG-GCCAGGCAUUCUGG------------CUAAAAGCCAUCC---GGGCGGGCCCAAAUGCUUUGGCCACUUAAU ...((((((((.....((((....)))).....))))-)))).--.(-((((((((((..((------------((....(((....---.))).))))..))))).))))))....... ( -43.10) >DroEre_CAF1 7487 102 - 1 AUCGCCGGUCAGCUUAGAUUGCUCAAUUGCAAAUGGC-CGGGU--CGUGCCGGGCAUUCUGG------------CUAAAGGCCAUCC---GGGCAGGCCCAAAUGCUUUGGCCACUUAAU ...((((((((.......((((......)))).))))-)((((--(.((((.((.....(((------------(.....)))).))---.))))))))).........)))........ ( -38.40) >DroYak_CAF1 8546 101 - 1 AUCGCCGGUCAGCUUAGAUUGCUCAAUUGCGAAUGGC-CGGGU--UG-GCCAGGCAUUCUGG------------GUAAAAGCCAUCC---GGGCAGGCCCAAAUGCUUUGGCCACUUAAU ....(((((((.....((((....)))).....))))-)))..--((-((((((((((.(((------------((....(((....---.)))..)))))))))).)))))))...... ( -42.80) >DroAna_CAF1 6628 115 - 1 CU--CCGGCUGUUUACGAUUGCCGGAUUGCAAAUGCC-AGACUGGCA-GACAGGCAGACUGGUACAGGAACAGGAUAACAGCCUGGCCAGAGGCAGG-ACAACUGCUUUGGCCACUAAAC ..--..(((((((..(...((((((.((((...((((-.....))))-.....)))).))))))........)...)))))))(((((((((.(((.-....))))))))))))...... ( -47.00) >consensus AUCGCCGGUCAGCUUAGAUUGCUCAAUUGCGAAUGGC_CGGUU__CG_GCCAGGCAUUCUGG____________CUAAAAGCCAUCC___GGGCAGGCCCAAAUGCUUUGGCCACUUAAU ......(((((.......((((......)))).((((.((.....)).))))((((((.(((..................(((........)))....))))))))).)))))....... (-21.46 = -21.10 + -0.36)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:09:58 2006