
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 6,124,706 – 6,124,854 |
| Length | 148 |
| Max. P | 0.999234 |

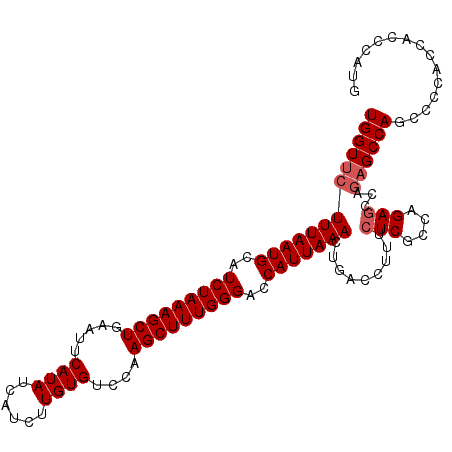
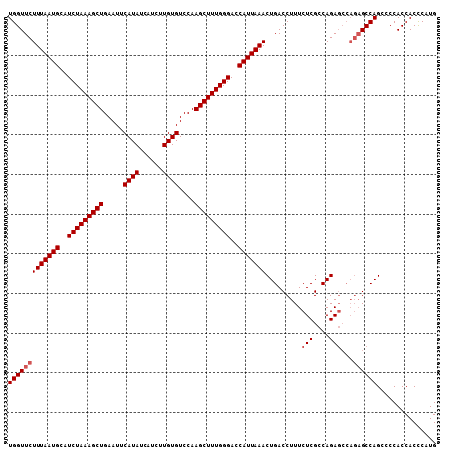
| Location | 6,124,706 – 6,124,814 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 97.28 |
| Mean single sequence MFE | -26.45 |
| Consensus MFE | -24.97 |
| Energy contribution | -25.47 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.17 |
| Structure conservation index | 0.94 |
| SVM decision value | 3.41 |
| SVM RNA-class probability | 0.999174 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 6124706 108 - 20766785 UGGUUCUUUAAUGCAUCUAAAGCUGAAUUCAUAUCAUCUUGUGUCCAAGCUUUGGGACCAUUAAACUGACCUUUCUCGCCAGAGCCAGAGCCAGCCCCACCACUCAUG (((((((((((((..(((((((((.....((((......))))....)))))))))..))))))..........(((....)))..)))))))............... ( -26.90) >DroPse_CAF1 127909 108 - 1 UGGUUCUUUAAUGCAUCUAAAGCUGAAUUCAUAUCAUCUUGUGUCCAAGCUUUGGGACCAUUAAACUGACCUUUCUCGCCAGAGCCAGAGCCAGCCCCACCACCCAUG (((((((((((((..(((((((((.....((((......))))....)))))))))..))))))..........(((....)))..)))))))............... ( -26.90) >DroEre_CAF1 137862 108 - 1 UGGUUCUUUAAUGCAUCUAAAGCUGAAUUCAUAUCAUCUUGUGUCCAAGCUUUGGGACCAUUAAACUGACCUUUCUCGCCAGAGCCAGAGCCAGCCCCACCACUCAUG (((((((((((((..(((((((((.....((((......))))....)))))))))..))))))..........(((....)))..)))))))............... ( -26.90) >DroYak_CAF1 137033 102 - 1 UGGUUCUUUAAUGCAUCUAAAGCUGAAUUCAUAUCAUCUUGUGUCCAAGCUUUGGGACCAUUAAACUGACCUUUCUCGCCAGA------GCCAGCCCCACCACUCAUG (((((((((((((..(((((((((.....((((......))))....)))))))))..))))))...((......))...)))------))))............... ( -24.20) >DroAna_CAF1 165225 108 - 1 UGGUUCUUUAAUGCAUCUAAAGCUGAAUUCAUAUCAUCUUGUGUCCAAGCUUUGGGACCAUUAAACUGACCUUUCUCGCCAGAGCCAGAGCCAGCACCACCACCCAUG (((((((((((((..(((((((((.....((((......))))....)))))))))..))))))..........(((....)))..)))))))............... ( -26.90) >DroPer_CAF1 127745 108 - 1 UGGUUCUUUAAUGCAUCUAAAGCUGAAUUCAUAUCAUCUUGUGUCCAAGCUUUGGGACCAUUAAACUGACCUUUCUCGCCAGAGCCAGAGCCAGCCCCACCACCCAUG (((((((((((((..(((((((((.....((((......))))....)))))))))..))))))..........(((....)))..)))))))............... ( -26.90) >consensus UGGUUCUUUAAUGCAUCUAAAGCUGAAUUCAUAUCAUCUUGUGUCCAAGCUUUGGGACCAUUAAACUGACCUUUCUCGCCAGAGCCAGAGCCAGCCCCACCACCCAUG (((((((((((((..(((((((((.....((((......))))....)))))))))..))))))).........(((....)))...))))))............... (-24.97 = -25.47 + 0.50)

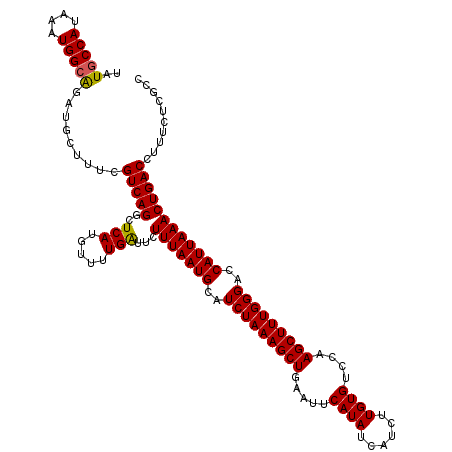
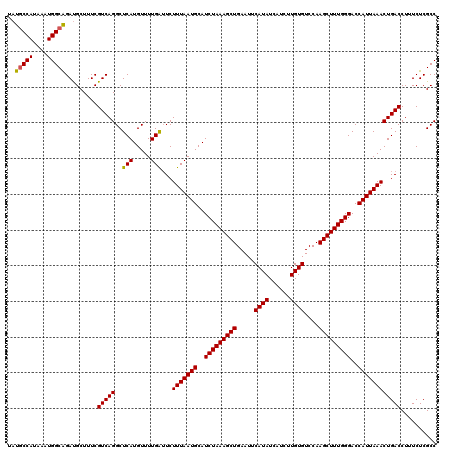
| Location | 6,124,734 – 6,124,854 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.44 |
| Mean single sequence MFE | -31.85 |
| Consensus MFE | -29.47 |
| Energy contribution | -29.13 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.44 |
| Structure conservation index | 0.93 |
| SVM decision value | 3.45 |
| SVM RNA-class probability | 0.999234 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 6124734 120 - 20766785 GAUGCCAUAAAUGGCGGAUGCUUUCGUCAGGCUCAUGUUUUGGUUCUUUAAUGCAUCUAAAGCUGAAUUCAUAUCAUCUUGUGUCCAAGCUUUGGGACCAUUAAACUGACCUUUCUCGCC ((.(((.....(((((((....)))))))))))).......((((.(((((((..(((((((((.....((((......))))....)))))))))..)))))))..))))......... ( -32.80) >DroVir_CAF1 159228 120 - 1 UAUGCCAUAAAUGGCAGAUGCUUUCGUCAGGCUCAUGUUUUGAUUCUUUAAUGCAUCUAAAGCUGAAUUCAUAUCAUCUUGUGUCCAAGCUUUGGGACCAUUAAACUGACCUUUCUCGCC ..(((((....))))).........(((((..(((.....)))...(((((((..(((((((((.....((((......))))....)))))))))..)))))))))))).......... ( -30.70) >DroGri_CAF1 150776 120 - 1 UAUGCCAUAAAUGGCAGAUGCUUUCGUCAGGCUCAUGUUUUGAUUCUUUAAUGCAUCUAAAGCUGAAUUCAUAUCAUCUUGUGUCCAAGCUUUGGGACCAUUAAACUGACCUUUCUCGCC ..(((((....))))).........(((((..(((.....)))...(((((((..(((((((((.....((((......))))....)))))))))..)))))))))))).......... ( -30.70) >DroWil_CAF1 148258 120 - 1 UAUACCAUAUAUGGCGGAUGCUUUCGUCAGGCUCAUGUUUUGAUUCUUUAAUGCAUCUAAAGCUGAAUUCAUAUCAUCUUGUGUCCAAGCUUUGGGACCAUUAAACUGACCUUUCUCGCC ............((((((.....))(((((..(((.....)))...(((((((..(((((((((.....((((......))))....)))))))))..))))))))))))......)))) ( -30.00) >DroMoj_CAF1 158674 120 - 1 UAUGCCAUAAAUGGCAGAUGCUUUCGUCAGGCUCAUGUUUUGGGUCUUUAAUGCAUCUAAAGCUGAAUUCAUAUCAUCUUGUGUCCAAGCUUUGGGACCAUUAAACUGACCUUUCUCGCC ..(((((....)))))((((....)))).(((....(....((((((((((((..(((((((((.....((((......))))....)))))))))..)))))))..)))))..)..))) ( -34.30) >DroAna_CAF1 165253 120 - 1 GAUGCCAUAAAUGGCGGACGCUUUCGUCAGGCUCAUGUUUUGGUUCUUUAAUGCAUCUAAAGCUGAAUUCAUAUCAUCUUGUGUCCAAGCUUUGGGACCAUUAAACUGACCUUUCUCGCC ..(((((....)))))((((....)))).(((....(....((((.(((((((..(((((((((.....((((......))))....)))))))))..)))))))..))))...)..))) ( -32.60) >consensus UAUGCCAUAAAUGGCAGAUGCUUUCGUCAGGCUCAUGUUUUGAUUCUUUAAUGCAUCUAAAGCUGAAUUCAUAUCAUCUUGUGUCCAAGCUUUGGGACCAUUAAACUGACCUUUCUCGCC ..(((((....))))).........(((((..(((.....)))...(((((((..(((((((((.....((((......))))....)))))))))..)))))))))))).......... (-29.47 = -29.13 + -0.33)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:09:17 2006