
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 5,944,321 – 5,944,507 |
| Length | 186 |
| Max. P | 0.999993 |

| Location | 5,944,321 – 5,944,439 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.44 |
| Mean single sequence MFE | -32.39 |
| Consensus MFE | -25.78 |
| Energy contribution | -25.98 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.37 |
| Structure conservation index | 0.80 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.510262 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 5944321 118 + 20766785 GCUCUGGCACACACACGCAC--AGGGCACAGACAGGUGCUCUCAGCUGUGCACUGGUGGUUUUUGUUUUUCAUAACCCACUUCUCCCACGCUCAUUCGCGUGGCUGUUAAACCGCUAACA ((((((((........)).)--)))))...(((((((((.(......).)))).(((((.((.((.....)).)).)))))....((((((......)))))))))))............ ( -38.10) >DroSec_CAF1 86111 120 + 1 GCUCUGACACACACACACACAUAGAGCAAUGACAGGUGCUCUCAGCUGUGCACUGGUGGUUUUUGUUUUUCAUUACCCACUUCUCCCACGCUCAUUCGCGUGGCUGUUAAACGGCUAACA ((((((...............))))))..((((((((((.(......).)))).(((((.(..((.....))..).)))))....((((((......))))))))))))........... ( -32.86) >DroSim_CAF1 84021 120 + 1 GCUCUGACACACACACACACAUAGGGCACAGACAGGUGCUCUCAGCUGUGCACUGGUGGUUUUUGUUUUUCAUUACCCACUUCUCCCACGCUCAUUCGCGUGGCUGUUAAACCGCUAACA ((..(((((......(((((..(((((((......)))))))..).))))....(((((.(..((.....))..).)))))....((((((......)))))).)))))....))..... ( -31.10) >DroEre_CAF1 71538 115 + 1 GCUCUGGAACACACACACAU-----GCACAGACAGGUGCUCUCAGCUGUGCGCUGGUGGUUUUUGUUUUUCAUUACUCACUUCUCCCACGCUCAUUCGCGUGGCUGUUAAGCCGCUAACA ((..(((((.((((((.(((-----((((((...((....))...))))))).))))).....))).)))))...............((((......))))((((....))))))..... ( -29.20) >DroYak_CAF1 89093 115 + 1 ACUCUGGCACACUCACACAU-----GCACAGACAGGUUCUCUCAGCUGUGCGCUGGUGGUUUUUGUUUUUCGUUACUCACUUCUCCCACGCUCAUUCGCGUGGCUGUUAAGCCGCUAACA .....(((....((((.(((-----((((((...((....))...))))))).))))))..........................((((((......)))))).......)))....... ( -30.70) >consensus GCUCUGGCACACACACACAC__AG_GCACAGACAGGUGCUCUCAGCUGUGCACUGGUGGUUUUUGUUUUUCAUUACCCACUUCUCCCACGCUCAUUCGCGUGGCUGUUAAACCGCUAACA .........................((((((...((....))...))))))..(((((((((.((.....)).............((((((......)))))).....)))))))))... (-25.78 = -25.98 + 0.20)

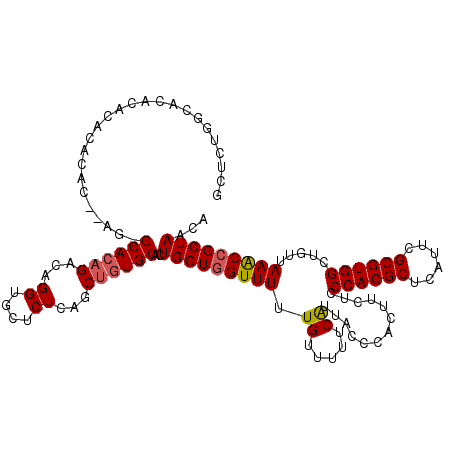
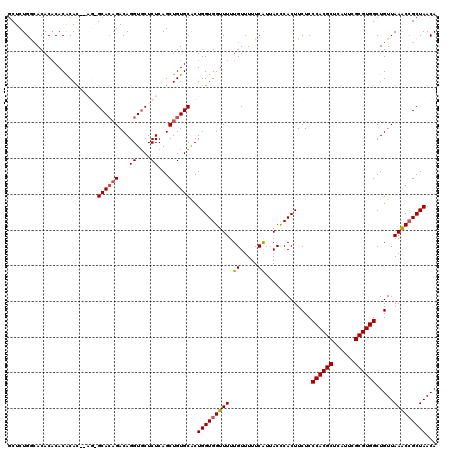
| Location | 5,944,359 – 5,944,477 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.95 |
| Mean single sequence MFE | -37.50 |
| Consensus MFE | -36.64 |
| Energy contribution | -36.24 |
| Covariance contribution | -0.40 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.33 |
| Structure conservation index | 0.98 |
| SVM decision value | 5.74 |
| SVM RNA-class probability | 0.999993 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 5944359 118 + 20766785 CUCAGCUGUGCACUGGUGGUUUUUGUUUUUCAUAACCCACUUCUCCCACGCUCAUUCGCGUGGCUGUUAAACCGCUAACAGCAACAGCUGGCUUACCAGUGCGACAUA--ACAUGUAAAA ......(((((((((((((((.(((((...................(((((......)))))(((((((......))))))))))))..))).))))))))).)))..--.......... ( -37.30) >DroSec_CAF1 86151 118 + 1 CUCAGCUGUGCACUGGUGGUUUUUGUUUUUCAUUACCCACUUCUCCCACGCUCAUUCGCGUGGCUGUUAAACGGCUAACAGCAACAGCUGGCUUACCAGUGCGACAUA--ACAUGUAAAA ......(((((((((((((((.(((((...................(((((......)))))(((((((......))))))))))))..))).))))))))).)))..--.......... ( -37.30) >DroSim_CAF1 84061 118 + 1 CUCAGCUGUGCACUGGUGGUUUUUGUUUUUCAUUACCCACUUCUCCCACGCUCAUUCGCGUGGCUGUUAAACCGCUAACAGCAACAGCUGGCCUACCAGUGCGACAUA--ACAUGUAAAA ......(((((((((((((((.(((((...................(((((......)))))(((((((......))))))))))))..)))).)))))))).)))..--.......... ( -39.60) >DroEre_CAF1 71573 118 + 1 CUCAGCUGUGCGCUGGUGGUUUUUGUUUUUCAUUACUCACUUCUCCCACGCUCAUUCGCGUGGCUGUUAAGCCGCUAACAGCAACAGCUGUAUUACCAGUGCAACAUA--ACAUGUAAAA ......((((((((((((((..(((((...................(((((......)))))(((((((......))))))))))))....))))))))))).)))..--.......... ( -36.60) >DroYak_CAF1 89128 120 + 1 CUCAGCUGUGCGCUGGUGGUUUUUGUUUUUCGUUACUCACUUCUCCCACGCUCAUUCGCGUGGCUGUUAAGCCGCUAACAGCAACAGCUGGCUUACCAGUGCAAUAUAAAACAUGUAAAA ........(((((((((((((.(((((...................(((((......)))))(((((((......))))))))))))..))).))))))))))................. ( -36.70) >consensus CUCAGCUGUGCACUGGUGGUUUUUGUUUUUCAUUACCCACUUCUCCCACGCUCAUUCGCGUGGCUGUUAAACCGCUAACAGCAACAGCUGGCUUACCAGUGCGACAUA__ACAUGUAAAA ........(((((((((((((.(((((...................(((((......)))))(((((((......))))))))))))..)))).)))))))))((((.....)))).... (-36.64 = -36.24 + -0.40)

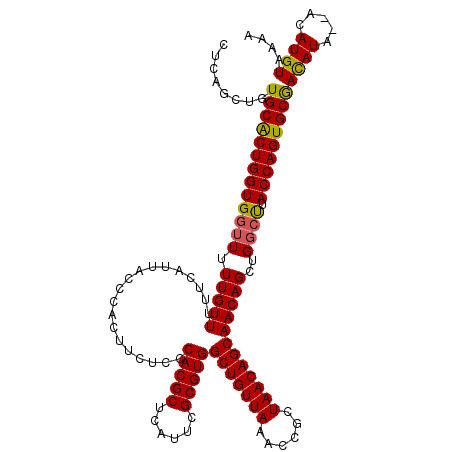
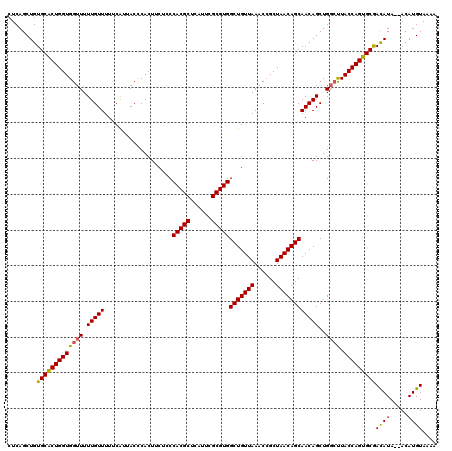
| Location | 5,944,359 – 5,944,477 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.95 |
| Mean single sequence MFE | -37.94 |
| Consensus MFE | -32.22 |
| Energy contribution | -32.78 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.19 |
| Structure conservation index | 0.85 |
| SVM decision value | 2.22 |
| SVM RNA-class probability | 0.990527 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 5944359 118 - 20766785 UUUUACAUGU--UAUGUCGCACUGGUAAGCCAGCUGUUGCUGUUAGCGGUUUAACAGCCACGCGAAUGAGCGUGGGAGAAGUGGGUUAUGAAAAACAAAAACCACCAGUGCACAGCUGAG .....((.((--..(((.((((((((..((((((....))))...))(((((.....((((((......)))))).........(((......)))..)))))))))))))))))))).. ( -39.50) >DroSec_CAF1 86151 118 - 1 UUUUACAUGU--UAUGUCGCACUGGUAAGCCAGCUGUUGCUGUUAGCCGUUUAACAGCCACGCGAAUGAGCGUGGGAGAAGUGGGUAAUGAAAAACAAAAACCACCAGUGCACAGCUGAG (((..(((((--(...((((.((((....))))..((.((((((((....)))))))).))))))...))))))..))).((((....((.....))....))))((((.....)))).. ( -38.10) >DroSim_CAF1 84061 118 - 1 UUUUACAUGU--UAUGUCGCACUGGUAGGCCAGCUGUUGCUGUUAGCGGUUUAACAGCCACGCGAAUGAGCGUGGGAGAAGUGGGUAAUGAAAAACAAAAACCACCAGUGCACAGCUGAG .....((.((--..(((.((((((((.(((((((((((((((....))))..))))))(((((......))))).......)))((........)).....))))))))))))))))).. ( -39.20) >DroEre_CAF1 71573 118 - 1 UUUUACAUGU--UAUGUUGCACUGGUAAUACAGCUGUUGCUGUUAGCGGCUUAACAGCCACGCGAAUGAGCGUGGGAGAAGUGAGUAAUGAAAAACAAAAACCACCAGCGCACAGCUGAG .(((((.(((--((.(((((.........(((((....)))))..))))).))))).((((((......)))))).....)))))....................((((.....)))).. ( -34.40) >DroYak_CAF1 89128 120 - 1 UUUUACAUGUUUUAUAUUGCACUGGUAAGCCAGCUGUUGCUGUUAGCGGCUUAACAGCCACGCGAAUGAGCGUGGGAGAAGUGAGUAACGAAAAACAAAAACCACCAGCGCACAGCUGAG .......((((((.(.((((.(((.((((((.((((.......)))))))))).)))((((((......)))))).........)))).).))))))........((((.....)))).. ( -38.50) >consensus UUUUACAUGU__UAUGUCGCACUGGUAAGCCAGCUGUUGCUGUUAGCGGUUUAACAGCCACGCGAAUGAGCGUGGGAGAAGUGGGUAAUGAAAAACAAAAACCACCAGUGCACAGCUGAG ..............(((.((((((((......((((((((((....))))..))))))(((((......))))).............................)))))))))))...... (-32.22 = -32.78 + 0.56)

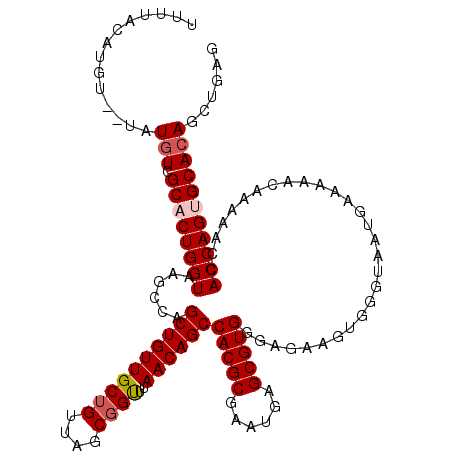
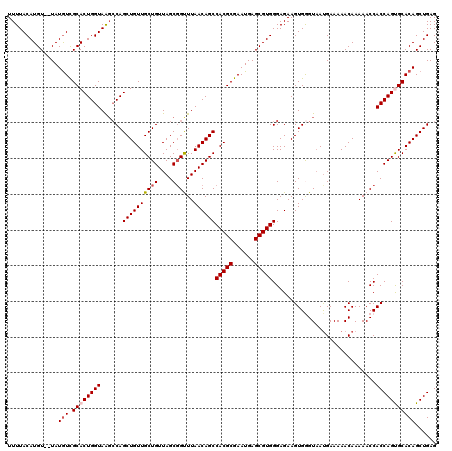
| Location | 5,944,399 – 5,944,507 |
|---|---|
| Length | 108 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 85.54 |
| Mean single sequence MFE | -29.87 |
| Consensus MFE | -25.76 |
| Energy contribution | -25.92 |
| Covariance contribution | 0.16 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.12 |
| Structure conservation index | 0.86 |
| SVM decision value | 1.64 |
| SVM RNA-class probability | 0.969300 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 5944399 108 + 20766785 UUCUCCCACGCUCAUUCGCGUGGCUGUUAAACCGCUAACAGCAACAGCUGGCUUACCAGUGCGACAUA--ACAUGUAAAAUGCGCGUAAAAUGUAUAA----------GUUAUUACGUAG .......((((.((((.((((((((((((......)))))))....(((((....)))))........--.)))))..)))).))))...(((((...----------.....))))).. ( -30.50) >DroSec_CAF1 86191 118 + 1 UUCUCCCACGCUCAUUCGCGUGGCUGUUAAACGGCUAACAGCAACAGCUGGCUUACCAGUGCGACAUA--ACAUGUAAAAUGCGCGUAAUAUGUAAAAAUUUAUAAAGGUUAUUACGUAG .......((((.((((.((((((((((((......)))))))....(((((....)))))........--.)))))..)))).))))..(((((((.(((((....))))).))))))). ( -33.90) >DroSim_CAF1 84101 118 + 1 UUCUCCCACGCUCAUUCGCGUGGCUGUUAAACCGCUAACAGCAACAGCUGGCCUACCAGUGCGACAUA--ACAUGUAAAAUUCGCGUAAUAUGUAAAAAUUUAUAAAGGUUAUUAUGUAG ......(((((......)))))(((((((......))))))).((.(((((....)))))((((....--...........))))))..(((((((.(((((....))))).))))))). ( -28.96) >DroEre_CAF1 71613 106 + 1 UUCUCCCACGCUCAUUCGCGUGGCUGUUAAGCCGCUAACAGCAACAGCUGUAUUACCAGUGCAACAUA--ACAUGUAAAAUGCGUGUAAAAUGUAUAGAUUUA---------UU---UAU ......(((((......((((((((....))))))..(((((....))))).........)).((((.--..)))).....))))).................---------..---... ( -26.00) >DroYak_CAF1 89168 111 + 1 UUCUCCCACGCUCAUUCGCGUGGCUGUUAAGCCGCUAACAGCAACAGCUGGCUUACCAGUGCAAUAUAAAACAUGUAAAAUGCGUGUAAAAUGUAUGGAUUUA---------UUGUGUAG ...((((((((......)))))(((((((......))))))).((((((((....)))))..........((((((.....))))))....)))..)))....---------........ ( -30.00) >consensus UUCUCCCACGCUCAUUCGCGUGGCUGUUAAACCGCUAACAGCAACAGCUGGCUUACCAGUGCGACAUA__ACAUGUAAAAUGCGCGUAAAAUGUAUAAAUUUA_____GUUAUUACGUAG .......((((.((((.((((((((((((......)))))))....(((((....)))))...........)))))..)))).))))................................. (-25.76 = -25.92 + 0.16)

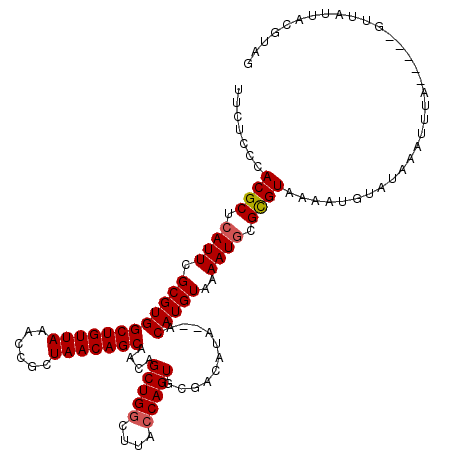
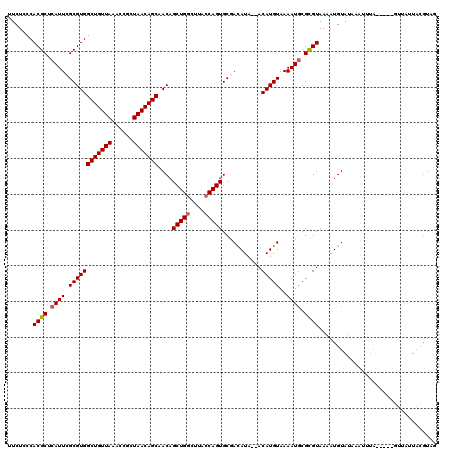
| Location | 5,944,399 – 5,944,507 |
|---|---|
| Length | 108 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 85.54 |
| Mean single sequence MFE | -30.67 |
| Consensus MFE | -26.04 |
| Energy contribution | -26.00 |
| Covariance contribution | -0.04 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.63 |
| SVM RNA-class probability | 0.804067 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 5944399 108 - 20766785 CUACGUAAUAAC----------UUAUACAUUUUACGCGCAUUUUACAUGU--UAUGUCGCACUGGUAAGCCAGCUGUUGCUGUUAGCGGUUUAACAGCCACGCGAAUGAGCGUGGGAGAA ...(((((....----------.........)))))....(((..(((((--(...((((.((((....))))..((.((((((((....)))))))).))))))...))))))..))). ( -32.82) >DroSec_CAF1 86191 118 - 1 CUACGUAAUAACCUUUAUAAAUUUUUACAUAUUACGCGCAUUUUACAUGU--UAUGUCGCACUGGUAAGCCAGCUGUUGCUGUUAGCCGUUUAACAGCCACGCGAAUGAGCGUGGGAGAA ...(((((((...................)))))))....(((..(((((--(...((((.((((....))))..((.((((((((....)))))))).))))))...))))))..))). ( -34.01) >DroSim_CAF1 84101 118 - 1 CUACAUAAUAACCUUUAUAAAUUUUUACAUAUUACGCGAAUUUUACAUGU--UAUGUCGCACUGGUAGGCCAGCUGUUGCUGUUAGCGGUUUAACAGCCACGCGAAUGAGCGUGGGAGAA ........................................(((..(((((--(...((((.((((....))))..((.((((((((....)))))))).))))))...))))))..))). ( -30.80) >DroEre_CAF1 71613 106 - 1 AUA---AA---------UAAAUCUAUACAUUUUACACGCAUUUUACAUGU--UAUGUUGCACUGGUAAUACAGCUGUUGCUGUUAGCGGCUUAACAGCCACGCGAAUGAGCGUGGGAGAA ...---..---------....(((.......................(((--((.(((((.........(((((....)))))..))))).))))).((((((......)))))).))). ( -27.20) >DroYak_CAF1 89168 111 - 1 CUACACAA---------UAAAUCCAUACAUUUUACACGCAUUUUACAUGUUUUAUAUUGCACUGGUAAGCCAGCUGUUGCUGUUAGCGGCUUAACAGCCACGCGAAUGAGCGUGGGAGAA ........---------....................(((.................))).(((.((((((.((((.......)))))))))).)))((((((......))))))..... ( -28.53) >consensus CUACAUAAUAAC_____UAAAUUUAUACAUUUUACGCGCAUUUUACAUGU__UAUGUCGCACUGGUAAGCCAGCUGUUGCUGUUAGCGGUUUAACAGCCACGCGAAUGAGCGUGGGAGAA ........................................(((..(((((......((((.((((....))))..((.((((((((....)))))))).))))))....)))))..))). (-26.04 = -26.00 + -0.04)

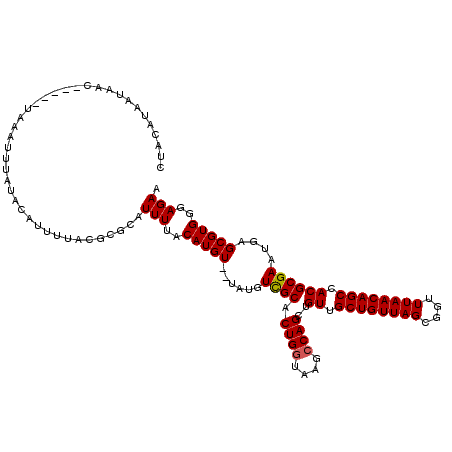
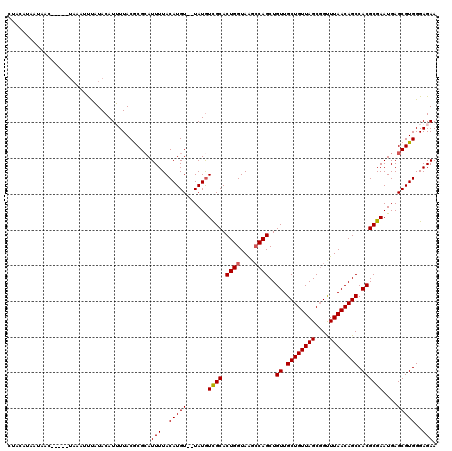
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:08:12 2006