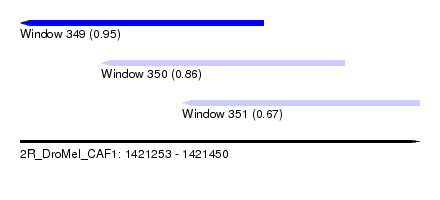
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 1,421,253 – 1,421,450 |
| Length | 197 |
| Max. P | 0.949859 |

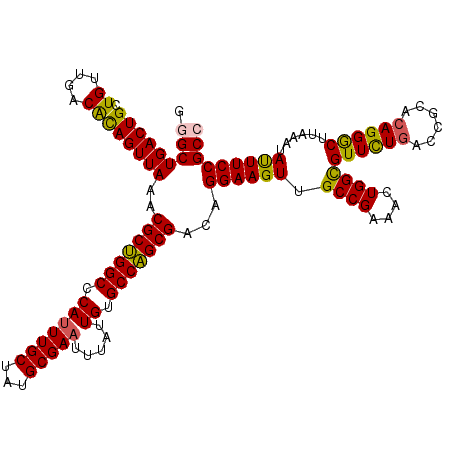
| Location | 1,421,253 – 1,421,373 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.17 |
| Mean single sequence MFE | -40.92 |
| Consensus MFE | -39.72 |
| Energy contribution | -39.16 |
| Covariance contribution | -0.56 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.36 |
| Structure conservation index | 0.97 |
| SVM decision value | 1.40 |
| SVM RNA-class probability | 0.949859 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1421253 120 - 20766785 GGGCUGACUGCUGUUGACACAGUUAAACGCUGGCCCAUUUGCUAUGCGAAUUUAUUGUGCCAGCGACAGGAAGUUGCCGAAACUGGCGUUUUGACCGCGCAGGGCUUAAAUAUUUCCGCC .(((((((((.((....))))))))..(((((((.(((((((...))))).....)).)))))))...((((((.(((....((((((.......))).))))))......))))))))) ( -39.50) >DroSec_CAF1 39698 120 - 1 GGGCUGACUGCUGUUGACACAGUUAAACGCUGGCCCAUUUGCUAUGCGAAUUUAUUGUGCCAGCGACAGGAAGUUGCCGAAACUGGCGUUCUGACCGCACAGGGCUUAAAUACUUCCGCC .(((((((((.((....))))))))..(((((((.(((((((...))))).....)).)))))))...((((((.((((....))))((((((......))))))......))))))))) ( -43.90) >DroSim_CAF1 44750 120 - 1 GGGCUGACUGCUGUUGACACAGUUAAACGCUGGCCCAUUUGCUAUGCGAAUUUAUUGUGCCAGCGACAGGAAGUUGCCGAAACUGGCGUUCUGACCGCACAGGGCUUAAAUACUUCCGCC .(((((((((.((....))))))))..(((((((.(((((((...))))).....)).)))))))...((((((.((((....))))((((((......))))))......))))))))) ( -43.90) >DroEre_CAF1 38042 119 - 1 G-GCUGACUGCUGUUGACGCAGUUAAACGCCGGCCCACUUGCUAUGCGAAUUUAUUGCGCCGGCGACAGGAAGUUGCCGAAACUGGCGUUCUGACCGCACAGGGCUUAAAUAUUUCCGCC (-(((((((((.......)))))))..(((((((.((.((((...))))......)).)))))))...((((((.((((....))))((((((......))))))......))))))))) ( -43.20) >DroYak_CAF1 39075 120 - 1 GGGCUGACUGCUGUUGACAUAGUUAAACGCUGGCCCAUUUGCUAUGCGAAUUUAUUGUGCCAGCGACAGGAAGUUGCCGAAACUGGUGUUCUGACCGCAAAGGACUUAAAUAUUUCCGCG ..((((((((.((....))))))))..(((((((.(((((((...))))).....)).)))))))...((((((.((((....))))(((((........)))))......)))))))). ( -34.10) >consensus GGGCUGACUGCUGUUGACACAGUUAAACGCUGGCCCAUUUGCUAUGCGAAUUUAUUGUGCCAGCGACAGGAAGUUGCCGAAACUGGCGUUCUGACCGCACAGGGCUUAAAUAUUUCCGCC .(((((((((.((....))))))))..(((((((.(((((((...))))).....)).)))))))...((((((.((((....))))((((((......))))))......))))))))) (-39.72 = -39.16 + -0.56)

| Location | 1,421,293 – 1,421,413 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.98 |
| Mean single sequence MFE | -42.10 |
| Consensus MFE | -36.74 |
| Energy contribution | -36.54 |
| Covariance contribution | -0.20 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.52 |
| Structure conservation index | 0.87 |
| SVM decision value | 0.83 |
| SVM RNA-class probability | 0.860291 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1421293 120 - 20766785 GUCAAGUCCGGGCUUGUUUACUCUUCUUGAGAUGCGAGUCGGGCUGACUGCUGUUGACACAGUUAAACGCUGGCCCAUUUGCUAUGCGAAUUUAUUGUGCCAGCGACAGGAAGUUGCCGA ((((.((((((.(((((...(((.....)))..))))))))))))))).(((((....)))))....(((((((.(((((((...))))).....)).)))))))...((......)).. ( -42.50) >DroSec_CAF1 39738 119 - 1 GUCAAGUC-GGGCUUGUUUACUCUCCUUGAGAUGCGAGUCGGGCUGACUGCUGUUGACACAGUUAAACGCUGGCCCAUUUGCUAUGCGAAUUUAUUGUGCCAGCGACAGGAAGUUGCCGA ((((.(((-.(((((((...(((.....)))..))))))).))))))).(((((....)))))....(((((((.(((((((...))))).....)).)))))))...((......)).. ( -42.00) >DroSim_CAF1 44790 119 - 1 GUCAAGUC-GGGCUUGUUUACUCUCCUUGAGAUGCGAGUCGGGCUGACUGCUGUUGACACAGUUAAACGCUGGCCCAUUUGCUAUGCGAAUUUAUUGUGCCAGCGACAGGAAGUUGCCGA ((((.(((-.(((((((...(((.....)))..))))))).))))))).(((((....)))))....(((((((.(((((((...))))).....)).)))))))...((......)).. ( -42.00) >DroEre_CAF1 38082 118 - 1 GUCAAGUC-GGGCGUGUUUACUCUGCUUGAGAUGCGAGUCG-GCUGACUGCUGUUGACGCAGUUAAACGCCGGCCCACUUGCUAUGCGAAUUUAUUGCGCCGGCGACAGGAAGUUGCCGA (.(((((.-((((......((((.((.......))))))((-(((((((((.......)))))))...))))))))))))))...((((.....))))..(((((((.....))))))). ( -46.30) >DroYak_CAF1 39115 119 - 1 GUCAAGUC-GGGUUUGUUUACUCUUCUUGAGAUGCGAGUCGGGCUGACUGCUGUUGACAUAGUUAAACGCUGGCCCAUUUGCUAUGCGAAUUUAUUGUGCCAGCGACAGGAAGUUGCCGA .(((((..-((((......))))..)))))........(((((((..(((.(((((.((((((....(((((((......)))).))).....)))))).))))).)))..)))..)))) ( -37.70) >consensus GUCAAGUC_GGGCUUGUUUACUCUCCUUGAGAUGCGAGUCGGGCUGACUGCUGUUGACACAGUUAAACGCUGGCCCAUUUGCUAUGCGAAUUUAUUGUGCCAGCGACAGGAAGUUGCCGA ..........(((((((...(((.....)))..)))))))(((((..(((.(((((.((((((....(((((((......)))).))).....)))))).))))).)))..)))..)).. (-36.74 = -36.54 + -0.20)

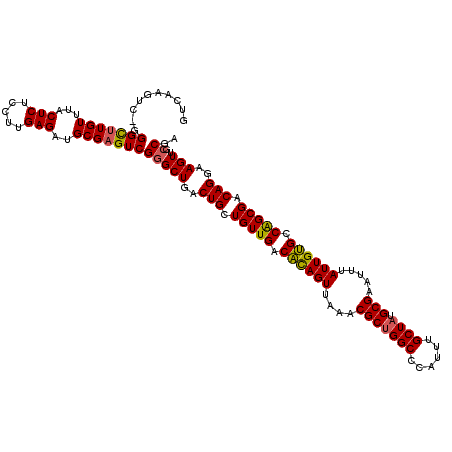
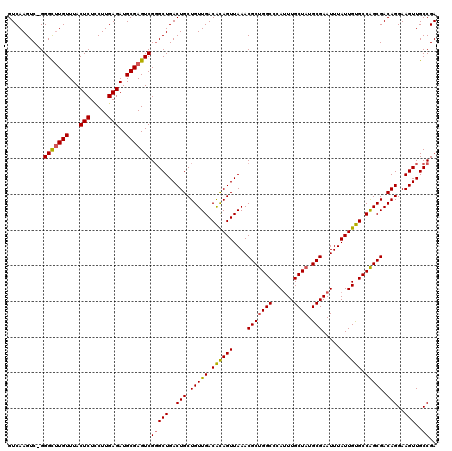
| Location | 1,421,333 – 1,421,450 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.08 |
| Mean single sequence MFE | -47.10 |
| Consensus MFE | -37.24 |
| Energy contribution | -37.44 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.60 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.29 |
| SVM RNA-class probability | 0.671874 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1421333 117 - 20766785 UGGCCGGAGUAG---CAGGUGCUCCGCUCUGACCGGGCUCGUCAAGUCCGGGCUUGUUUACUCUUCUUGAGAUGCGAGUCGGGCUGACUGCUGUUGACACAGUUAAACGCUGGCCCAUUU .((.((((((((---(....)))..)))))).))(((((.((((.((((((.(((((...(((.....)))..))))))))))))))).(((((....)))))........))))).... ( -47.80) >DroSec_CAF1 39778 119 - 1 UGGCCGGAGUAGCAGCAGGUGCUCCGCUCUGACCGGGCUCGUCAAGUC-GGGCUUGUUUACUCUCCUUGAGAUGCGAGUCGGGCUGACUGCUGUUGACACAGUUAAACGCUGGCCCAUUU .((.(((((..(.(((....))))..))))).))(((((.((((.(((-.(((((((...(((.....)))..))))))).))))))).(((((....)))))........))))).... ( -49.10) >DroSim_CAF1 44830 119 - 1 UGGCCGGAGUAGCAGCAGGUGCUCCGCUCUGACCGGGCUCGUCAAGUC-GGGCUUGUUUACUCUCCUUGAGAUGCGAGUCGGGCUGACUGCUGUUGACACAGUUAAACGCUGGCCCAUUU .((.(((((..(.(((....))))..))))).))(((((.((((.(((-.(((((((...(((.....)))..))))))).))))))).(((((....)))))........))))).... ( -49.10) >DroEre_CAF1 38122 115 - 1 UGCCCGGAGUAG---CAGGUGCUCCGCCCUGACCGGGCUCGUCAAGUC-GGGCGUGUUUACUCUGCUUGAGAUGCGAGUCG-GCUGACUGCUGUUGACGCAGUUAAACGCCGGCCCACUU .((((((.....---((((.((...)))))).)))))).....((((.-((((......((((.((.......))))))((-(((((((((.......)))))))...)))))))))))) ( -48.10) >DroYak_CAF1 39155 116 - 1 UAGUCGGGGUAG---CAGGUGCUCCGCUCUGACCGUGCUCGUCAAGUC-GGGUUUGUUUACUCUUCUUGAGAUGCGAGUCGGGCUGACUGCUGUUGACAUAGUUAAACGCUGGCCCAUUU .....(((.(((---(.((....))((.(((((((((.((.(((((..-((((......))))..))))))))))).)))))))((((((.((....))))))))...)))).))).... ( -41.40) >consensus UGGCCGGAGUAG___CAGGUGCUCCGCUCUGACCGGGCUCGUCAAGUC_GGGCUUGUUUACUCUCCUUGAGAUGCGAGUCGGGCUGACUGCUGUUGACACAGUUAAACGCUGGCCCAUUU .((((((((((........))))))((.....((.((((((((((((....)))))....(((.....)))..))))))).)).((((((.((....))))))))...)).))))..... (-37.24 = -37.44 + 0.20)

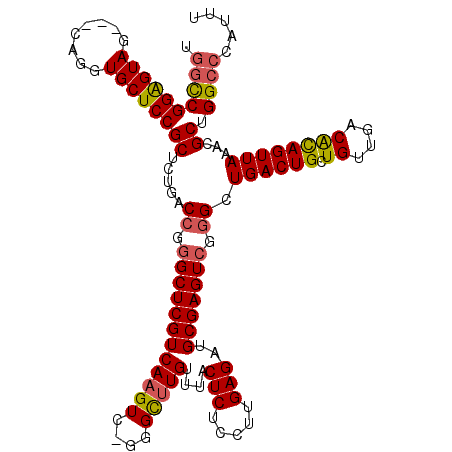
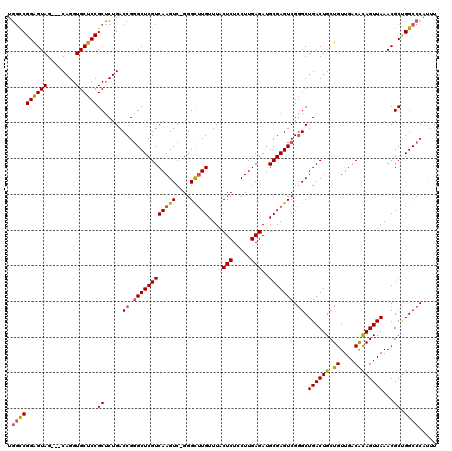
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:30:26 2006