
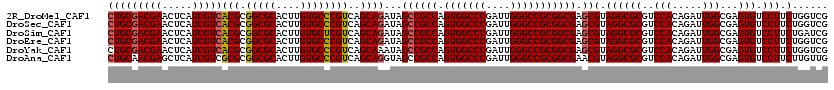
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 5,924,378 – 5,924,532 |
| Length | 154 |
| Max. P | 0.992758 |

| Location | 5,924,378 – 5,924,492 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 98.60 |
| Mean single sequence MFE | -49.42 |
| Consensus MFE | -48.72 |
| Energy contribution | -48.96 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.84 |
| Structure conservation index | 0.99 |
| SVM decision value | 2.35 |
| SVM RNA-class probability | 0.992758 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 5924378 114 + 20766785 UUAUCCUUGCCCUGAUCCAGAAGAUGCCUCCGUCCUGCGACGAACUCAUCGUCACGCGGCGCACUUGUGCCCGUCAGCAGAUAGCCGCCAGUGGCCCGAUUGGGCCGCGGCGAG .....(((((((((........((((....))))(((((((((.....)))))(((.(((((....))))))))..)))).)))......(((((((....))))))))))))) ( -47.20) >DroSec_CAF1 66314 114 + 1 UUAUCCUUGCCCUGAUCCAGGAGAUGCCUCCGUCCUGCGACGAACUCAUCGUCACGCGGCGCACUUGUGCCCGUCAGCAGAUAGCCGCCAGUGGCCCGAUUGGGCCGCGGCGAG .....(((((((((.....((((....))))...(((((((((.....)))))(((.(((((....))))))))..)))).)))......(((((((....))))))))))))) ( -51.50) >DroSim_CAF1 60113 114 + 1 UUAUCCUUGCCCUGAUCCAGGAGAUGCCUCCGUCCUGCGACGAACUCAUCGUCACGCGGCGCACUUGUGCUCGUCAGCAGAUAGCCGCCAGUGGCCCGAUUGGGCCGCGGCGAG .....(((((((((.....((((....))))...(((((((((.....)))))..(((((((....)))).)))..)))).)))......(((((((....))))))))))))) ( -48.50) >DroEre_CAF1 57238 114 + 1 UUAUCCUUGCCCUGAUCCAGGAGAUGCCUCCGUCCUGCGACGAACUCAUCGUCACGCGGCGCACUUGUGCCCGUCAGCAGAUAGCCGCCAGUGGCCCGAUUGGGCCGCGGCGAG .....(((((((((.....((((....))))...(((((((((.....)))))(((.(((((....))))))))..)))).)))......(((((((....))))))))))))) ( -51.50) >DroYak_CAF1 60426 114 + 1 UUAUCCUUGCCCUGAUCCAGGAGACGCCUCCGUCCUGCGACGAACUCAUCGUCACGCGGCGCACUUGUGCCCGUCAGCAAAUAGCCGCCAGUGGCCCGAUUGGGCCGCGGCGAG ........((((((...)))).((((....)))).((((((((.....)))))(((.(((((....))))))))..)))....))((((.(((((((....))))))))))).. ( -48.40) >consensus UUAUCCUUGCCCUGAUCCAGGAGAUGCCUCCGUCCUGCGACGAACUCAUCGUCACGCGGCGCACUUGUGCCCGUCAGCAGAUAGCCGCCAGUGGCCCGAUUGGGCCGCGGCGAG .....(((((((((.....((((....))))...(((((((((.....)))))(((.(((((....))))))))..)))).)))......(((((((....))))))))))))) (-48.72 = -48.96 + 0.24)

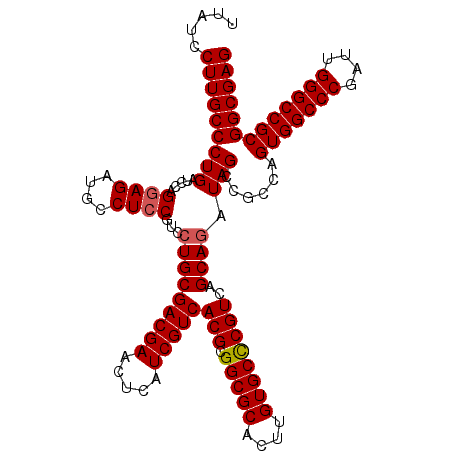
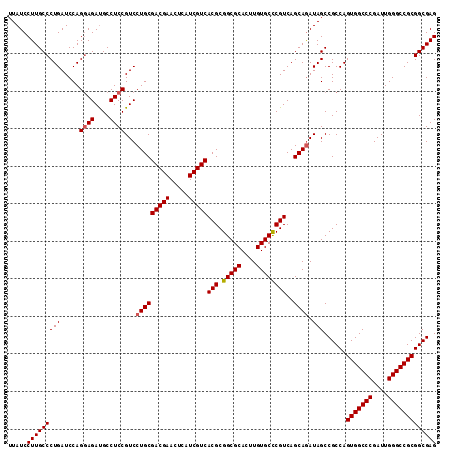
| Location | 5,924,378 – 5,924,492 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 98.60 |
| Mean single sequence MFE | -49.94 |
| Consensus MFE | -49.02 |
| Energy contribution | -48.70 |
| Covariance contribution | -0.32 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.16 |
| Structure conservation index | 0.98 |
| SVM decision value | 0.04 |
| SVM RNA-class probability | 0.551267 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 5924378 114 - 20766785 CUCGCCGCGGCCCAAUCGGGCCACUGGCGGCUAUCUGCUGACGGGCACAAGUGCGCCGCGUGACGAUGAGUUCGUCGCAGGACGGAGGCAUCUUCUGGAUCAGGGCAAGGAUAA ((.(((..(((((....))))).((((((((...(((....)))(((....))))))))(((((((.....)))))))....((((((...))))))...)))))).))..... ( -49.80) >DroSec_CAF1 66314 114 - 1 CUCGCCGCGGCCCAAUCGGGCCACUGGCGGCUAUCUGCUGACGGGCACAAGUGCGCCGCGUGACGAUGAGUUCGUCGCAGGACGGAGGCAUCUCCUGGAUCAGGGCAAGGAUAA ((.(((..(((((....))))).((((((((...(((....)))(((....))))))))(((((((.....))))))).....((((....)))).....)))))).))..... ( -50.00) >DroSim_CAF1 60113 114 - 1 CUCGCCGCGGCCCAAUCGGGCCACUGGCGGCUAUCUGCUGACGAGCACAAGUGCGCCGCGUGACGAUGAGUUCGUCGCAGGACGGAGGCAUCUCCUGGAUCAGGGCAAGGAUAA ...((((((((((....)))))....)))))((((((((..((.(((....))).((..(((((((.....))))))).)).))..)))...(((((...)))))...))))). ( -49.80) >DroEre_CAF1 57238 114 - 1 CUCGCCGCGGCCCAAUCGGGCCACUGGCGGCUAUCUGCUGACGGGCACAAGUGCGCCGCGUGACGAUGAGUUCGUCGCAGGACGGAGGCAUCUCCUGGAUCAGGGCAAGGAUAA ((.(((..(((((....))))).((((((((...(((....)))(((....))))))))(((((((.....))))))).....((((....)))).....)))))).))..... ( -50.00) >DroYak_CAF1 60426 114 - 1 CUCGCCGCGGCCCAAUCGGGCCACUGGCGGCUAUUUGCUGACGGGCACAAGUGCGCCGCGUGACGAUGAGUUCGUCGCAGGACGGAGGCGUCUCCUGGAUCAGGGCAAGGAUAA ((.(((..(((((....))))).((((((((((((((.((.....)))))))).)))))(((((((.....))))))).....((((....)))).....)))))).))..... ( -50.10) >consensus CUCGCCGCGGCCCAAUCGGGCCACUGGCGGCUAUCUGCUGACGGGCACAAGUGCGCCGCGUGACGAUGAGUUCGUCGCAGGACGGAGGCAUCUCCUGGAUCAGGGCAAGGAUAA ...((((((((((....)))))....)))))((((((((..((.(((....))).((..(((((((.....))))))).)).))..)))...(((((...)))))...))))). (-49.02 = -48.70 + -0.32)

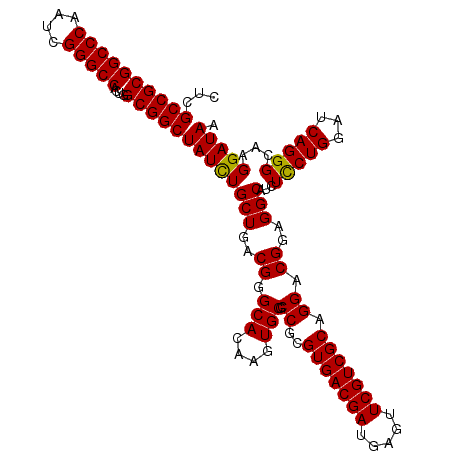
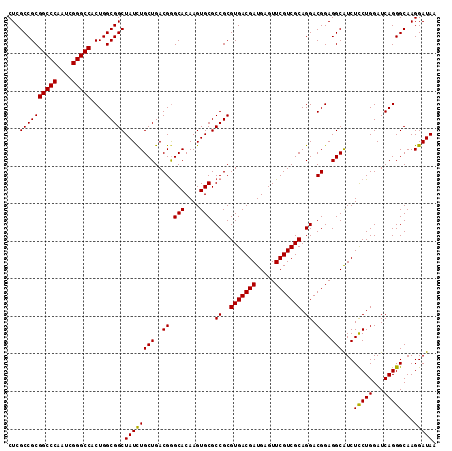
| Location | 5,924,412 – 5,924,532 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.22 |
| Mean single sequence MFE | -55.25 |
| Consensus MFE | -53.14 |
| Energy contribution | -53.37 |
| Covariance contribution | 0.22 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.16 |
| Structure conservation index | 0.96 |
| SVM decision value | 1.32 |
| SVM RNA-class probability | 0.941998 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 5924412 120 + 20766785 CUGCGACGAACUCAUCGUCACGCGGCGCACUUGUGCCCGUCAGCAGAUAGCCGCCAGUGGCCCGAUUGGGCCGCGGCGAGCGUAGGCGCGUCCACAGAUUGGCGAGUGUCCUUCUGGUCG (((((((((.....)))))(((.(((((....))))))))..))))...((((((.(((((((....))))))))))).))(.((((((..(((.....)))...))).))).)...... ( -56.20) >DroSec_CAF1 66348 120 + 1 CUGCGACGAACUCAUCGUCACGCGGCGCACUUGUGCCCGUCAGCAGAUAGCCGCCAGUGGCCCGAUUGGGCCGCGGCGAGCGUAGGCGCGUCCACAGAUUGGCGAGUGUCCUUCUGGUCG (((((((((.....)))))(((.(((((....))))))))..))))...((((((.(((((((....))))))))))).))(.((((((..(((.....)))...))).))).)...... ( -56.20) >DroSim_CAF1 60147 120 + 1 CUGCGACGAACUCAUCGUCACGCGGCGCACUUGUGCUCGUCAGCAGAUAGCCGCCAGUGGCCCGAUUGGGCCGCGGCGAGCGUAGGCGCGUCCACAGAUUGGCGAGUGUCCUUCUGAUCG ..(.(((((.....))))).)(.(((((.((((((((((((.((.....)).....(((((((....))))))))))))))))))).)))))).((((..(((....)))..)))).... ( -55.00) >DroEre_CAF1 57272 120 + 1 CUGCGACGAACUCAUCGUCACGCGGCGCACUUGUGCCCGUCAGCAGAUAGCCGCCAGUGGCCCGAUUGGGCCGCGGCGAGCGUAGGCGCGUCCACAGAUUGGCGAGUGUCCUUCUGGUCG (((((((((.....)))))(((.(((((....))))))))..))))...((((((.(((((((....))))))))))).))(.((((((..(((.....)))...))).))).)...... ( -56.20) >DroYak_CAF1 60460 120 + 1 CUGCGACGAACUCAUCGUCACGCGGCGCACUUGUGCCCGUCAGCAAAUAGCCGCCAGUGGCCCGAUUGGGCCGCGGCGAGCGUAGGCGCGUCCACAGAUUGGCGAGUGUCCUUCUGGUCG ....(((((.....)))))(((((((((....))))).....((.....((((((.(((((((....))))))))))).))....))))))...((((..(((....)))..)))).... ( -54.10) >DroAna_CAF1 64524 120 + 1 CUGCAACGAGCUCAUCGUCGCGCGGCGCACUUGUGCCCGUCAGCAGGUAGCCGCCAGUGGCCCGAUUGGGCCGCGGCGAACGUAGGCGCGUCCACAGAUUGGCGAGUGUCCUUCUUGUUG ...(((((((....((((((((((((..((((((........)))))).)))))....(((((....)))))))))))).....(((((..(((.....)))...)))))...))))))) ( -53.80) >consensus CUGCGACGAACUCAUCGUCACGCGGCGCACUUGUGCCCGUCAGCAGAUAGCCGCCAGUGGCCCGAUUGGGCCGCGGCGAGCGUAGGCGCGUCCACAGAUUGGCGAGUGUCCUUCUGGUCG (((((((((.....)))))(((.(((((....))))))))..))))...((((((.(((((((....))))))))))).))(.((((((..(((.....)))...))).))).)...... (-53.14 = -53.37 + 0.22)
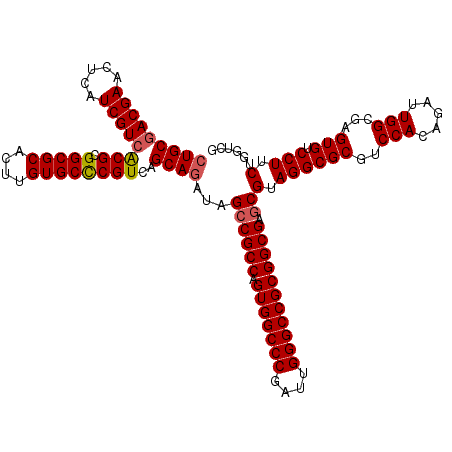
| Location | 5,924,412 – 5,924,532 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.22 |
| Mean single sequence MFE | -51.27 |
| Consensus MFE | -51.22 |
| Energy contribution | -50.67 |
| Covariance contribution | -0.55 |
| Combinations/Pair | 1.11 |
| Mean z-score | -0.82 |
| Structure conservation index | 1.00 |
| SVM decision value | 0.82 |
| SVM RNA-class probability | 0.858540 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 5924412 120 - 20766785 CGACCAGAAGGACACUCGCCAAUCUGUGGACGCGCCUACGCUCGCCGCGGCCCAAUCGGGCCACUGGCGGCUAUCUGCUGACGGGCACAAGUGCGCCGCGUGACGAUGAGUUCGUCGCAG ....(((..(((.(..(((((....((((.((.((....)).))))))(((((....)))))..)))))..).))).))).((((((....))).))).(((((((.....))))))).. ( -51.90) >DroSec_CAF1 66348 120 - 1 CGACCAGAAGGACACUCGCCAAUCUGUGGACGCGCCUACGCUCGCCGCGGCCCAAUCGGGCCACUGGCGGCUAUCUGCUGACGGGCACAAGUGCGCCGCGUGACGAUGAGUUCGUCGCAG ....(((..(((.(..(((((....((((.((.((....)).))))))(((((....)))))..)))))..).))).))).((((((....))).))).(((((((.....))))))).. ( -51.90) >DroSim_CAF1 60147 120 - 1 CGAUCAGAAGGACACUCGCCAAUCUGUGGACGCGCCUACGCUCGCCGCGGCCCAAUCGGGCCACUGGCGGCUAUCUGCUGACGAGCACAAGUGCGCCGCGUGACGAUGAGUUCGUCGCAG .........((.(....))).....((((.(((((....((((((((.(((((....))))).).)))(((.....)))...))))....)))))))))(((((((.....))))))).. ( -52.00) >DroEre_CAF1 57272 120 - 1 CGACCAGAAGGACACUCGCCAAUCUGUGGACGCGCCUACGCUCGCCGCGGCCCAAUCGGGCCACUGGCGGCUAUCUGCUGACGGGCACAAGUGCGCCGCGUGACGAUGAGUUCGUCGCAG ....(((..(((.(..(((((....((((.((.((....)).))))))(((((....)))))..)))))..).))).))).((((((....))).))).(((((((.....))))))).. ( -51.90) >DroYak_CAF1 60460 120 - 1 CGACCAGAAGGACACUCGCCAAUCUGUGGACGCGCCUACGCUCGCCGCGGCCCAAUCGGGCCACUGGCGGCUAUUUGCUGACGGGCACAAGUGCGCCGCGUGACGAUGAGUUCGUCGCAG ...((....))..............((((.(((((....((((((((.(((((....))))).).)))(((.....)))...))))....)))))))))(((((((.....))))))).. ( -51.20) >DroAna_CAF1 64524 120 - 1 CAACAAGAAGGACACUCGCCAAUCUGUGGACGCGCCUACGUUCGCCGCGGCCCAAUCGGGCCACUGGCGGCUACCUGCUGACGGGCACAAGUGCGCCGCGCGACGAUGAGCUCGUUGCAG .....((.(((....((((((....(((((((......)))))))...(((((....)))))..))))))...))).))(.((((((....))).))))(((((((.....))))))).. ( -48.70) >consensus CGACCAGAAGGACACUCGCCAAUCUGUGGACGCGCCUACGCUCGCCGCGGCCCAAUCGGGCCACUGGCGGCUAUCUGCUGACGGGCACAAGUGCGCCGCGUGACGAUGAGUUCGUCGCAG .........((.(....))).....((((.(((((....((((((((.(((((....))))).).)))(((.....)))...))))....)))))))))(((((((.....))))))).. (-51.22 = -50.67 + -0.55)

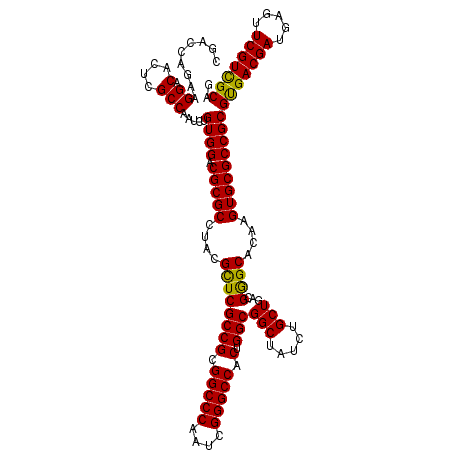
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:07:48 2006