
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 1,403,425 – 1,403,880 |
| Length | 455 |
| Max. P | 0.999955 |

| Location | 1,403,425 – 1,403,536 |
|---|---|
| Length | 111 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.36 |
| Mean single sequence MFE | -39.80 |
| Consensus MFE | -32.74 |
| Energy contribution | -32.78 |
| Covariance contribution | 0.04 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.13 |
| SVM RNA-class probability | 0.919511 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1403425 111 - 20766785 GCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGUUUGUUAUGUGCCAGUUUAUCAGCUGCUUAUGCAACGGCAGCACCCAUCCCAGGGUGGGUUCCUCUCCCA---------AU ((((((..((((((((((((((.((((((.....)))))).)))))).)))))..)))..)))))).........((.((.(((((((....)))))))....)).)).---------.. ( -41.60) >DroSec_CAF1 29257 111 - 1 GCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGUUUGUUAUGCGCCAGUUUAUCAGCUGCUUAUGCAACGGCAGCGGCGGUCCCAGGGUGGGCUCCUCUCCCA---------AU ((((((..((((((((((((((.((((((.....)))))).)))))).)))))..)))..)))))).........((.((.((.(((((......))))))).)).)).---------.. ( -42.40) >DroSim_CAF1 34240 111 - 1 GCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGUUUGUUAUGCGCCAGUUUAUCAGCUGCUUAUGCAACGGCAGCGGCGGUCCCAGGGUGGGCUCCUCUCCCA---------AU ((((((..((((((((((((((.((((((.....)))))).)))))).)))))..)))..)))))).........((.((.((.(((((......))))))).)).)).---------.. ( -42.40) >DroEre_CAF1 28097 108 - 1 GCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGUUUGCUCUGUGCCGCUUUAUCAGCUGCUUAUGCAACGGCAGC---GCUCCCAGGCUGGGCUCCUAUCCAA---------AU (.(((((.....((((..((((.((((((.....)))))).))))...))))((((.....((((((........))))))---......)))).))))).).......---------.. ( -35.40) >DroYak_CAF1 29886 117 - 1 GCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGUUUGUUAUGUGCCACUUUUUCAGCUGCUUAUGCAACAGCAGU---GGUCCCAGACUGGCCUCCUAUCCCAUAGGAUCCCAU ((((((.....(((((((((((.((((((.....)))))).)))))).))))).......))))))...(((....)))..---((((...))))((..((((((...))))))..)).. ( -37.20) >consensus GCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGUUUGUUAUGUGCCAGUUUAUCAGCUGCUUAUGCAACGGCAGC__CGGUCCCAGGGUGGGCUCCUCUCCCA_________AU ((((((..((((((((((((((.((((((.....)))))).)))))).)))))..)))..))))))...(((....)))........(((.....)))...................... (-32.74 = -32.78 + 0.04)


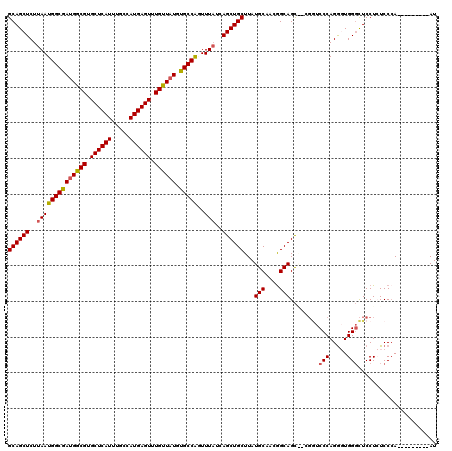
| Location | 1,403,456 – 1,403,576 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.00 |
| Mean single sequence MFE | -32.84 |
| Consensus MFE | -29.77 |
| Energy contribution | -30.17 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.48 |
| Structure conservation index | 0.91 |
| SVM decision value | 3.36 |
| SVM RNA-class probability | 0.999071 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1403456 120 + 20766785 CUGCCGUUGCAUAAGCAGCUGAUAAACUGGCACAUAACAAACUCAUGGCAAAUGAGCACGCCAUCGCCAUUAAGAGCUGCUGCAUUUUAAUUAAUCAAGUUAACAAACGGAGCAAUGCAG ((((.(((((...(((((((..(((..((((..........(((((.....))))).........)))))))..))))))).....((((((.....))))))........))))))))) ( -31.11) >DroSec_CAF1 29288 120 + 1 CUGCCGUUGCAUAAGCAGCUGAUAAACUGGCGCAUAACAAACUCAUGGCAAAUGAGCACGCCAUCGCCAUUAAGAGCUGCUGCAUUUUAAUUAAUCAAGUUAACAAACGGAGCAAUGCAG ((((.(((((...(((((((..(((..(((((.........(((((.....)))))........))))))))..))))))).....((((((.....))))))........))))))))) ( -33.63) >DroSim_CAF1 34271 120 + 1 CUGCCGUUGCAUAAGCAGCUGAUAAACUGGCGCAUAACAAACUCAUGGCAAAUGAGCACGCCAUCGCCAUUAAGAGCUGCUGCAUUUUAAUUAAUCAAGUUAACAAACGGAGCAAUGCAG ((((.(((((...(((((((..(((..(((((.........(((((.....)))))........))))))))..))))))).....((((((.....))))))........))))))))) ( -33.63) >DroEre_CAF1 28125 120 + 1 CUGCCGUUGCAUAAGCAGCUGAUAAAGCGGCACAGAGCAAACUCAUGGCAAAUGAGCACGCCAUCGCCAUUAAGAGCUGCUGCAUUUUAAUUAAUCAAGUUAACAAACGGAGCAAUGCAG ((((.(((((...(((((((..(((...(((...(((....)))(((((..........))))).))).)))..))))))).....((((((.....))))))........))))))))) ( -33.70) >DroYak_CAF1 29923 120 + 1 CUGCUGUUGCAUAAGCAGCUGAAAAAGUGGCACAUAACAAACUCAUGGCAAAUGAGCACGCCAUCGCCAUUAAGAGCUGCUGCAUUUUAAUUAAUCAAGUUAACAAACAGAGCAAUGCAG ((((.(((((...(((((((.....((((((..........(((((.....))))).........))))))...))))))).....((((((.....))))))........))))))))) ( -32.11) >consensus CUGCCGUUGCAUAAGCAGCUGAUAAACUGGCACAUAACAAACUCAUGGCAAAUGAGCACGCCAUCGCCAUUAAGAGCUGCUGCAUUUUAAUUAAUCAAGUUAACAAACGGAGCAAUGCAG ((((.(((((...(((((((..(((..((((..........(((((.....))))).........)))))))..))))))).....((((((.....))))))........))))))))) (-29.77 = -30.17 + 0.40)

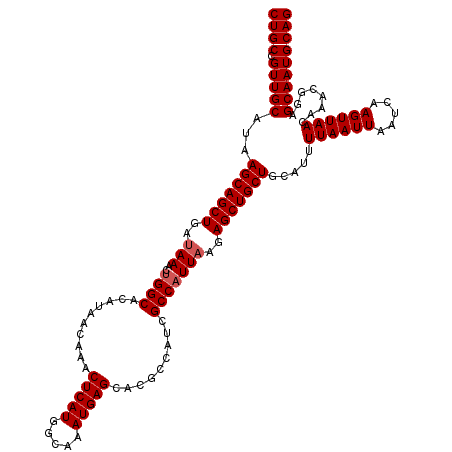
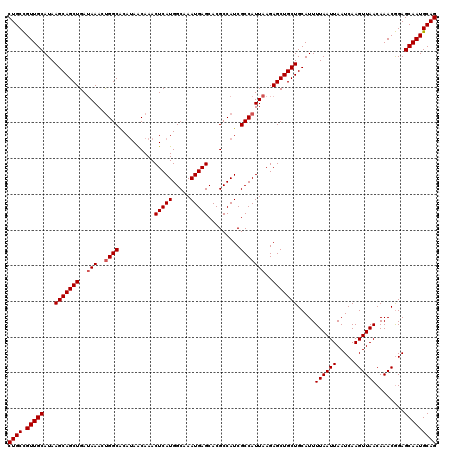
| Location | 1,403,456 – 1,403,576 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.00 |
| Mean single sequence MFE | -37.98 |
| Consensus MFE | -37.64 |
| Energy contribution | -37.32 |
| Covariance contribution | -0.32 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.63 |
| Structure conservation index | 0.99 |
| SVM decision value | 4.72 |
| SVM RNA-class probability | 0.999942 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1403456 120 - 20766785 CUGCAUUGCUCCGUUUGUUAACUUGAUUAAUUAAAAUGCAGCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGUUUGUUAUGUGCCAGUUUAUCAGCUGCUUAUGCAACGGCAG ((((.((((..((((((((((.....)))))..))))).(((((((..((((((((((((((.((((((.....)))))).)))))).)))))..)))..)))))))...))))..)))) ( -37.70) >DroSec_CAF1 29288 120 - 1 CUGCAUUGCUCCGUUUGUUAACUUGAUUAAUUAAAAUGCAGCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGUUUGUUAUGCGCCAGUUUAUCAGCUGCUUAUGCAACGGCAG ((((.((((..((((((((((.....)))))..))))).(((((((..((((((((((((((.((((((.....)))))).)))))).)))))..)))..)))))))...))))..)))) ( -39.80) >DroSim_CAF1 34271 120 - 1 CUGCAUUGCUCCGUUUGUUAACUUGAUUAAUUAAAAUGCAGCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGUUUGUUAUGCGCCAGUUUAUCAGCUGCUUAUGCAACGGCAG ((((.((((..((((((((((.....)))))..))))).(((((((..((((((((((((((.((((((.....)))))).)))))).)))))..)))..)))))))...))))..)))) ( -39.80) >DroEre_CAF1 28125 120 - 1 CUGCAUUGCUCCGUUUGUUAACUUGAUUAAUUAAAAUGCAGCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGUUUGCUCUGUGCCGCUUUAUCAGCUGCUUAUGCAACGGCAG ((((.((((..((((((((((.....)))))..))))).(((((((..((((((((..((((.((((((.....)))))).))))...)))))..)))..)))))))...))))..)))) ( -37.30) >DroYak_CAF1 29923 120 - 1 CUGCAUUGCUCUGUUUGUUAACUUGAUUAAUUAAAAUGCAGCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGUUUGUUAUGUGCCACUUUUUCAGCUGCUUAUGCAACAGCAG ((((((((.((.(((....)))..)).)))).....((((((((((.....(((((((((((.((((((.....)))))).)))))).))))).......))))))...))))...)))) ( -35.30) >consensus CUGCAUUGCUCCGUUUGUUAACUUGAUUAAUUAAAAUGCAGCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGUUUGUUAUGUGCCAGUUUAUCAGCUGCUUAUGCAACGGCAG ((((.((((..((((((((((.....)))))..))))).(((((((..((((((((((((((.((((((.....)))))).)))))).)))))..)))..)))))))...))))..)))) (-37.64 = -37.32 + -0.32)

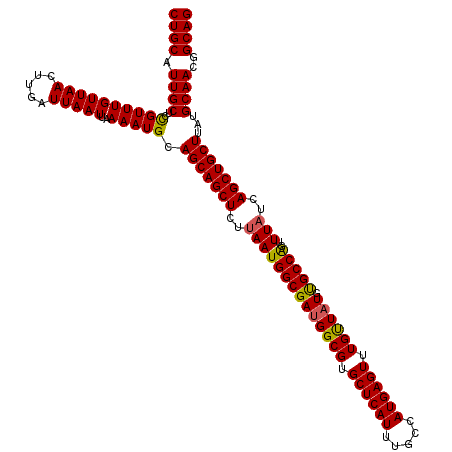
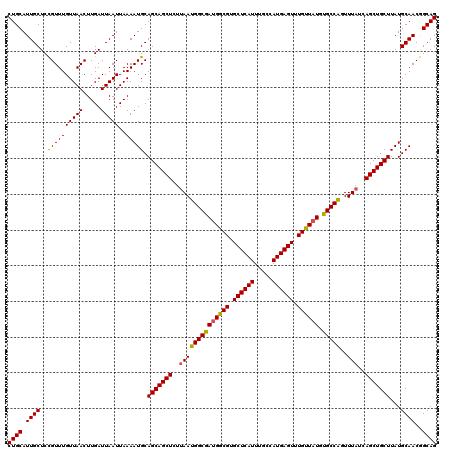
| Location | 1,403,496 – 1,403,616 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 99.33 |
| Mean single sequence MFE | -36.62 |
| Consensus MFE | -36.34 |
| Energy contribution | -36.18 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.69 |
| Structure conservation index | 0.99 |
| SVM decision value | 3.76 |
| SVM RNA-class probability | 0.999593 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1403496 120 + 20766785 ACUCAUGGCAAAUGAGCACGCCAUCGCCAUUAAGAGCUGCUGCAUUUUAAUUAAUCAAGUUAACAAACGGAGCAAUGCAGCUGCAAUGGCGGCAGCCUCAUCUGUCAACAACAAUGCGCG .....(((((.(((((...((..((((((((....((.((((((((((((((.....))))))..........)))))))).))))))))))..))))))).)))))............. ( -36.10) >DroSec_CAF1 29328 120 + 1 ACUCAUGGCAAAUGAGCACGCCAUCGCCAUUAAGAGCUGCUGCAUUUUAAUUAAUCAAGUUAACAAACGGAGCAAUGCAGCUGCAAUGGCGGCAGCCUCAUCUGUCAACAACAAUGCGCG .....(((((.(((((...((..((((((((....((.((((((((((((((.....))))))..........)))))))).))))))))))..))))))).)))))............. ( -36.10) >DroSim_CAF1 34311 120 + 1 ACUCAUGGCAAAUGAGCACGCCAUCGCCAUUAAGAGCUGCUGCAUUUUAAUUAAUCAAGUUAACAAACGGAGCAAUGCAGCUGCAAUGGCGGCAGCCUCAUCUGUCAACAACAAUGCGCG .....(((((.(((((...((..((((((((....((.((((((((((((((.....))))))..........)))))))).))))))))))..))))))).)))))............. ( -36.10) >DroEre_CAF1 28165 120 + 1 ACUCAUGGCAAAUGAGCACGCCAUCGCCAUUAAGAGCUGCUGCAUUUUAAUUAAUCAAGUUAACAAACGGAGCAAUGCAGCUGCGAUGGCGGCAGCCUCAUCUGUCAACAACAAUGCGCG .....(((((.((((((.(((((((((.......(((((((((...((((((.....))))))(....)..)))..)))))))))))))))...).))))).)))))............. ( -38.71) >DroYak_CAF1 29963 120 + 1 ACUCAUGGCAAAUGAGCACGCCAUCGCCAUUAAGAGCUGCUGCAUUUUAAUUAAUCAAGUUAACAAACAGAGCAAUGCAGCUGCAAUGGCGGCAGCCUCAUCUGUCAACAACAAUGCGCG .....(((((.(((((...((..((((((((....((.((((((((((((((.....))))))..........)))))))).))))))))))..))))))).)))))............. ( -36.10) >consensus ACUCAUGGCAAAUGAGCACGCCAUCGCCAUUAAGAGCUGCUGCAUUUUAAUUAAUCAAGUUAACAAACGGAGCAAUGCAGCUGCAAUGGCGGCAGCCUCAUCUGUCAACAACAAUGCGCG .....(((((.(((((...((..((((((((....((.((((((((((((((.....))))))..........)))))))).))))))))))..))))))).)))))............. (-36.34 = -36.18 + -0.16)

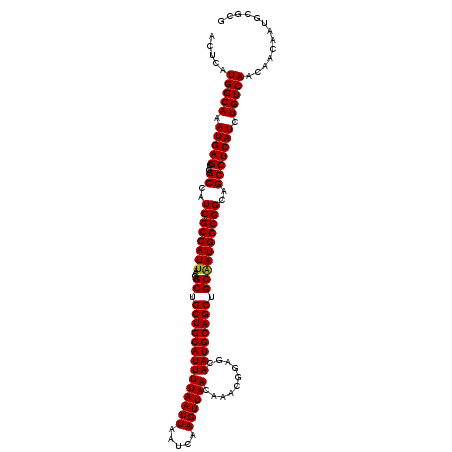
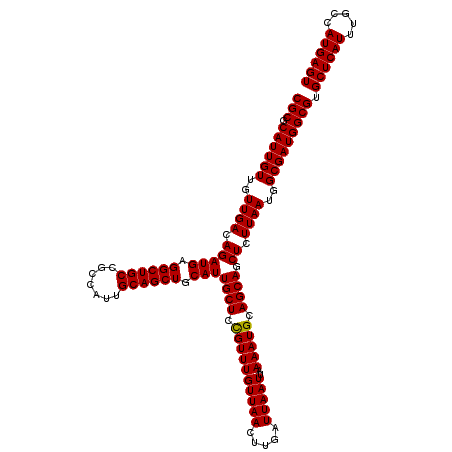
| Location | 1,403,496 – 1,403,616 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 99.33 |
| Mean single sequence MFE | -38.56 |
| Consensus MFE | -38.12 |
| Energy contribution | -37.96 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.02 |
| Mean z-score | -1.63 |
| Structure conservation index | 0.99 |
| SVM decision value | 1.43 |
| SVM RNA-class probability | 0.953417 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1403496 120 - 20766785 CGCGCAUUGUUGUUGACAGAUGAGGCUGCCGCCAUUGCAGCUGCAUUGCUCCGUUUGUUAACUUGAUUAAUUAAAAUGCAGCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGU (((.((((((..((((.(((((.((((((.......)))))).)))((((.((((((((((.....)))))..))))).)))).)).))))..))))))))).((((((.....)))))) ( -38.40) >DroSec_CAF1 29328 120 - 1 CGCGCAUUGUUGUUGACAGAUGAGGCUGCCGCCAUUGCAGCUGCAUUGCUCCGUUUGUUAACUUGAUUAAUUAAAAUGCAGCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGU (((.((((((..((((.(((((.((((((.......)))))).)))((((.((((((((((.....)))))..))))).)))).)).))))..))))))))).((((((.....)))))) ( -38.40) >DroSim_CAF1 34311 120 - 1 CGCGCAUUGUUGUUGACAGAUGAGGCUGCCGCCAUUGCAGCUGCAUUGCUCCGUUUGUUAACUUGAUUAAUUAAAAUGCAGCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGU (((.((((((..((((.(((((.((((((.......)))))).)))((((.((((((((((.....)))))..))))).)))).)).))))..))))))))).((((((.....)))))) ( -38.40) >DroEre_CAF1 28165 120 - 1 CGCGCAUUGUUGUUGACAGAUGAGGCUGCCGCCAUCGCAGCUGCAUUGCUCCGUUUGUUAACUUGAUUAAUUAAAAUGCAGCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGU .............((.((((((((.(...(((((((((((((((..(((....((.(((((.....))))).))...))))))))).......))))))))).))))))))).))..... ( -38.61) >DroYak_CAF1 29963 120 - 1 CGCGCAUUGUUGUUGACAGAUGAGGCUGCCGCCAUUGCAGCUGCAUUGCUCUGUUUGUUAACUUGAUUAAUUAAAAUGCAGCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGU (((.((((((..((((..((((.((((((.......)))))).))))(((((((...((((.........))))...)))).)))..))))..))))))))).((((((.....)))))) ( -39.00) >consensus CGCGCAUUGUUGUUGACAGAUGAGGCUGCCGCCAUUGCAGCUGCAUUGCUCCGUUUGUUAACUUGAUUAAUUAAAAUGCAGCAGCUCUUAAUGGCGAUGGCGUGCUCAUUUGCCAUGAGU (((.((((((..((((.(((((.((((((.......)))))).)))((((.((((((((((.....)))))..))))).)))).)).))))..))))))))).((((((.....)))))) (-38.12 = -37.96 + -0.16)

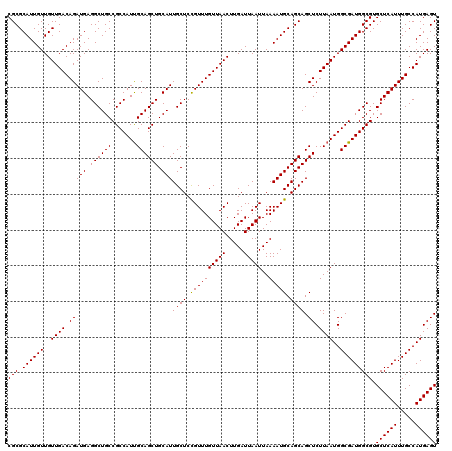
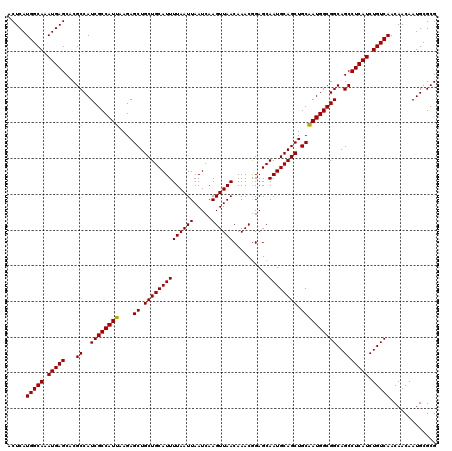
| Location | 1,403,576 – 1,403,690 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.36 |
| Mean single sequence MFE | -36.31 |
| Consensus MFE | -28.28 |
| Energy contribution | -28.72 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.69 |
| Structure conservation index | 0.78 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.510095 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1403576 114 + 20766785 CUGCAAUGGCGGCAGCCUCAUCUGUCAACAACAAUGCGCGCCGCAAACUAAUUACAUGCAACUCGACGGAAAGGGGAAGCCGAAACCCAAAUCCCAAUUGA------GGCGGCCGCGCAU .....((((((((.((((((((((((........((((...))))............(.....)))))))..(((..........)))..........)))------))).))))).))) ( -35.90) >DroSec_CAF1 29408 114 + 1 CUGCAAUGGCGGCAGCCUCAUCUGUCAACAACAAUGCGCGCCGCAAACUAAUUACAUGCAACUCGACGGAUAGGGGAAGCCGAAACCCAAAUCCCAAUUGA------GGCGGCCGCGCAU .....((((((((.((((((((((((........((((...))))............(.....)))))))).(((..........)))...........))------))).))))).))) ( -36.20) >DroSim_CAF1 34391 114 + 1 CUGCAAUGGCGGCAGCCUCAUCUGUCAACAACAAUGCGCGCCGCAAACUAAUUACAUGCAACUCGGCGGAUAGGGGAAGCCGAAACCCAAAUCCCAAUUGA------GGCGGCCGCGCAU .....((((((((.((((((..((...............((((....................))))((((..(((.........)))..))))))..)))------))).))))).))) ( -37.35) >DroEre_CAF1 28245 119 + 1 CUGCGAUGGCGGCAGCCUCAUCUGUCAACAACAAUGCGCGCCGCAAACUAAUUACAUGCAACUCGACGGAU-GGGGAAGCCGAAACCCAAAUCCCAAUUGAGUGUGAGGCGGCCGCCCAU .......((((((.(((((((.............((((...))))...............((((((.((((-(((..........))))..)))...))))))))))))).))))))... ( -41.90) >DroYak_CAF1 30043 114 + 1 CUGCAAUGGCGGCAGCCUCAUCUGUCAACAACAAUGCGCGCCGCAAACUAAUUACAUGCAACUCGACGGAUAGGGGAAGCCGAAACCCAAAUCCCAAUUGA------CGAGGCCGCCCAU ..((....))(((.(((((....(((((......((((...))))......................((((..(((.........)))..))))...))))------)))))).)))... ( -30.20) >consensus CUGCAAUGGCGGCAGCCUCAUCUGUCAACAACAAUGCGCGCCGCAAACUAAUUACAUGCAACUCGACGGAUAGGGGAAGCCGAAACCCAAAUCCCAAUUGA______GGCGGCCGCGCAU .....((((((((.(((..(((((((........((((...))))............(.....)))))))).(((..........)))...................))).))))).))) (-28.28 = -28.72 + 0.44)

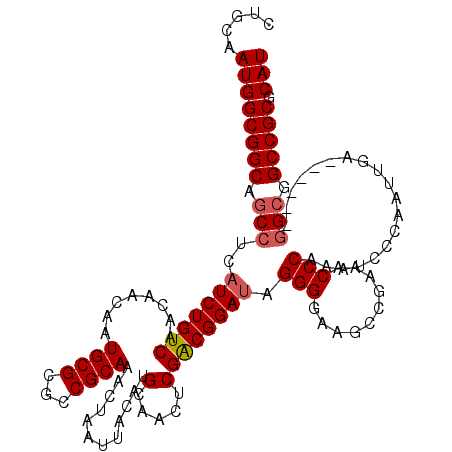
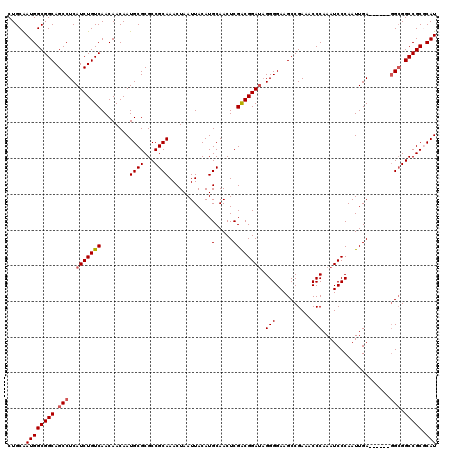
| Location | 1,403,690 – 1,403,800 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.58 |
| Mean single sequence MFE | -37.10 |
| Consensus MFE | -30.54 |
| Energy contribution | -30.78 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.14 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.87 |
| SVM RNA-class probability | 0.980783 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1403690 110 + 20766785 UAUUGUUGUUCGAUGUGGAGCUCGGCUUUGCA----------UUUGUGCAUAUGUGCGAUUACCAAAUUAGAUUUCUGUCUAAUUAUGGCAGUGGGACUGCAGCGCAAGCAGAGCGAGUC (((((.....)))))....(((((.((((((.----------.((((((....((.(.(((.(((((((((((....)))))))).))).))).).))....))))))))))))))))). ( -37.50) >DroSec_CAF1 29522 110 + 1 UAUUGUUGUUCGAUGCGAAGCUCGGCUUUGCA----------UAUGUGCAUAUGUGCGAUUACCAAAUUAGAUUUCUGUCUAAUUAUGGCAGUGGGACUGCAGCGCAAGCAGAGCGAGUC .....((((((.(((((((((...))))))))----------)...((((...((.(.(((.(((((((((((....)))))))).))).))).).))))))((....)).))))))... ( -38.40) >DroSim_CAF1 34505 110 + 1 UAUUGUUGUUCGAUGCGAAGCUCGGCUUUGCA----------UAUGUGCAUAUGUGCGAUUACCAAAUUAGAUUUCUGUCUAAUUAUGGCAGUGGGACUGCAGCGCAAGCAGAGCGAGUC .....((((((.(((((((((...))))))))----------)...((((...((.(.(((.(((((((((((....)))))))).))).))).).))))))((....)).))))))... ( -38.40) >DroEre_CAF1 28364 120 + 1 UAUUGUUGUUCGAUGUGCAGCUCGGCUCGGCUCGGCAUCGAGUAUGUGCAUAUGUGCGAUUACCAAAUUAGAUUUCUGUCUAAUUAUGGCAGUGGGACUGCAGCGCCAGCAGAGCGCGUC ...........(((((((.(((.(((.(.(((((....)))))...((((...((.(.(((.(((((((((((....)))))))).))).))).).))))))).))))))...))))))) ( -40.80) >DroYak_CAF1 30157 110 + 1 UAUUGUUGUUCGAUGUGGAACUUGGGUUUGCU----------UAUGUGCAUAUGUGCGAUUACCAAAUUAGAUUUCUGUCUAAUUAUGGCAGUGGGACUGCAGUGCAAGCAGAGCGAGUC .......((((......))))...(..(((((----------(...(((((..((.(.(((.(((((((((((....)))))))).))).))).).))....)))))....))))))..) ( -30.40) >consensus UAUUGUUGUUCGAUGUGAAGCUCGGCUUUGCA__________UAUGUGCAUAUGUGCGAUUACCAAAUUAGAUUUCUGUCUAAUUAUGGCAGUGGGACUGCAGCGCAAGCAGAGCGAGUC .....((((((...(((((((...)))))))...............((((...((.(.(((.(((((((((((....)))))))).))).))).).))))))((....)).))))))... (-30.54 = -30.78 + 0.24)

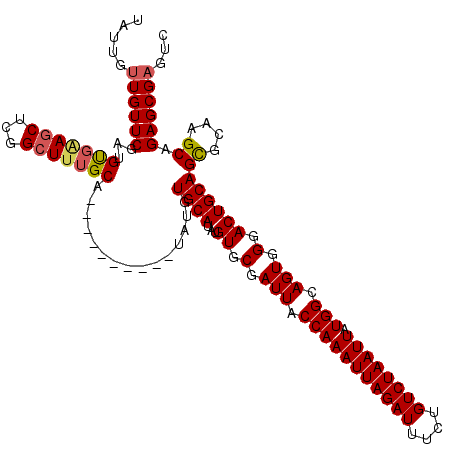
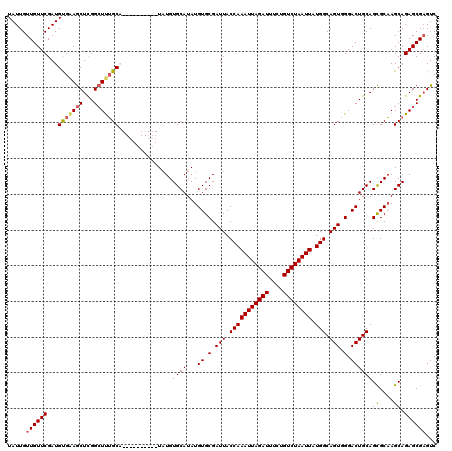
| Location | 1,403,690 – 1,403,800 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.58 |
| Mean single sequence MFE | -27.64 |
| Consensus MFE | -24.44 |
| Energy contribution | -24.20 |
| Covariance contribution | -0.24 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.12 |
| Structure conservation index | 0.88 |
| SVM decision value | 2.74 |
| SVM RNA-class probability | 0.996742 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1403690 110 - 20766785 GACUCGCUCUGCUUGCGCUGCAGUCCCACUGCCAUAAUUAGACAGAAAUCUAAUUUGGUAAUCGCACAUAUGCACAAA----------UGCAAAGCCGAGCUCCACAUCGAACAACAAUA .....((((.(((((((..((((.....))))((.(((((((......))))))))).....))).....((((....----------)))))))).))))................... ( -26.30) >DroSec_CAF1 29522 110 - 1 GACUCGCUCUGCUUGCGCUGCAGUCCCACUGCCAUAAUUAGACAGAAAUCUAAUUUGGUAAUCGCACAUAUGCACAUA----------UGCAAAGCCGAGCUUCGCAUCGAACAACAAUA .....((...(((((.((((((.......(((((.(((((((......))))))))))))...(((....))).....----------)))..))))))))...)).............. ( -27.30) >DroSim_CAF1 34505 110 - 1 GACUCGCUCUGCUUGCGCUGCAGUCCCACUGCCAUAAUUAGACAGAAAUCUAAUUUGGUAAUCGCACAUAUGCACAUA----------UGCAAAGCCGAGCUUCGCAUCGAACAACAAUA .....((...(((((.((((((.......(((((.(((((((......))))))))))))...(((....))).....----------)))..))))))))...)).............. ( -27.30) >DroEre_CAF1 28364 120 - 1 GACGCGCUCUGCUGGCGCUGCAGUCCCACUGCCAUAAUUAGACAGAAAUCUAAUUUGGUAAUCGCACAUAUGCACAUACUCGAUGCCGAGCCGAGCCGAGCUGCACAUCGAACAACAAUA ...((((((.((((((.(.(((.((....(((((.(((((((......))))))))))))...(((....)))........))))).).))).))).)))).))................ ( -35.20) >DroYak_CAF1 30157 110 - 1 GACUCGCUCUGCUUGCACUGCAGUCCCACUGCCAUAAUUAGACAGAAAUCUAAUUUGGUAAUCGCACAUAUGCACAUA----------AGCAAACCCAAGUUCCACAUCGAACAACAAUA ((((.((..((....))..))))))....(((((.(((((((......))))))))))))..........(((.....----------.))).......((((......))))....... ( -22.10) >consensus GACUCGCUCUGCUUGCGCUGCAGUCCCACUGCCAUAAUUAGACAGAAAUCUAAUUUGGUAAUCGCACAUAUGCACAUA__________UGCAAAGCCGAGCUCCACAUCGAACAACAAUA .....((((.((((((.............(((((.(((((((......))))))))))))...(((....)))................)))).)).))))................... (-24.44 = -24.20 + -0.24)

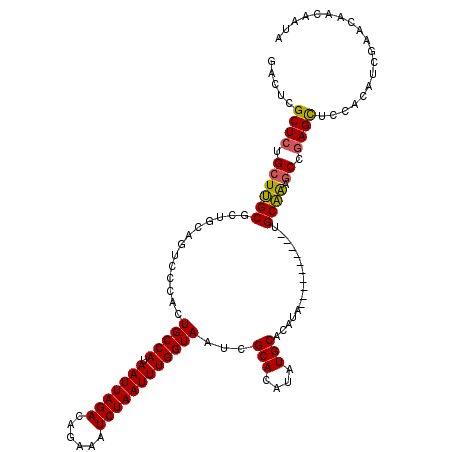
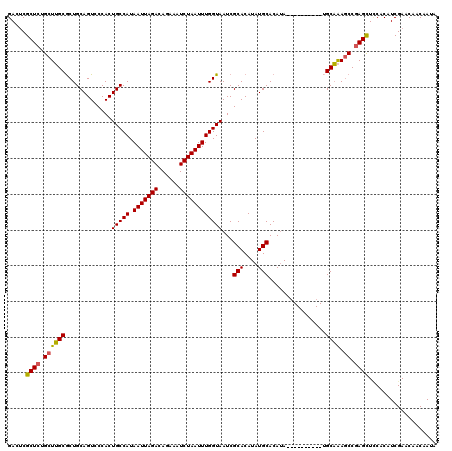
| Location | 1,403,722 – 1,403,840 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.31 |
| Mean single sequence MFE | -39.74 |
| Consensus MFE | -38.70 |
| Energy contribution | -38.54 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.97 |
| Structure conservation index | 0.97 |
| SVM decision value | 4.81 |
| SVM RNA-class probability | 0.999953 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1403722 118 + 20766785 --UUUGUGCAUAUGUGCGAUUACCAAAUUAGAUUUCUGUCUAAUUAUGGCAGUGGGACUGCAGCGCAAGCAGAGCGAGUCCCGAAGAAAUCGCAACUGAAAUUAAUAGCCAUGGCUGCAG --....(((.....(((((((.(((((((((((....)))))))).)))...(((((((((.((....))...)).)))))))....)))))))............(((....)))))). ( -40.90) >DroSec_CAF1 29554 118 + 1 --UAUGUGCAUAUGUGCGAUUACCAAAUUAGAUUUCUGUCUAAUUAUGGCAGUGGGACUGCAGCGCAAGCAGAGCGAGUCCCGAAGAAAUCGCAACUGAAAUUAAUAGCCAUGGCUGCAG --....(((.....(((((((.(((((((((((....)))))))).)))...(((((((((.((....))...)).)))))))....)))))))............(((....)))))). ( -40.90) >DroSim_CAF1 34537 118 + 1 --UAUGUGCAUAUGUGCGAUUACCAAAUUAGAUUUCUGUCUAAUUAUGGCAGUGGGACUGCAGCGCAAGCAGAGCGAGUCCCGAAGAAAUCGCAACUGAAAUUAAUAGCCAUGGCUGCAG --....(((.....(((((((.(((((((((((....)))))))).)))...(((((((((.((....))...)).)))))))....)))))))............(((....)))))). ( -40.90) >DroEre_CAF1 28404 120 + 1 AGUAUGUGCAUAUGUGCGAUUACCAAAUUAGAUUUCUGUCUAAUUAUGGCAGUGGGACUGCAGCGCCAGCAGAGCGCGUCCCGAAGAAAUCGCAACUGAAAUUGAUAGCCAUGGCUGCAG ......(((.....(((((((.(((((((((((....)))))))).)))...((((((....(((((....).))))))))))....)))))))............(((....)))))). ( -37.80) >DroYak_CAF1 30189 118 + 1 --UAUGUGCAUAUGUGCGAUUACCAAAUUAGAUUUCUGUCUAAUUAUGGCAGUGGGACUGCAGUGCAAGCAGAGCGAGUCCCGAAGAAAUCGCAACUGAAAGUAAUAGCCAUGGCUGCAG --....(((.....(((((((.(((((((((((....)))))))).)))...(((((((((..((....))..)).)))))))....)))))))............(((....)))))). ( -38.20) >consensus __UAUGUGCAUAUGUGCGAUUACCAAAUUAGAUUUCUGUCUAAUUAUGGCAGUGGGACUGCAGCGCAAGCAGAGCGAGUCCCGAAGAAAUCGCAACUGAAAUUAAUAGCCAUGGCUGCAG ......(((.....(((((((.(((((((((((....)))))))).)))...(((((((((.((....))...))).))))))....)))))))............(((....)))))). (-38.70 = -38.54 + -0.16)

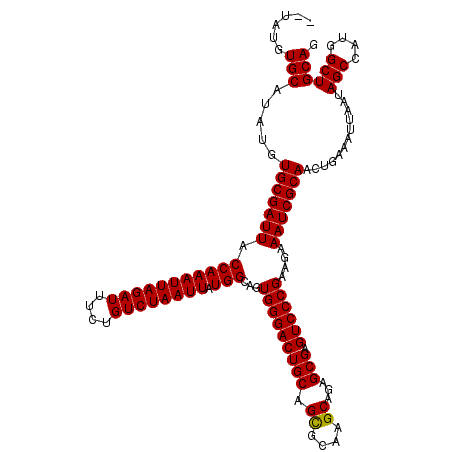
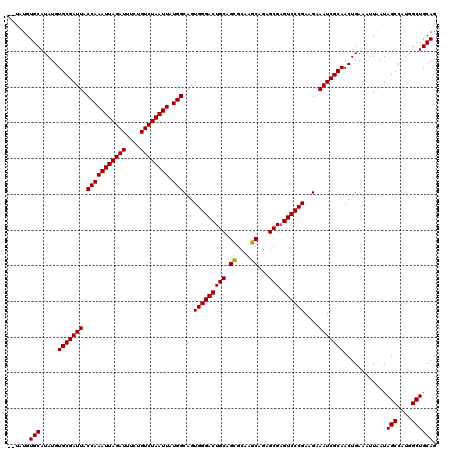
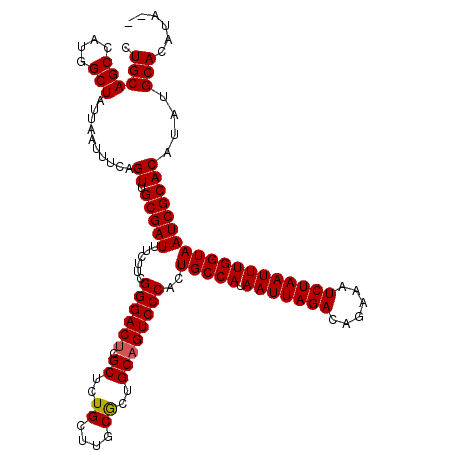
| Location | 1,403,722 – 1,403,840 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.31 |
| Mean single sequence MFE | -37.80 |
| Consensus MFE | -36.36 |
| Energy contribution | -36.40 |
| Covariance contribution | 0.04 |
| Combinations/Pair | 1.03 |
| Mean z-score | -3.61 |
| Structure conservation index | 0.96 |
| SVM decision value | 4.84 |
| SVM RNA-class probability | 0.999955 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1403722 118 - 20766785 CUGCAGCCAUGGCUAUUAAUUUCAGUUGCGAUUUCUUCGGGACUCGCUCUGCUUGCGCUGCAGUCCCACUGCCAUAAUUAGACAGAAAUCUAAUUUGGUAAUCGCACAUAUGCACAAA-- .((((((....)))..........((.(((((......((((((.((...((....)).))))))))..(((((.(((((((......)))))))))))))))))))....)))....-- ( -37.70) >DroSec_CAF1 29554 118 - 1 CUGCAGCCAUGGCUAUUAAUUUCAGUUGCGAUUUCUUCGGGACUCGCUCUGCUUGCGCUGCAGUCCCACUGCCAUAAUUAGACAGAAAUCUAAUUUGGUAAUCGCACAUAUGCACAUA-- .((((((....)))..........((.(((((......((((((.((...((....)).))))))))..(((((.(((((((......)))))))))))))))))))....)))....-- ( -37.70) >DroSim_CAF1 34537 118 - 1 CUGCAGCCAUGGCUAUUAAUUUCAGUUGCGAUUUCUUCGGGACUCGCUCUGCUUGCGCUGCAGUCCCACUGCCAUAAUUAGACAGAAAUCUAAUUUGGUAAUCGCACAUAUGCACAUA-- .((((((....)))..........((.(((((......((((((.((...((....)).))))))))..(((((.(((((((......)))))))))))))))))))....)))....-- ( -37.70) >DroEre_CAF1 28404 120 - 1 CUGCAGCCAUGGCUAUCAAUUUCAGUUGCGAUUUCUUCGGGACGCGCUCUGCUGGCGCUGCAGUCCCACUGCCAUAAUUAGACAGAAAUCUAAUUUGGUAAUCGCACAUAUGCACAUACU .((((((....)))..........((.(((((......((((((((((.....)))))....)))))..(((((.(((((((......)))))))))))))))))))....)))...... ( -38.10) >DroYak_CAF1 30189 118 - 1 CUGCAGCCAUGGCUAUUACUUUCAGUUGCGAUUUCUUCGGGACUCGCUCUGCUUGCACUGCAGUCCCACUGCCAUAAUUAGACAGAAAUCUAAUUUGGUAAUCGCACAUAUGCACAUA-- .((((((....)))..........((.(((((......((((((.((..((....))..))))))))..(((((.(((((((......)))))))))))))))))))....)))....-- ( -37.80) >consensus CUGCAGCCAUGGCUAUUAAUUUCAGUUGCGAUUUCUUCGGGACUCGCUCUGCUUGCGCUGCAGUCCCACUGCCAUAAUUAGACAGAAAUCUAAUUUGGUAAUCGCACAUAUGCACAUA__ .((((((....)))..........((.(((((......((((((.((..((....))..))))))))..(((((.(((((((......)))))))))))))))))))....)))...... (-36.36 = -36.40 + 0.04)

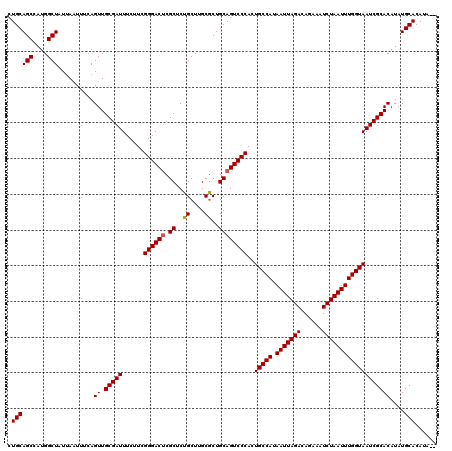
| Location | 1,403,760 – 1,403,880 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.33 |
| Mean single sequence MFE | -36.60 |
| Consensus MFE | -34.34 |
| Energy contribution | -34.78 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.77 |
| Structure conservation index | 0.94 |
| SVM decision value | 4.16 |
| SVM RNA-class probability | 0.999820 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1403760 120 + 20766785 UAAUUAUGGCAGUGGGACUGCAGCGCAAGCAGAGCGAGUCCCGAAGAAAUCGCAACUGAAAUUAAUAGCCAUGGCUGCAGAAGUUUGCGCAAGCGACGAUAAACAAAUGGAUGCAGUAAA ....((((((..(((((((((.((....))...)).)))))))......(((....)))........))))))((((((.....((((....))))(.((......)).).))))))... ( -38.40) >DroSec_CAF1 29592 120 + 1 UAAUUAUGGCAGUGGGACUGCAGCGCAAGCAGAGCGAGUCCCGAAGAAAUCGCAACUGAAAUUAAUAGCCAUGGCUGCAGAAGUUUGCGCAAGCGACGAUAAACAAAUGGAUGCAGUAAA ....((((((..(((((((((.((....))...)).)))))))......(((....)))........))))))((((((.....((((....))))(.((......)).).))))))... ( -38.40) >DroSim_CAF1 34575 120 + 1 UAAUUAUGGCAGUGGGACUGCAGCGCAAGCAGAGCGAGUCCCGAAGAAAUCGCAACUGAAAUUAAUAGCCAUGGCUGCAGAAGUUUGCGCAAGCGACGAUAAACAAAUGGAUGCAGUAAA ....((((((..(((((((((.((....))...)).)))))))......(((....)))........))))))((((((.....((((....))))(.((......)).).))))))... ( -38.40) >DroEre_CAF1 28444 119 + 1 UAAUUAUGGCAGUGGGACUGCAGCGCCAGCAGAGCGCGUCCCGAAGAAAUCGCAACUGAAAUUGAUAGCCAUGGCUGCAGAAGUUU-CGUAAGCGACGAUAAACAAAUGGAUGCAGUAAA ...((((.(((.((((((....(((((....).))))))))))......((((...(((((((..((((....))))....)))))-))...))))...............))).)))). ( -32.60) >DroYak_CAF1 30227 120 + 1 UAAUUAUGGCAGUGGGACUGCAGUGCAAGCAGAGCGAGUCCCGAAGAAAUCGCAACUGAAAGUAAUAGCCAUGGCUGCAGAAGUUUGCGCGAGCGACGAUAAACAAAUGGAUGCAGUAAA ....((((((..(((((((((..((....))..)).)))))))......(((....)))........))))))((((((.....((((....))))(.((......)).).))))))... ( -35.20) >consensus UAAUUAUGGCAGUGGGACUGCAGCGCAAGCAGAGCGAGUCCCGAAGAAAUCGCAACUGAAAUUAAUAGCCAUGGCUGCAGAAGUUUGCGCAAGCGACGAUAAACAAAUGGAUGCAGUAAA ....((((((..(((((((((.((....))...))).))))))......(((....)))........))))))((((((.....((((....))))(.((......)).).))))))... (-34.34 = -34.78 + 0.44)

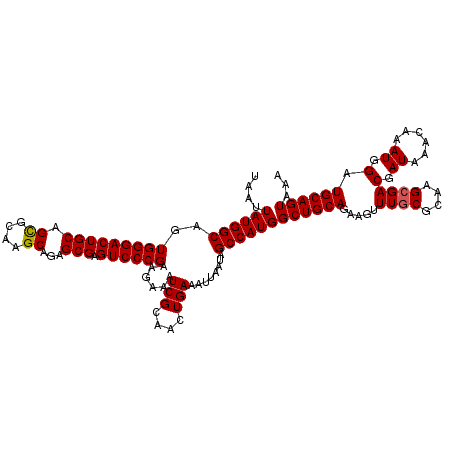
| Location | 1,403,760 – 1,403,880 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.33 |
| Mean single sequence MFE | -29.72 |
| Consensus MFE | -27.36 |
| Energy contribution | -27.44 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.62 |
| Structure conservation index | 0.92 |
| SVM decision value | 1.45 |
| SVM RNA-class probability | 0.955458 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1403760 120 - 20766785 UUUACUGCAUCCAUUUGUUUAUCGUCGCUUGCGCAAACUUCUGCAGCCAUGGCUAUUAAUUUCAGUUGCGAUUUCUUCGGGACUCGCUCUGCUUGCGCUGCAGUCCCACUGCCAUAAUUA ...((((((.......((........))..((((((........(((....)))........(((..((((.(((....))).)))).))).))))))))))))................ ( -30.00) >DroSec_CAF1 29592 120 - 1 UUUACUGCAUCCAUUUGUUUAUCGUCGCUUGCGCAAACUUCUGCAGCCAUGGCUAUUAAUUUCAGUUGCGAUUUCUUCGGGACUCGCUCUGCUUGCGCUGCAGUCCCACUGCCAUAAUUA ...((((((.......((........))..((((((........(((....)))........(((..((((.(((....))).)))).))).))))))))))))................ ( -30.00) >DroSim_CAF1 34575 120 - 1 UUUACUGCAUCCAUUUGUUUAUCGUCGCUUGCGCAAACUUCUGCAGCCAUGGCUAUUAAUUUCAGUUGCGAUUUCUUCGGGACUCGCUCUGCUUGCGCUGCAGUCCCACUGCCAUAAUUA ...((((((.......((........))..((((((........(((....)))........(((..((((.(((....))).)))).))).))))))))))))................ ( -30.00) >DroEre_CAF1 28444 119 - 1 UUUACUGCAUCCAUUUGUUUAUCGUCGCUUACG-AAACUUCUGCAGCCAUGGCUAUCAAUUUCAGUUGCGAUUUCUUCGGGACGCGCUCUGCUGGCGCUGCAGUCCCACUGCCAUAAUUA ....(((((.......(((..((((.....)))-))))...)))))..(((((...((((....))))..........((((((((((.....)))))....)))))...)))))..... ( -29.00) >DroYak_CAF1 30227 120 - 1 UUUACUGCAUCCAUUUGUUUAUCGUCGCUCGCGCAAACUUCUGCAGCCAUGGCUAUUACUUUCAGUUGCGAUUUCUUCGGGACUCGCUCUGCUUGCACUGCAGUCCCACUGCCAUAAUUA ......(((..............(((((..(((((......)))(((....)))..........)).)))))......((((((.((..((....))..))))))))..)))........ ( -29.60) >consensus UUUACUGCAUCCAUUUGUUUAUCGUCGCUUGCGCAAACUUCUGCAGCCAUGGCUAUUAAUUUCAGUUGCGAUUUCUUCGGGACUCGCUCUGCUUGCGCUGCAGUCCCACUGCCAUAAUUA ......(((..............(((((..(((((......)))(((....)))..........)).)))))......((((((.((..((....))..))))))))..)))........ (-27.36 = -27.44 + 0.08)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:29:42 2006