
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 5,400,264 – 5,400,368 |
| Length | 104 |
| Max. P | 0.698135 |

| Location | 5,400,264 – 5,400,368 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | forward |
| Mean pairwise identity | 73.08 |
| Mean single sequence MFE | -35.77 |
| Consensus MFE | -20.07 |
| Energy contribution | -20.83 |
| Covariance contribution | 0.76 |
| Combinations/Pair | 1.53 |
| Mean z-score | -1.52 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.35 |
| SVM RNA-class probability | 0.698135 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 5400264 104 + 20766785 A-ACAG----UGAGUUCGUCCUCUGGCUGGUUACACCCAGCGACGGACUGCAGUUGAACGUUACGCUCAGUCUCUAUGGAAGAGAUGUUGCACUAAGAACCAGCUGUGU .-((((----(..((((((((..(.(((((......))))).).))))(((((.(((.((...)).)))((((((.....)))))).)))))....))))..))))).. ( -35.90) >DroVir_CAF1 704 109 + 1 CCAAGGAUCAGGUGUUUGUCAUUUGGCUGGCAACGCCAAACGAUCAGCUGCAGUUGAAUGCCGCACUGAGACUGUGCGGACGCGAUGCGGCCCUACAGGCUAGCACCAU ..........((((((.(((.((((((.(....)))))))......((((((.(((....((((((.......))))))...)))))))))......))).)))))).. ( -44.00) >DroGri_CAF1 716 109 + 1 CCACGGACCAGGUAUUUGUCAUCUGGCUGGUGACGCCGCACAAUGAACUGCAGCUGAAUGCCACGUUAUGUCUUUAUGGACGGGAUGCAGCCAUCAAGGCGAGCAGCAU .......((.(((((((((((((.....))))))((.(((........))).)).))))))).......((((....))))))..(((.(((.....)))..))).... ( -33.90) >DroSec_CAF1 1939 104 + 1 A-AGAG----CGAGUUCGUCCUCUGGCUGGUUACACCCAGCGAUGGACUGCAGUUGAACGUUACGCUUAGUCUCUAUGGAAGAGAUGUUGCACUAAAAACCAGCUGUGU .-....----..((((((((..((((.((....)).)))).))))))))(((((((....(((.((...((((((.....))))))...))..)))....))))))).. ( -33.40) >DroSim_CAF1 436 104 + 1 A-ACAG----CGAGUUCGUUUUUUGGCUGGUUACACCCAGCGACGGACUGCAGUUGAACGUUACGCUUAGUCUCUAUGGAAGAGAUGUUGCACUAAAAACCAGCUGUGU .-....----..((((((((.....(((((......)))))))))))))(((((((....(((.((...((((((.....))))))...))..)))....))))))).. ( -33.00) >DroYak_CAF1 747 104 + 1 A-ACAG----CGAGUUCGUGCUCUGGCUGGUCACACCCAGCGACGGAAUGCAGCUGAACGUUACGCUCAGUCUCUACGGAAGAGAUAUUGCAGUAAAAACCGGCUGUGU .-((((----(..(((((((((((((((((......)))))..))))..)))..))))).((((((...((((((.....))))))...)).))))......))))).. ( -34.40) >consensus A_ACAG____CGAGUUCGUCCUCUGGCUGGUUACACCCAGCGACGGACUGCAGUUGAACGUUACGCUCAGUCUCUAUGGAAGAGAUGUUGCACUAAAAACCAGCUGUGU ............((((((((..((((.((....)).)))).))))))))(((((((...(((..((...((((((.....))))))...))......)))))))))).. (-20.07 = -20.83 + 0.76)

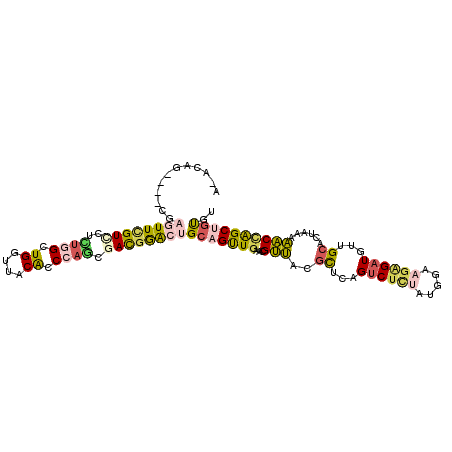
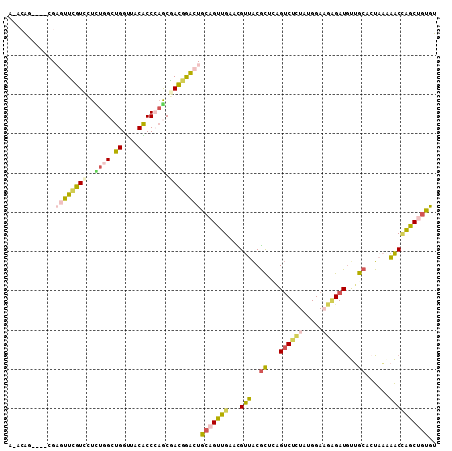
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:03:47 2006