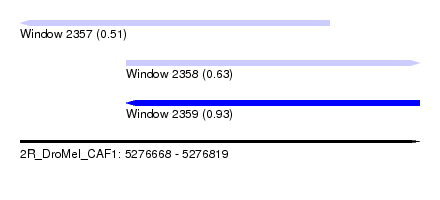
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 5,276,668 – 5,276,819 |
| Length | 151 |
| Max. P | 0.929510 |

| Location | 5,276,668 – 5,276,785 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.06 |
| Mean single sequence MFE | -47.37 |
| Consensus MFE | -22.50 |
| Energy contribution | -23.03 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.31 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.48 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.512226 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 5276668 117 - 20766785 UAUUGGCGGCCAUUGCCCGGGCAGGCACUGUCUUAAGUGGUGGCCGCCAGAUGG---GCCUAAUUUUAGGUUUUUCACUAUCGCUAAUCUCAGCACUGAUUUGGUUGAGUAGCUCCAGCA ...((((((((((..(..((((((...))))))...)..))))))))))((..(---(((((....))))))..))......((((..((((((.(......)))))))))))....... ( -49.50) >DroPse_CAF1 480 117 - 1 UAUUGGCUGCCAAGGCCCGGGUGGCCACGGAUUUCAGCGGAGGCCGCCAGGUGG---GUUUGAUUCUCGGACGCUCGCGUUCGCUGAUCUCUUCGCUGAUAUGGUUGAGCAGCUCCAGCA ....(((((((((((((.....)))).((....((((((((((((((...))))---....((((..((((((....))))))..))))))))))))))..)).))).))))))...... ( -47.30) >DroGri_CAF1 529 120 - 1 UAUUGGCAGCCAUGGCGCGUCCAGCAACAGAUUUUGUAGGUGGUCGCCAGCAGGGUGGCUUCACUCGUGGUCGCUCACAGCUGCUGAUCUCAUCGCUGAUCUGAUUGAGCAGCUCCAGUA ((((((..((..(((((...(((.(.((((...)))).).))).))))))).((((((((........))))))))..(((((((((((..(((...)))..)))).))))))))))))) ( -43.30) >DroEre_CAF1 474 117 - 1 UGUUGGCGGCCAUUGCCCGGGCAGGCACGGUCCUGAGUGGUGGCCGCCAGGCGG---CCUUUAUUUUGGAUUUUUCACCGUCGCUAAUCUCAGCACUGAUUUGGUUGAGCAGCUCCAGCA ...((((((((((..((.((((.......)))).).)..))))))))))(((((---((........))........)))))(((...((((((.(......))))))).)))....... ( -47.00) >DroYak_CAF1 434 117 - 1 UAUUGGCGGCCAUUGCCCGGGCAGGUACGGUUCUGAGUGGUGGCCGCCAGGUGG---UCUUAAUUUUUGAUUUUUCACCAUCGCUGAUCUCAGCACUGAUUUGGUUGAGCAGCUCCAGCA ...((((((((((..((.((((.......)))).).)..))))))))))(((((---(...((........))...))))))((((..((((((.(......)))))))))))....... ( -46.40) >DroPer_CAF1 480 117 - 1 UAUUGGCUGCCAAGGCCCGGGCGGCCACGGAUUUCAGCGGAGGCCGCCAGGUGG---GUUUGAUUCUCGGACGCUCGCGUUCGCUGAUCUCUUCGCUGAUCUGGUUGAGCAGCUCCAGCA ....(((((((((((((.....)))).((((..((((((((((((((...))))---....((((..((((((....))))))..)))))))))))))))))).))).))))))...... ( -50.70) >consensus UAUUGGCGGCCAUGGCCCGGGCAGCCACGGAUUUCAGUGGUGGCCGCCAGGUGG___GCUUAAUUCUCGGUCGCUCACCAUCGCUGAUCUCAGCACUGAUUUGGUUGAGCAGCUCCAGCA ...((((.(((((.(((((........)))......)).))))).))))(.(((..............(((........)))((((..((((..(((.....))))))))))).))).). (-22.50 = -23.03 + 0.53)
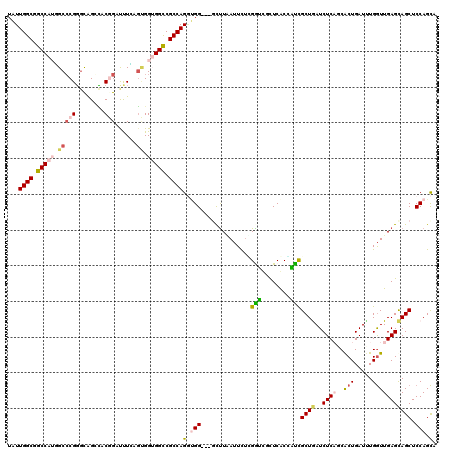
| Location | 5,276,708 – 5,276,819 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 80.24 |
| Mean single sequence MFE | -36.42 |
| Consensus MFE | -24.02 |
| Energy contribution | -23.97 |
| Covariance contribution | -0.05 |
| Combinations/Pair | 1.18 |
| Mean z-score | -1.47 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.632833 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
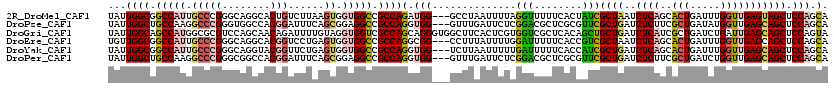
>2R_DroMel_CAF1 5276708 111 + 20766785 UAGUGAAAAACCUAAAAUUAGGCCCAUCUGGCGGCCACCACUUAAGACAGUGCCUGCCCGGGCAAUGGCCGCCAAUAACACCAGGUCUCUCACAGCUCCCGAGCUGAAGAU ..(((.....((((....))))......(((((((((..(((......)))((((....))))..)))))))))....)))......(((..(((((....))))).))). ( -36.70) >DroPse_CAF1 520 111 + 1 ACGCGAGCGUCCGAGAAUCAAACCCACCUGGCGGCCUCCGCUGAAAUCCGUGGCCACCCGGGCCUUGGCAGCCAAUAAGGUCAGAUCUUACACGGCGCCCGAGCUGAAGAU ...((.(((((.((((.((..(((....((((.(((...((.(....).))((((.....))))..))).))))....)))..))))))....))))).)).......... ( -33.40) >DroSim_CAF1 1096 111 + 1 UAGUGAAAAACCUAAAAUUAGGCCCACCUGGCGGCCACCACUCAAGACAGUGCCUGCCCGGGCAAUGGCCGCCAAUAACACCAGGUCUCUCACAGCUCCCGAGCUGAAGAU ..((((.(.((((......(((....)))((((((((..(((......)))((((....))))..)))))))).........)))).).))))((((....))))...... ( -37.90) >DroEre_CAF1 514 111 + 1 CGGUGAAAAAUCCAAAAUAAAGGCCGCCUGGCGGCCACCACUCAGGACCGUGCCUGCCCGGGCAAUGGCCGCCAACAACGUCCGGUCUUUCACAGCUCCCGAGCUGAAGAU .((........)).....((((((((..(((((((((((.....))....(((((....))))).)))))))))........))))))))..(((((....)))))..... ( -42.10) >DroYak_CAF1 474 111 + 1 UGGUGAAAAAUCAAAAAUUAAGACCACCUGGCGGCCACCACUCAGAACCGUACCUGCCCGGGCAAUGGCCGCCAAUAAUGUCAGGUCUCUCACAGCCCCCGAGCUGAAGAU ((((.....)))).......(((((((.(((((((((((...(((........)))....))...))))))))).....))..)))))....((((......))))..... ( -31.40) >DroPer_CAF1 520 111 + 1 ACGCGAGCGUCCGAGAAUCAAACCCACCUGGCGGCCUCCGCUGAAAUCCGUGGCCGCCCGGGCCUUGGCAGCCAAUAAGGUCAGAUCUUACACGGCGCCCGAGCUGAAGAU ...((.(((((.((((.((..(((..((.(((((((..((........)).))))))).))((....)).........)))..))))))....))))).)).......... ( -37.00) >consensus UAGUGAAAAACCGAAAAUUAAGCCCACCUGGCGGCCACCACUCAAAACCGUGCCUGCCCGGGCAAUGGCCGCCAAUAACGUCAGGUCUCUCACAGCUCCCGAGCUGAAGAU ............................((((((((...((........))((((....))))...))))))))..........((((....((((......)))).)))) (-24.02 = -23.97 + -0.05)

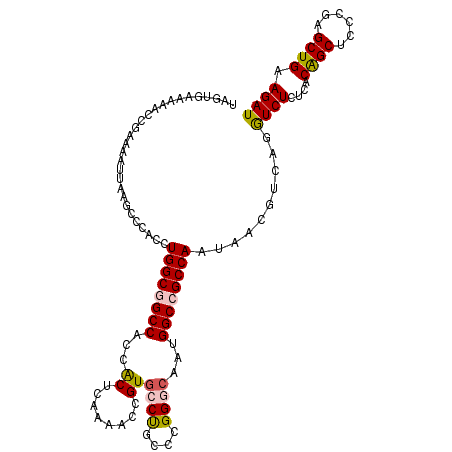

| Location | 5,276,708 – 5,276,819 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 80.24 |
| Mean single sequence MFE | -48.92 |
| Consensus MFE | -31.13 |
| Energy contribution | -32.00 |
| Covariance contribution | 0.87 |
| Combinations/Pair | 1.42 |
| Mean z-score | -2.55 |
| Structure conservation index | 0.64 |
| SVM decision value | 1.20 |
| SVM RNA-class probability | 0.929510 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
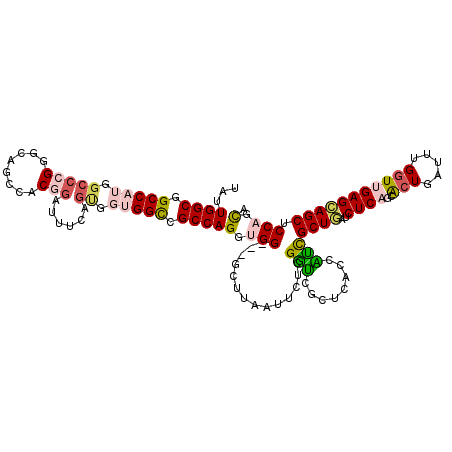
>2R_DroMel_CAF1 5276708 111 - 20766785 AUCUUCAGCUCGGGAGCUGUGAGAGACCUGGUGUUAUUGGCGGCCAUUGCCCGGGCAGGCACUGUCUUAAGUGGUGGCCGCCAGAUGGGCCUAAUUUUAGGUUUUUCACUA ......((((....))))((((((((((((((.(..(((((((((((..(..((((((...))))))...)..)))))))))))..).))).......))))))))))).. ( -52.01) >DroPse_CAF1 520 111 - 1 AUCUUCAGCUCGGGCGCCGUGUAAGAUCUGACCUUAUUGGCUGCCAAGGCCCGGGUGGCCACGGAUUUCAGCGGAGGCCGCCAGGUGGGUUUGAUUCUCGGACGCUCGCGU .......((.(((((((((.(...((((.(((((..(((((.(((..((((.....)))).((........))..))).)))))..))))).))))).))).)))))).)) ( -46.20) >DroSim_CAF1 1096 111 - 1 AUCUUCAGCUCGGGAGCUGUGAGAGACCUGGUGUUAUUGGCGGCCAUUGCCCGGGCAGGCACUGUCUUGAGUGGUGGCCGCCAGGUGGGCCUAAUUUUAGGUUUUUCACUA ......((((....))))(((((((((((((.((((.((((((((((..((.((((((...)))))).).)..))))))))))((....)))))).))))))))))))).. ( -55.40) >DroEre_CAF1 514 111 - 1 AUCUUCAGCUCGGGAGCUGUGAAAGACCGGACGUUGUUGGCGGCCAUUGCCCGGGCAGGCACGGUCCUGAGUGGUGGCCGCCAGGCGGCCUUUAUUUUGGAUUUUUCACCG ......((((....))))(((((((((((((.(((((((((((((((..((.((((.......)))).).)..)))))))))).)))))......))))).)))))))).. ( -48.60) >DroYak_CAF1 474 111 - 1 AUCUUCAGCUCGGGGGCUGUGAGAGACCUGACAUUAUUGGCGGCCAUUGCCCGGGCAGGUACGGUUCUGAGUGGUGGCCGCCAGGUGGUCUUAAUUUUUGAUUUUUCACCA .((..(((((....))))).))((((((........(((((((((((..((.((((.......)))).).)..)))))))))))..))))))......(((....)))... ( -43.50) >DroPer_CAF1 520 111 - 1 AUCUUCAGCUCGGGCGCCGUGUAAGAUCUGACCUUAUUGGCUGCCAAGGCCCGGGCGGCCACGGAUUUCAGCGGAGGCCGCCAGGUGGGUUUGAUUCUCGGACGCUCGCGU .......((.(((((((((.(..((((((.((((....((((.....))))..(((((((.((........))..)))))))))))))))))....).))).)))))).)) ( -47.80) >consensus AUCUUCAGCUCGGGAGCUGUGAAAGACCUGACGUUAUUGGCGGCCAUUGCCCGGGCAGGCACGGUCUUGAGUGGUGGCCGCCAGGUGGGCUUAAUUUUAGGUUUUUCACCA .......(((....))).(((((((((((((.((((((((((((((((((..((((.......))))...))))))))))))).))))).)))......)))))))))).. (-31.13 = -32.00 + 0.87)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:03:14 2006