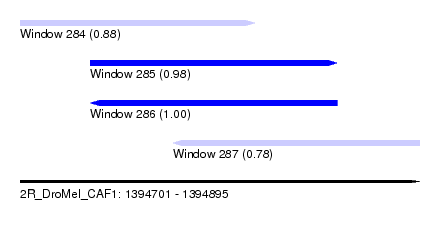
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 1,394,701 – 1,394,895 |
| Length | 194 |
| Max. P | 0.999980 |

| Location | 1,394,701 – 1,394,815 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.18 |
| Mean single sequence MFE | -34.52 |
| Consensus MFE | -25.30 |
| Energy contribution | -25.74 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.54 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.93 |
| SVM RNA-class probability | 0.883174 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1394701 114 + 20766785 AUCGUCCAGCUGUGGCUCUGGUUGUUG------UUGUUGCUGCCGAGCACAAUGGCUUCUAUUGGGCCUAUAGCAUUUAAUUUAUAGUUUAGGCAAACGCCAAAUUUGAUUUAUUGGUCA ...(.((((((((((((((((..((..------((((.(((....)))))))..))..)))..))))).)))))...(((.(((.(((((.(((....))))))))))).))).))).). ( -36.10) >DroSec_CAF1 21165 120 + 1 AUCGUCCAGCUGUGGCUCUGGUUGUUGUUGUUGUUGCUGCUGCCUAGCACAAUGGCUUCUAUUGGGCCUAUAGCAUUUAAUUUAUAGUUUAGGCAAACGCCAAAUUUGAUUUAUUGGUCA ...(.(((((((((((.(..((....((..((((.((((.....))))))))..))....))..)))).)))))...(((.(((.(((((.(((....))))))))))).))).))).). ( -36.60) >DroSim_CAF1 26232 120 + 1 AUCGUCCAGCUGUGGCUCUGCUUGCUGUUGUUGUUGCUGCUGCCUAGCACAAUGGCUUCUAUUGGGCCUAUAGCAUUUAAUUUAUAGUUUAGGCAAACGCCAAAUUUGAUUUAUUGGUCA ...(.(((((((((((((.....(..((..((((.((((.....))))))))..))..)....))))).)))))...(((.(((.(((((.(((....))))))))))).))).))).). ( -35.60) >DroEre_CAF1 20059 110 + 1 AUCGUCCAGCUGUGGCCCUAGUUG---------UUGUUGCUGCAGAGCACAAUGGCUUCUAUUGGGCCUAUAGCAUUUAAUUUAUAGU-UAGGCAAACGCCAAAUUUGAUUUAUUGGUCA ...(.(((((((((((((.(((..---------((((.(((....)))))))..)))......))))).)))))...(((.(((.(((-(.(((....))).))))))).))).))).). ( -37.70) >DroYak_CAF1 21640 111 + 1 AUCGUCCAGCUGUGACUCUAGUUG---------UUUCUGCUGCAGAGCACUAUGGCUUCUAUUGGGCCUAUAGCAUUUAAUUUAUAGUUUAGGCAAACGCCAAAUUUGAUUUAUUGGUCA ...(.(((((((((.((((.((.(---------(....)).)))))))))...(((((.....)))))...)))...(((.(((.(((((.(((....))))))))))).))).))).). ( -26.60) >consensus AUCGUCCAGCUGUGGCUCUGGUUG_UG______UUGCUGCUGCCGAGCACAAUGGCUUCUAUUGGGCCUAUAGCAUUUAAUUUAUAGUUUAGGCAAACGCCAAAUUUGAUUUAUUGGUCA ...(.((((((((((((....................((((....))))((((((...)))))))))).)))))...(((.(((.(((((.(((....))))))))))).))).))).). (-25.30 = -25.74 + 0.44)

| Location | 1,394,735 – 1,394,855 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.50 |
| Mean single sequence MFE | -30.32 |
| Consensus MFE | -28.49 |
| Energy contribution | -28.73 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.94 |
| SVM decision value | 1.95 |
| SVM RNA-class probability | 0.983555 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1394735 120 + 20766785 UGCCGAGCACAAUGGCUUCUAUUGGGCCUAUAGCAUUUAAUUUAUAGUUUAGGCAAACGCCAAAUUUGAUUUAUUGGUCAAGAGACUAUUGACCAUUAUGCUGAGCAUAUUAUUUGGUUG ((((..((.((((((...)))))).))((((((........))))))....))))...(((((((.........(((((((.......))))))).(((((...)))))..))))))).. ( -30.70) >DroSec_CAF1 21205 120 + 1 UGCCUAGCACAAUGGCUUCUAUUGGGCCUAUAGCAUUUAAUUUAUAGUUUAGGCAAACGCCAAAUUUGAUUUAUUGGUCAAGAGGCUAUUGACCAUUAUGCUGAGCAUAUUAUUUGCUUG (((.((((((((((((((((.(((((((((.(((............)))))))).....))))...(((((....)))))))))))))))).......))))).)))............. ( -31.81) >DroSim_CAF1 26272 120 + 1 UGCCUAGCACAAUGGCUUCUAUUGGGCCUAUAGCAUUUAAUUUAUAGUUUAGGCAAACGCCAAAUUUGAUUUAUUGGUCAAGAGGCUAUUGACCAUUAUGCUGAGCAUAUUAUUUGCUUG (((.((((((((((((((((.(((((((((.(((............)))))))).....))))...(((((....)))))))))))))))).......))))).)))............. ( -31.81) >DroEre_CAF1 20090 119 + 1 UGCAGAGCACAAUGGCUUCUAUUGGGCCUAUAGCAUUUAAUUUAUAGU-UAGGCAAACGCCAAAUUUGAUUUAUUGGUCAAGAGGCUGUUGACCAUUAUGCUGAGCAUAUUAUUUGGUUG .........(((((((((((....((((.((((.(((.(((((.....-..(((....)))))))).))))))).)))).)))))))))))((((.(((((...))))).....)))).. ( -30.01) >DroYak_CAF1 21671 120 + 1 UGCAGAGCACUAUGGCUUCUAUUGGGCCUAUAGCAUUUAAUUUAUAGUUUAGGCAAACGCCAAAUUUGAUUUAUUGGUCAAGAGGCUAUUGACUAUUAUGCUGAGCAUAUUAUUUGGCUG .....(((.....(((((.....)))))..((((((.......(((((((.(((....)))...(((((((....))))))))))))))........)))))).............))). ( -27.26) >consensus UGCCGAGCACAAUGGCUUCUAUUGGGCCUAUAGCAUUUAAUUUAUAGUUUAGGCAAACGCCAAAUUUGAUUUAUUGGUCAAGAGGCUAUUGACCAUUAUGCUGAGCAUAUUAUUUGGUUG (((..(((((((((((((((....((((.((((.(((.(((((........(((....)))))))).))))))).)))).))))))))))).......))))..)))............. (-28.49 = -28.73 + 0.24)

| Location | 1,394,735 – 1,394,855 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.50 |
| Mean single sequence MFE | -28.45 |
| Consensus MFE | -27.84 |
| Energy contribution | -28.08 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.03 |
| Mean z-score | -3.07 |
| Structure conservation index | 0.98 |
| SVM decision value | 5.23 |
| SVM RNA-class probability | 0.999980 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1394735 120 - 20766785 CAACCAAAUAAUAUGCUCAGCAUAAUGGUCAAUAGUCUCUUGACCAAUAAAUCAAAUUUGGCGUUUGCCUAAACUAUAAAUUAAAUGCUAUAGGCCCAAUAGAAGCCAUUGUGCUCGGCA .............((((.(((((((((((......(((.(((.((((..........)))).....(((((.(.(((.......))).).))))).))).))).))))))))))).)))) ( -29.20) >DroSec_CAF1 21205 120 - 1 CAAGCAAAUAAUAUGCUCAGCAUAAUGGUCAAUAGCCUCUUGACCAAUAAAUCAAAUUUGGCGUUUGCCUAAACUAUAAAUUAAAUGCUAUAGGCCCAAUAGAAGCCAUUGUGCUAGGCA .............(((..(((((((((((.....(((..((((........))))....)))....(((((.(.(((.......))).).))))).........)))))))))))..))) ( -30.90) >DroSim_CAF1 26272 120 - 1 CAAGCAAAUAAUAUGCUCAGCAUAAUGGUCAAUAGCCUCUUGACCAAUAAAUCAAAUUUGGCGUUUGCCUAAACUAUAAAUUAAAUGCUAUAGGCCCAAUAGAAGCCAUUGUGCUAGGCA .............(((..(((((((((((.....(((..((((........))))....)))....(((((.(.(((.......))).).))))).........)))))))))))..))) ( -30.90) >DroEre_CAF1 20090 119 - 1 CAACCAAAUAAUAUGCUCAGCAUAAUGGUCAACAGCCUCUUGACCAAUAAAUCAAAUUUGGCGUUUGCCUA-ACUAUAAAUUAAAUGCUAUAGGCCCAAUAGAAGCCAUUGUGCUCUGCA .............(((..(((((((((((.....(((..((((........))))....)))....(((((-..................))))).........)))))))))))..))) ( -28.87) >DroYak_CAF1 21671 120 - 1 CAGCCAAAUAAUAUGCUCAGCAUAAUAGUCAAUAGCCUCUUGACCAAUAAAUCAAAUUUGGCGUUUGCCUAAACUAUAAAUUAAAUGCUAUAGGCCCAAUAGAAGCCAUAGUGCUCUGCA ..(((((((..(((((...)))))...(((((.......)))))...........)))))))((((....))))............(((((.(((.........))))))))........ ( -22.40) >consensus CAACCAAAUAAUAUGCUCAGCAUAAUGGUCAAUAGCCUCUUGACCAAUAAAUCAAAUUUGGCGUUUGCCUAAACUAUAAAUUAAAUGCUAUAGGCCCAAUAGAAGCCAUUGUGCUCGGCA .............(((..(((((((((((.....(((..((((........))))....)))....(((((...(((.......)))...))))).........)))))))))))..))) (-27.84 = -28.08 + 0.24)

| Location | 1,394,775 – 1,394,895 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.11 |
| Mean single sequence MFE | -25.74 |
| Consensus MFE | -17.32 |
| Energy contribution | -17.76 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.59 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.55 |
| SVM RNA-class probability | 0.778622 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1394775 120 - 20766785 GGGCCAAAGGGAGAAUCAUUGCUCACUCCAACGUAUAUUACAACCAAAUAAUAUGCUCAGCAUAAUGGUCAAUAGUCUCUUGACCAAUAAAUCAAAUUUGGCGUUUGCCUAAACUAUAAA ..((((((.((((............))))...((((((((........)))))))).........(((((((.......)))))))..........))))))((((....))))...... ( -25.10) >DroSec_CAF1 21245 98 - 1 ----------------------UCACUCCAACGUAUAUUACAAGCAAAUAAUAUGCUCAGCAUAAUGGUCAAUAGCCUCUUGACCAAUAAAUCAAAUUUGGCGUUUGCCUAAACUAUAAA ----------------------..((.((((.((((((((........)))))))).........(((((((.......)))))))...........)))).))................ ( -17.20) >DroSim_CAF1 26312 119 - 1 GGGCCAGAGGG-GCAUCAUUGCUCACUCCAACGUAUAUUACAAGCAAAUAAUAUGCUCAGCAUAAUGGUCAAUAGCCUCUUGACCAAUAAAUCAAAUUUGGCGUUUGCCUAAACUAUAAA ((((..(((((-(((....))))).))).(((((..(((............(((((...))))).(((((((.......)))))))........)))...))))).)))).......... ( -29.50) >DroEre_CAF1 20130 118 - 1 CAGCAGGAGGG-GCCUCAUUGCUCACUCCAGCGUAUAUUACAACCAAAUAAUAUGCUCAGCAUAAUGGUCAACAGCCUCUUGACCAAUAAAUCAAAUUUGGCGUUUGCCUA-ACUAUAAA ..((.((((((-((......)))).)))).(.((((((((........)))))))).).))....(((((((.......))))))).............(((....)))..-........ ( -31.10) >DroYak_CAF1 21711 119 - 1 GAGCAAAAGGG-GCAUCAUUGCUCACUCCAACGUAUAUUACAGCCAAAUAAUAUGCUCAGCAUAAUAGUCAAUAGCCUCUUGACCAAUAAAUCAAAUUUGGCGUUUGCCUAAACUAUAAA ..(((((..((-(((....)))))..................(((((((..(((((...)))))...(((((.......)))))...........))))))).)))))............ ( -25.80) >consensus GAGCAAAAGGG_GCAUCAUUGCUCACUCCAACGUAUAUUACAACCAAAUAAUAUGCUCAGCAUAAUGGUCAAUAGCCUCUUGACCAAUAAAUCAAAUUUGGCGUUUGCCUAAACUAUAAA ....................((..((.((((.((((((((........)))))))).........(((((((.......)))))))...........)))).))..))............ (-17.32 = -17.76 + 0.44)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:28:55 2006