
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 5,153,553 – 5,153,687 |
| Length | 134 |
| Max. P | 0.932641 |

| Location | 5,153,553 – 5,153,662 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.30 |
| Mean single sequence MFE | -27.09 |
| Consensus MFE | -20.93 |
| Energy contribution | -21.09 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.55 |
| Structure conservation index | 0.77 |
| SVM decision value | 1.20 |
| SVM RNA-class probability | 0.929533 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
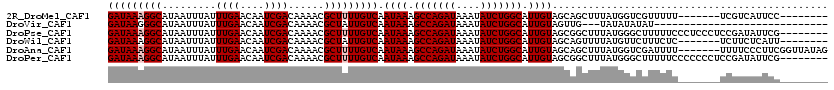
>2R_DroMel_CAF1 5153553 109 + 20766785 GCACG--AG-G-----AUCGGGAACCUA--GACU-UUAUUGAUAAAGGCAUAAUUUAUUUGAACAAUCGACAAAACGCUUUUGUCAAUAAAGCCAGAUAAAUAUCUGGCAUUGUAGCAGC (((((--(.-.-----............--...(-((((((((((((((.........((((....))))......)))))))))))))))(((((((....))))))).)))).))... ( -30.46) >DroVir_CAF1 38639 106 + 1 CCACA--AC-AC--------ACAGCGCA--GACUUUUAUUGAUAAGGGCAUAAUUUAUUUGAACAAUCGACAAAACGCUAUUGUCAAUAAAGCCAGAUAAAUAUCUGGCAUUGUAGUUG- .....--..-..--------.(((((((--(...(((((((((((.(((.........((((....))))......))).)))))))))))(((((((....))))))).)))).))))- ( -27.56) >DroPse_CAF1 30135 113 + 1 GCACA--AG-GCAAUGGACGGGAA-CUA--GACU-UUAUUGAUAAAGGCAUAAUUUAUUUGAACAAUCGACAAAACGCUUUUGUCAAUAAAGCCAGAUAAAUAUCUGGCAUUGUAGCGGC ((...--..-((((((........-...--..((-((((((((((((((.........((((....))))......))))))))))))))))((((((....))))))))))))....)) ( -31.66) >DroWil_CAF1 37952 103 + 1 GUAUG--GG-G-----UAAGGG--------AACU-UUAUUGAUAAAGGCAUAAUUUAUUUGAACAAUCGACAAAACGCUAUUGUCAAUAAAGCCAGAUAAAUAUCUGGCAUUGUAGCAGU .((..--(.-.-----......--------...(-((((((((((.(((.........((((....))))......))).)))))))))))(((((((....))))))).)..))..... ( -23.26) >DroMoj_CAF1 39624 108 + 1 CAGAAACGC-AC--------AGAGCGCA--GACUUUUAUUGAUAAGGGCAUAAUUUAUUUGAACAAUCGACAAAACGCUAUUGUCAAUAAGGCCAGAUAAAUAUCUUGCAUUGUAUAUA- .......((-..--------...))(((--(...(((((((((((.(((.........((((....))))......))).)))))))))))((.((((....)))).)).)))).....- ( -19.06) >DroAna_CAF1 51423 112 + 1 GCAUG--GGAG-----GCCGCCAACCUAGAGACU-UUAUUGAUAAAGGCAUAAUUUAUUUGAACAAUCGACAAAACGCUUUUGUCAAUAAAGCCAGAUAAAUAUCUGGCAUUGUAGCAGC ((..(--(..(-----(...))..)).......(-((((((((((((((.........((((....))))......)))))))))))))))(((((((....)))))))......))... ( -30.56) >consensus GCACA__AG_G_______CGGGAACCUA__GACU_UUAUUGAUAAAGGCAUAAUUUAUUUGAACAAUCGACAAAACGCUAUUGUCAAUAAAGCCAGAUAAAUAUCUGGCAUUGUAGCAGC ...................................((((((((((.(((.........((((....))))......))).)))))))))).(((((((....)))))))........... (-20.93 = -21.09 + 0.17)

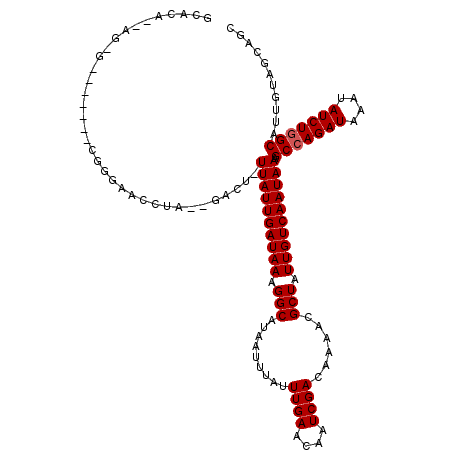
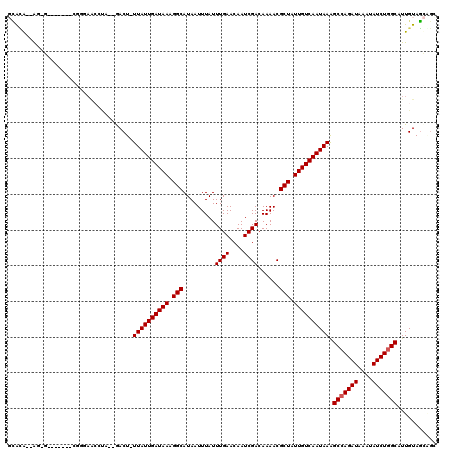
| Location | 5,153,553 – 5,153,662 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.30 |
| Mean single sequence MFE | -24.13 |
| Consensus MFE | -16.95 |
| Energy contribution | -17.12 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.76 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.48 |
| SVM RNA-class probability | 0.752737 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 5153553 109 - 20766785 GCUGCUACAAUGCCAGAUAUUUAUCUGGCUUUAUUGACAAAAGCGUUUUGUCGAUUGUUCAAAUAAAUUAUGCCUUUAUCAAUAA-AGUC--UAGGUUCCCGAU-----C-CU--CGUGC ...((.((...(((((((....)))))))((((((((.(((.(((((((((.((....))..)))))..)))).))).)))))))-)...--.(((........-----)-))--.)))) ( -22.90) >DroVir_CAF1 38639 106 - 1 -CAACUACAAUGCCAGAUAUUUAUCUGGCUUUAUUGACAAUAGCGUUUUGUCGAUUGUUCAAAUAAAUUAUGCCCUUAUCAAUAAAAGUC--UGCGCUGU--------GU-GU--UGUGG -...((((((((((((((....))))))).......(((.(((((((((((.(((.(..((.........))..)..))).)))))....--.)))))))--------))-))--))))) ( -26.00) >DroPse_CAF1 30135 113 - 1 GCCGCUACAAUGCCAGAUAUUUAUCUGGCUUUAUUGACAAAAGCGUUUUGUCGAUUGUUCAAAUAAAUUAUGCCUUUAUCAAUAA-AGUC--UAG-UUCCCGUCCAUUGC-CU--UGUGC ...((.((((.(((((((....)))))((((((((((.(((.(((((((((.((....))..)))))..)))).))).)))))))-))).--...-............))-.)--))))) ( -24.50) >DroWil_CAF1 37952 103 - 1 ACUGCUACAAUGCCAGAUAUUUAUCUGGCUUUAUUGACAAUAGCGUUUUGUCGAUUGUUCAAAUAAAUUAUGCCUUUAUCAAUAA-AGUU--------CCCUUA-----C-CC--CAUAC ...........(((((((....)))))))((((((((.((..(((((((((.((....))..)))))..))))..)).)))))))-)...--------......-----.-..--..... ( -20.30) >DroMoj_CAF1 39624 108 - 1 -UAUAUACAAUGCAAGAUAUUUAUCUGGCCUUAUUGACAAUAGCGUUUUGUCGAUUGUUCAAAUAAAUUAUGCCCUUAUCAAUAAAAGUC--UGCGCUCU--------GU-GCGUUUCUG -..........(((....((((((.((..(..((((((((.......)))))))).)..)).))))))..))).............((..--.((((...--------..-))))..)). ( -20.10) >DroAna_CAF1 51423 112 - 1 GCUGCUACAAUGCCAGAUAUUUAUCUGGCUUUAUUGACAAAAGCGUUUUGUCGAUUGUUCAAAUAAAUUAUGCCUUUAUCAAUAA-AGUCUCUAGGUUGGCGGC-----CUCC--CAUGC (((((((....(((((((....))))(((((((((((.(((.(((((((((.((....))..)))))..)))).))).)))))))-))))....))))))))))-----....--..... ( -31.00) >consensus GCUGCUACAAUGCCAGAUAUUUAUCUGGCUUUAUUGACAAAAGCGUUUUGUCGAUUGUUCAAAUAAAUUAUGCCUUUAUCAAUAA_AGUC__UAGGUUCCCG_______C_CC__CGUGC ...........(((((((....))))))).(((((((.((..(((((((((.((....))..)))))..))))..)).)))))))................................... (-16.95 = -17.12 + 0.17)

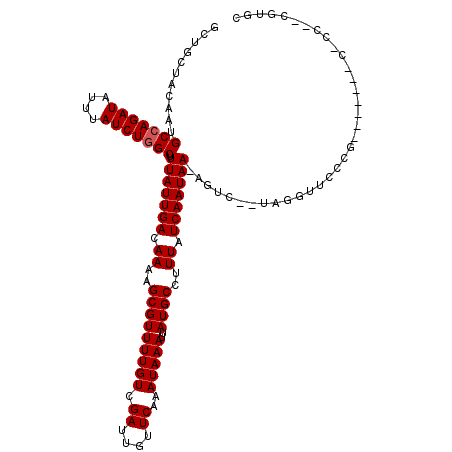
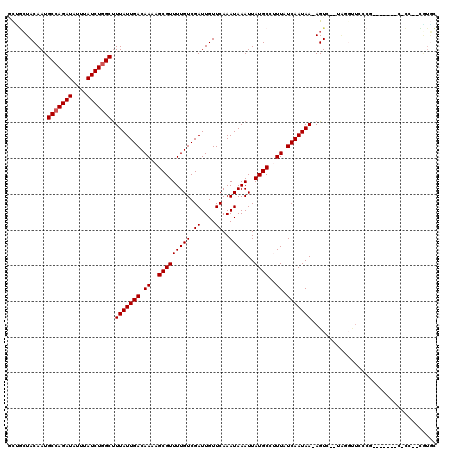
| Location | 5,153,582 – 5,153,687 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.39 |
| Mean single sequence MFE | -24.58 |
| Consensus MFE | -20.23 |
| Energy contribution | -20.56 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.16 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.22 |
| SVM RNA-class probability | 0.932641 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
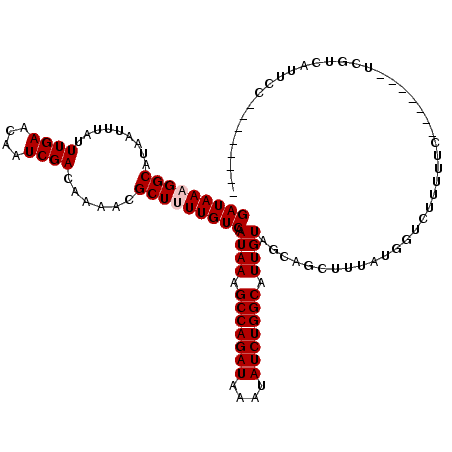
>2R_DroMel_CAF1 5153582 105 + 20766785 GAUAAAGGCAUAAUUUAUUUGAACAAUCGACAAAACGCUUUUGUCAAUAAAGCCAGAUAAAUAUCUGGCAUUGUAGCAGCUUUAUGGUCGUUUUU-------UCGUCAUUCC-------- (((.(((((...................((((((.....)))))).((((.(((((((....))))))).))))....)))))...)))......-------..........-------- ( -24.00) >DroVir_CAF1 38666 88 + 1 GAUAAGGGCAUAAUUUAUUUGAACAAUCGACAAAACGCUAUUGUCAAUAAAGCCAGAUAAAUAUCUGGCAUUGUAGUUG---UAUAUAUAU----------------------------- ......................(((((.(((((.......))))).((((.(((((((....))))))).)))).))))---)........----------------------------- ( -18.40) >DroPse_CAF1 30168 112 + 1 GAUAAAGGCAUAAUUUAUUUGAACAAUCGACAAAACGCUUUUGUCAAUAAAGCCAGAUAAAUAUCUGGCAUUGUAGCGGCUUUAUGGGCUUUUUCCCUCCCUCCGAUAUUCG-------- (((((((((.........((((....))))......))))))))).((((.(((((((....))))))).))))..(((......(((......))).....))).......-------- ( -27.36) >DroWil_CAF1 37975 105 + 1 GAUAAAGGCAUAAUUUAUUUGAACAAUCGACAAAACGCUAUUGUCAAUAAAGCCAGAUAAAUAUCUGGCAUUGUAGCAGUUUUAUGUUCUUUCUC-------UCUUCUCAUU-------- ((.((((((((((.....(((.(((((.(((((.......)))))......(((((((....))))))))))))..)))..))))).))))).))-------..........-------- ( -24.80) >DroAna_CAF1 51455 113 + 1 GAUAAAGGCAUAAUUUAUUUGAACAAUCGACAAAACGCUUUUGUCAAUAAAGCCAGAUAAAUAUCUGGCAUUGUAGCAGCUUUAUGGUCGAUUUU-------UUUUCCCUUCGGUUAUAG (((((((((.........((((....))))......))))))))).((((.(((((((....))))))).))))........((((..(((....-------........)))..)))). ( -26.26) >DroPer_CAF1 29909 112 + 1 GAUAAAGGCAUAAUUUAUUUGAACAAUCGACAAAACGCUUUUGUCAAUAAAGCCAGAUAAAUAUCUGGCAUUGUAGCGGCUUUAUGGGCUUUUUCCCCCCCUCCGAUAUUCG-------- (((((((((.........((((....))))......))))))))).((((.(((((((....))))))).))))..(((......(((......))).....))).......-------- ( -26.66) >consensus GAUAAAGGCAUAAUUUAUUUGAACAAUCGACAAAACGCUUUUGUCAAUAAAGCCAGAUAAAUAUCUGGCAUUGUAGCAGCUUUAUGGUCUUUUUC_______UCGUCAUUCC________ (((((((((.........((((....))))......))))))))).((((.(((((((....))))))).)))).............................................. (-20.23 = -20.56 + 0.33)
| Location | 5,153,582 – 5,153,687 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.39 |
| Mean single sequence MFE | -23.94 |
| Consensus MFE | -18.13 |
| Energy contribution | -18.49 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.91 |
| Structure conservation index | 0.76 |
| SVM decision value | 0.92 |
| SVM RNA-class probability | 0.882751 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
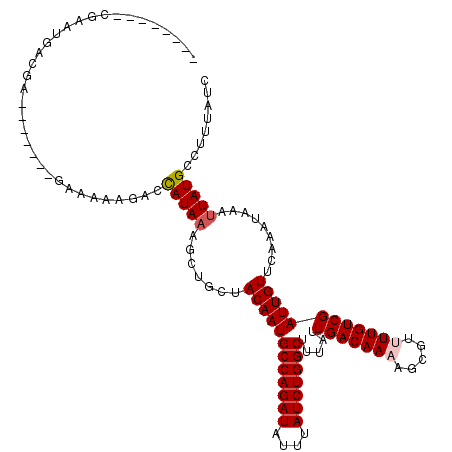
>2R_DroMel_CAF1 5153582 105 - 20766785 --------GGAAUGACGA-------AAAAACGACCAUAAAGCUGCUACAAUGCCAGAUAUUUAUCUGGCUUUAUUGACAAAAGCGUUUUGUCGAUUGUUCAAAUAAAUUAUGCCUUUAUC --------.(((..((((-------.((((((.(.(((((((.((......))(((((....)))))))))))).).......)))))).))).)..))).................... ( -22.30) >DroVir_CAF1 38666 88 - 1 -----------------------------AUAUAUAUA---CAACUACAAUGCCAGAUAUUUAUCUGGCUUUAUUGACAAUAGCGUUUUGUCGAUUGUUCAAAUAAAUUAUGCCCUUAUC -----------------------------.((((....---.....((((((((((((....))))))).....((((((.......)))))))))))..........))))........ ( -17.51) >DroPse_CAF1 30168 112 - 1 --------CGAAUAUCGGAGGGAGGGAAAAAGCCCAUAAAGCCGCUACAAUGCCAGAUAUUUAUCUGGCUUUAUUGACAAAAGCGUUUUGUCGAUUGUUCAAAUAAAUUAUGCCUUUAUC --------.((((((((......(((......)))(((((((.(((.(((((((((((....)))))))...)))).....))))))))))))).))))).................... ( -27.30) >DroWil_CAF1 37975 105 - 1 --------AAUGAGAAGA-------GAGAAAGAACAUAAAACUGCUACAAUGCCAGAUAUUUAUCUGGCUUUAUUGACAAUAGCGUUUUGUCGAUUGUUCAAAUAAAUUAUGCCUUUAUC --------..........-------......(((((...............(((((((....)))))))...((((((((.......))))))))))))).................... ( -22.10) >DroAna_CAF1 51455 113 - 1 CUAUAACCGAAGGGAAAA-------AAAAUCGACCAUAAAGCUGCUACAAUGCCAGAUAUUUAUCUGGCUUUAUUGACAAAAGCGUUUUGUCGAUUGUUCAAAUAAAUUAUGCCUUUAUC ........((((((((..-------..(((((((......(((....(((((((((((....)))))))...)))).....))).....))))))).)))..(((...))).)))))... ( -26.30) >DroPer_CAF1 29909 112 - 1 --------CGAAUAUCGGAGGGGGGGAAAAAGCCCAUAAAGCCGCUACAAUGCCAGAUAUUUAUCUGGCUUUAUUGACAAAAGCGUUUUGUCGAUUGUUCAAAUAAAUUAUGCCUUUAUC --------.(((((.(((.....(((......)))......))).......(((((((....)))))))...(((((((((.....)))))))))))))).................... ( -28.10) >consensus ________CGAAUGACGA_______GAAAAAGACCAUAAAGCUGCUACAAUGCCAGAUAUUUAUCUGGCUUUAUUGACAAAAGCGUUUUGUCGAUUGUUCAAAUAAAUUAUGCCUUUAUC ..................................(((((.......((((((((((((....))))))).....(((((((.....)))))))))))).........)))))........ (-18.13 = -18.49 + 0.36)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:02:29 2006