
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 5,035,726 – 5,035,914 |
| Length | 188 |
| Max. P | 0.991805 |

| Location | 5,035,726 – 5,035,842 |
|---|---|
| Length | 116 |
| Sequences | 3 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 97.70 |
| Mean single sequence MFE | -60.10 |
| Consensus MFE | -59.15 |
| Energy contribution | -58.93 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.02 |
| Mean z-score | -3.32 |
| Structure conservation index | 0.98 |
| SVM decision value | 2.29 |
| SVM RNA-class probability | 0.991805 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 5035726 116 - 20766785 CAGCGCCGCCAGCGGAUGCGUCUGCAGGUGAUUCUGAUGCUGGUGCACAUGGUACUGGUACUGCGGAUGCAUCUGCUGCUGCACCGACGCCUGGAUGUGCGAGUGUCCCAGCGGCG ...((((((((((((((((((((((((..........(((..((((.....))))..)))))))))))))))))))))..((((((.(((......))))).))))....)))))) ( -59.80) >DroEre_CAF1 15790 116 - 1 CAGCGCCGCCAGCGGAUGCGUCUGCAGAUGAUUCUGAUGCUGGUGCACAUGGUACUGGUACUGCGGAUGCAUCUGCUGCUGCACCGACGCCUGGAUGUGCGAGUGUCCCAGCGGCG ...((((((((((((((((((((((((..........(((..((((.....))))..)))))))))))))))))))))..((((((.(((......))))).))))....)))))) ( -59.80) >DroYak_CAF1 15907 116 - 1 CAGCGCCGCCAGCGGAUGUGUCUGCAGAUGGUUCUGGUGCUGGUGCACAUGGUACUGGUACUGCGGAUGCAUCUGCUGCUGCACCGACGCCUGGAUGUGCGAGUGUCCCAGCGGCG ...(((((((((((((((((((((((((....)))(((((..((((.....))))..)))))))))))))))))))))..((((((.(((......))))).))))....)))))) ( -60.70) >consensus CAGCGCCGCCAGCGGAUGCGUCUGCAGAUGAUUCUGAUGCUGGUGCACAUGGUACUGGUACUGCGGAUGCAUCUGCUGCUGCACCGACGCCUGGAUGUGCGAGUGUCCCAGCGGCG ...((((((((((((((((((((((((..........(((..((((.....))))..)))))))))))))))))))))..((((((.(((......))))).))))....)))))) (-59.15 = -58.93 + -0.22)

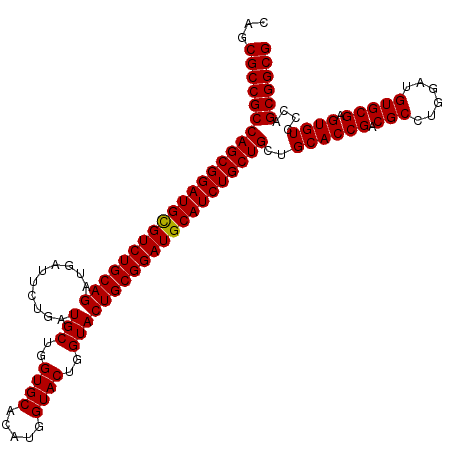
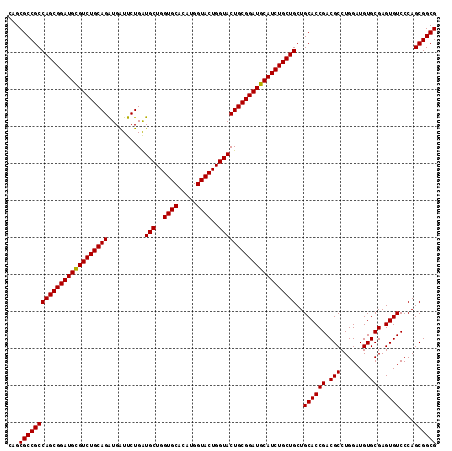
| Location | 5,035,766 – 5,035,880 |
|---|---|
| Length | 114 |
| Sequences | 4 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 87.87 |
| Mean single sequence MFE | -52.25 |
| Consensus MFE | -45.55 |
| Energy contribution | -45.30 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.52 |
| Structure conservation index | 0.87 |
| SVM decision value | 2.03 |
| SVM RNA-class probability | 0.986241 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 5035766 114 - 20766785 CUGGCCAGCGUCCCUCCGAACGGACCGCUCGUGUUGGCCAGCGCCGCCAGCGGAUGCGUCUGCAGGUGAUUCUGAUGCUGGUGCACAUGGUACUGGUACUGCGGAUGCAUCUGC (((((((((((((........))))(....).)))))))))........((((((((((((((((..........(((..((((.....))))..))))))))))))))))))) ( -54.70) >DroEre_CAF1 15830 114 - 1 CUGGCCAGCGUUCCGCCAAACGGACCGCUUGUGUUGGCCAGCGCCGCCAGCGGAUGCGUCUGCAGAUGAUUCUGAUGCUGGUGCACAUGGUACUGGUACUGCGGAUGCAUCUGC ((((((((((.((((.....)))).)))......)))))))........((((((((((((((((..........(((..((((.....))))..))))))))))))))))))) ( -56.30) >DroYak_CAF1 15947 114 - 1 CUGGCCAGCGUUCCUCCGAACGGACCGCUCGUGUUGGCCAGCGCCGCCAGCGGAUGUGUCUGCAGAUGGUUCUGGUGCUGGUGCACAUGGUACUGGUACUGCGGAUGCAUCUGC (((((((((((((....)))).((....))..)))))))))........(((((((((((((((((....)))(((((..((((.....))))..))))))))))))))))))) ( -55.50) >DroMoj_CAF1 23232 114 - 1 CUCGCUAGCGUACCGCCAAAUGGGCCGCUCGUAUUUGCCAACGCUGCCAGCGGAUGGGUUUGCAGGUGAUUUUGGUGCGAAUGCACAUGAUAUUGAUACUGCGGAUUCAUCUGC ...((.(((((...(((.....))).((........))..)))))))..((((((((((((((((.((((....((((....)))).....))))...)))))))))))))))) ( -42.50) >consensus CUGGCCAGCGUUCCGCCAAACGGACCGCUCGUGUUGGCCAGCGCCGCCAGCGGAUGCGUCUGCAGAUGAUUCUGAUGCUGGUGCACAUGGUACUGGUACUGCGGAUGCAUCUGC .((((..((((...(((((((((.....)))).)))))..)))).))))(((((((((((((((((....)))(((((((((((.....))))))))))))))))))))))))) (-45.55 = -45.30 + -0.25)

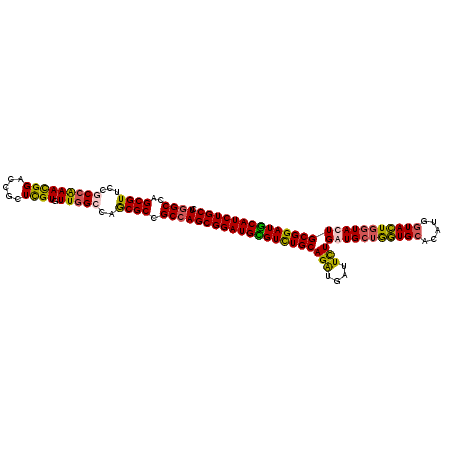
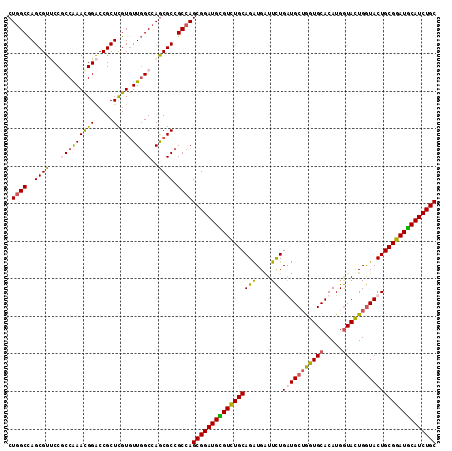
| Location | 5,035,804 – 5,035,914 |
|---|---|
| Length | 110 |
| Sequences | 4 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 81.93 |
| Mean single sequence MFE | -46.30 |
| Consensus MFE | -35.11 |
| Energy contribution | -36.67 |
| Covariance contribution | 1.56 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.05 |
| Structure conservation index | 0.76 |
| SVM decision value | 1.11 |
| SVM RNA-class probability | 0.916106 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 5035804 110 + 20766785 AUCAGAAUCACCUGCAGACGCAUCCGCUGGCGGCGCUGGCCAACACGAGCGGUCCGUUCGGAGGGACGCUGGCCAGCAGUGCCAGGGACUAUGUGGAUG-----GACUGCAUCAU ............(((((.(.((((((((((((..((((((((......(((..((.......))..)))))))))))..)))))........)))))))-----).))))).... ( -46.40) >DroEre_CAF1 15868 110 + 1 AUCAGAAUCAUCUGCAGACGCAUCCGCUGGCGGCGCUGGCCAACACAAGCGGUCCGUUUGGCGGAACGCUGGCCAGCAGUGCCAGGGACUAUGUGGAUG-----GACUGCAUCAU ..(((..(((((..((......(((.((((((..((((((((......(((.((((.....)))).)))))))))))..)))))))))...))..))))-----).)))...... ( -50.90) >DroYak_CAF1 15985 110 + 1 ACCAGAACCAUCUGCAGACACAUCCGCUGGCGGCGCUGGCCAACACGAGCGGUCCGUUCGGAGGAACGCUGGCCAGCAGUGCCAGGGACUAUGUGGAUG-----GACUGCAUCAU ..(((..(((((..((......(((.((((((..((((((((......(((.(((.......))).)))))))))))..)))))))))...))..))))-----).)))...... ( -51.00) >DroMoj_CAF1 23270 112 + 1 ACCAAAAUCACCUGCAAACCCAUCCGCUGGCAGCGUUGGCAAAUACGAGCGGCCCAUUUGGCGGUACGCUAGCGAGCAGUGCUCGAGA---UGUCCUUGGCGCUGGCUACGAUAC ......(((....((.........(((((((.((....))......(.((.(((.....))).)).))))))))..((((((.((((.---....))))))))))))...))).. ( -36.90) >consensus ACCAGAAUCACCUGCAGACGCAUCCGCUGGCGGCGCUGGCCAACACGAGCGGUCCGUUCGGAGGAACGCUGGCCAGCAGUGCCAGGGACUAUGUGGAUG_____GACUGCAUCAU ........((((((((......(((.((((((..(((((((......((((.(((.......))).)))))))))))..)))))))))...))))))))................ (-35.11 = -36.67 + 1.56)

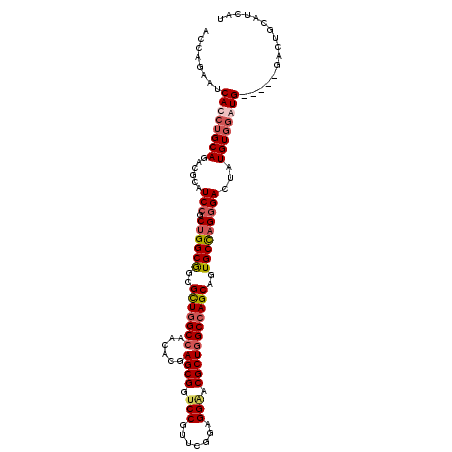

| Location | 5,035,804 – 5,035,914 |
|---|---|
| Length | 110 |
| Sequences | 4 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 81.93 |
| Mean single sequence MFE | -46.00 |
| Consensus MFE | -32.25 |
| Energy contribution | -33.00 |
| Covariance contribution | 0.75 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.75 |
| SVM RNA-class probability | 0.840982 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 5035804 110 - 20766785 AUGAUGCAGUC-----CAUCCACAUAGUCCCUGGCACUGCUGGCCAGCGUCCCUCCGAACGGACCGCUCGUGUUGGCCAGCGCCGCCAGCGGAUGCGUCUGCAGGUGAUUCUGAU ..(((((.((.-----.....))...(((((((((..(((((((((((((((........))))(....).)))))))))))..))))).)))))))))..((((....)))).. ( -47.00) >DroEre_CAF1 15868 110 - 1 AUGAUGCAGUC-----CAUCCACAUAGUCCCUGGCACUGCUGGCCAGCGUUCCGCCAAACGGACCGCUUGUGUUGGCCAGCGCCGCCAGCGGAUGCGUCUGCAGAUGAUUCUGAU ..(((((.((.-----.....))...(((((((((..((((((((((((.((((.....)))).)))......)))))))))..))))).)))))))))..((((....)))).. ( -49.50) >DroYak_CAF1 15985 110 - 1 AUGAUGCAGUC-----CAUCCACAUAGUCCCUGGCACUGCUGGCCAGCGUUCCUCCGAACGGACCGCUCGUGUUGGCCAGCGCCGCCAGCGGAUGUGUCUGCAGAUGGUUCUGGU ......(((.(-----((((.(((((.((((((((..(((((((((((((((....)))).((....))..)))))))))))..))))).)))))))).....)))))..))).. ( -49.50) >DroMoj_CAF1 23270 112 - 1 GUAUCGUAGCCAGCGCCAAGGACA---UCUCGAGCACUGCUCGCUAGCGUACCGCCAAAUGGGCCGCUCGUAUUUGCCAACGCUGCCAGCGGAUGGGUUUGCAGGUGAUUUUGGU ..............(((((((.((---((((((((.(((((.((.(((((...(((.....))).((........))..))))))).)))))....))))).))))).))))))) ( -38.00) >consensus AUGAUGCAGUC_____CAUCCACAUAGUCCCUGGCACUGCUGGCCAGCGUUCCGCCAAACGGACCGCUCGUGUUGGCCAGCGCCGCCAGCGGAUGCGUCUGCAGAUGAUUCUGAU ..((((((...................((((((((..((((((((((((.(((.......))).))))......))))))))..))))).)))))))))..((((....)))).. (-32.25 = -33.00 + 0.75)

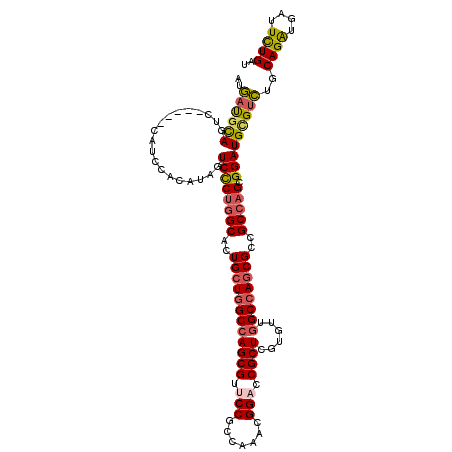
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:01:16 2006