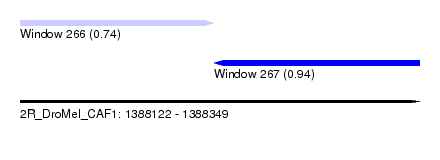
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 1,388,122 – 1,388,349 |
| Length | 227 |
| Max. P | 0.939303 |

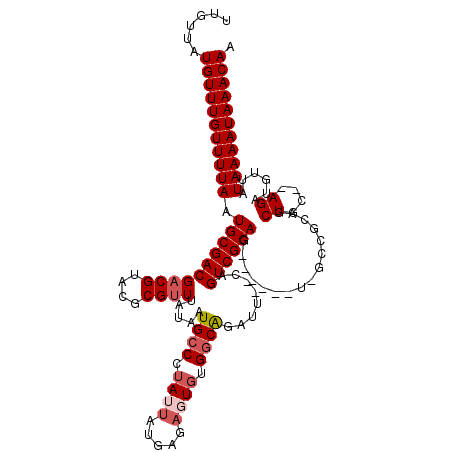
| Location | 1,388,122 – 1,388,232 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 86.49 |
| Mean single sequence MFE | -31.40 |
| Consensus MFE | -21.80 |
| Energy contribution | -22.84 |
| Covariance contribution | 1.04 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.09 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.741684 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1388122 110 + 20766785 UUGUUAUGUUUGUUUUAAUGCGACGACGUACGCGUUUAUAAUGCCCUAUUAUGAGAGUGUGGCAGCUU--CAGUCGCAG------UGGCCGCAGCUGC--AGAUGUUAUAAAAUAAACAA ......(((((((((((.((((((((((....)))).....((((.((((.....)))).))))....--..))))))(------..((....))..)--........))))))))))). ( -33.30) >DroSec_CAF1 15122 103 + 1 UUGUUAUGUUUGUUUUAAUGCGACGACGUACGCGUUUAUAAUGCCCUAUUAUGAGACUGUGGCAGCUU--CAGUCGCAG---------------CUGCAGAGAUGUUAUAAAAUAAACAA ......((((((((((((((((........)))))((((((((...))))))))..(((..((.((..--.....)).)---------------)..)))........))))))))))). ( -26.10) >DroSim_CAF1 19872 103 + 1 UUGUUAUGUUUGUUUUAAUGCGACGACGUACGCGUUUAUAAUGCCCUAUUAUGAGAGUGUGGCAGAUU--CAGUCGCAG---------------CUGCAGAGAUGUUAUAAAAUAAACAA ......(((((((((((.((((((((((....)))).....((((.((((.....)))).))))....--..)))))).---------------..(((....)))..))))))))))). ( -28.10) >DroEre_CAF1 13211 118 + 1 UUGUUAUGUUUGUUUUAAUGCGACGACGUACGCGUUUAUAAUGCCCUAUUAUGAGCUUCAGUCGCAUUCGCAGUCGCAGUCGCAGUCGCCGCAGCUGC--AGAUGUUAUAAAAUAAACAA ......(((((((((((.(((((((((......((((((((((...))))))))))....)))((....)).))))))((((((((.......)))))--.)))....))))))))))). ( -36.90) >DroYak_CAF1 11038 116 + 1 UUGUUAUGUUUGUUUUAAUGCGACGACGUACGCGCUUAUAAUGCCCUAUUAUGAGAGUGUGGCAGAUU--CAGUCGCAGUUACAUUCGCCGCAGCUGC--AGAUGUUAUAAAAUAAACAA ......(((((((((((.((((((((.((((((.(((((((((...))))))))).)))).))....)--).))))))...((((..((.......))--..))))..))))))))))). ( -32.60) >consensus UUGUUAUGUUUGUUUUAAUGCGACGACGUACGCGUUUAUAAUGCCCUAUUAUGAGAGUGUGGCAGAUU__CAGUCGCAG______U_GCCGCAGCUGC__AGAUGUUAUAAAAUAAACAA ......(((((((((((.((((((((((....)))).....((((.((((.....)))).))))........))))))................((....))......))))))))))). (-21.80 = -22.84 + 1.04)

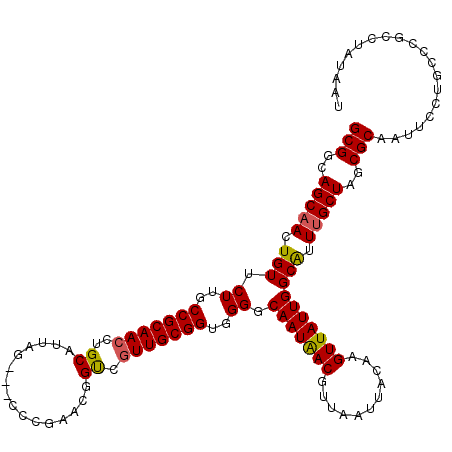
| Location | 1,388,232 – 1,388,349 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.86 |
| Mean single sequence MFE | -43.00 |
| Consensus MFE | -32.59 |
| Energy contribution | -32.43 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.54 |
| Structure conservation index | 0.76 |
| SVM decision value | 1.30 |
| SVM RNA-class probability | 0.939303 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1388232 117 - 20766785 GCGGCAGCAACUGUUCUUGCCGCAACCUGCAUUAG---CCCGAACGGCCGUUGCGGUGGGGCAAUAACGUUAAUUACAAGUUAUUGGCAUUUGCUAGCGCAAUUCCUGCCCGCCUAUAAU ((((((((((.((((((..(((((((..((....)---)((....))..)))))))..)))(((((((...........)))))))))).))))).))(((.....))).)))....... ( -39.90) >DroSec_CAF1 15225 120 - 1 GCGGCAGCAACUGUUCUUGCCGCAACCUGCUUUAGCAACCCCAACGGUCGUUGCGGUGGGGCAAUAACGUUAAUUACAAGUUAUUGGCAUUUGCUAGCGCAAUUGCUGGCCGUCUAUAAU ((((((((((.((((((..(((((((.(((....)))(((.....))).)))))))..)))(((((((...........)))))))))).)))))((((....)))).)))))....... ( -44.30) >DroSim_CAF1 19975 120 - 1 GCGGCAGCAACUGUUCUUGCCGCAACCUGCAUUAGCAGCCCGAACGGUCGUUGCGGUGGGGCAAUGACGUUAAUUACAAGUUAUUGGCAUUUGCUAGCGCAAUUGCUGGCCGUCUAUAAU ((((((((((.((((((..(((((((((((....)))).((....))..)))))))..)))(((((((...........)))))))))).)))))((((....)))).)))))....... ( -46.50) >DroEre_CAF1 13329 116 - 1 GCGGCAGCCACUGUUCUUGCCGCAAGCUGCAUUAG---CCCG-ACGGCCGUUGCGGUGGGCCAAUAACGUUAAUUACAAGUUAUUGGCGUUUGCUGGCGCAAUUACUCCCCGACUAUAAU (((.((((((((((....(((((..((((...)))---)..)-.))))....)))))).(((((((((...........)))))))))....)))).))).................... ( -43.00) >DroYak_CAF1 11154 117 - 1 GCGGCAGCAACUGUUCUUGCCGCAACAUGCAUUAG---CCCGAACGGUCGUUGCGGUGGGGCAAUAACGUUAAUUACAAGUUAUUGGCAUUUGCUAGCGCAAUUCCUCCCCGCCUAUUAU ...(((((.(((((((..((.((.....))....)---)..))))))).)))))((((((((((((((...........)))))))....((((....)))).....)))))))...... ( -41.30) >consensus GCGGCAGCAACUGUUCUUGCCGCAACCUGCAUUAG___CCCGAACGGUCGUUGCGGUGGGGCAAUAACGUUAAUUACAAGUUAUUGGCAUUUGCUAGCGCAAUUCCUGCCCGCCUAUAAU (((..(((((.(((.((..(((((((..((................)).)))))))..)).(((((((...........)))))))))).)))))..))).................... (-32.59 = -32.43 + -0.16)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:28:11 2006