
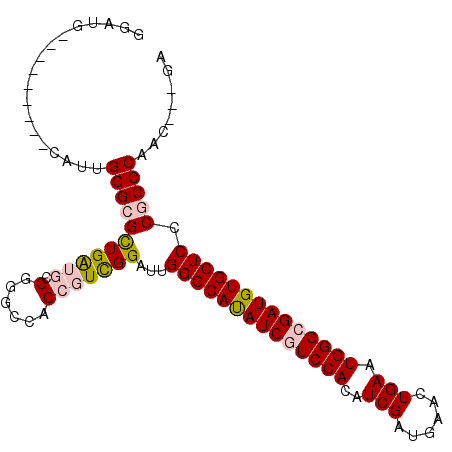
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 4,830,893 – 4,830,985 |
| Length | 92 |
| Max. P | 0.775475 |

| Location | 4,830,893 – 4,830,985 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | forward |
| Mean pairwise identity | 83.80 |
| Mean single sequence MFE | -43.28 |
| Consensus MFE | -32.15 |
| Energy contribution | -32.68 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.09 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.31 |
| SVM RNA-class probability | 0.684704 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
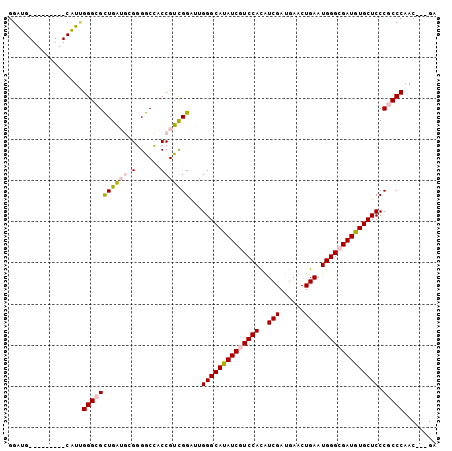
>2R_DroMel_CAF1 4830893 92 + 20766785 GGAUG---------CAUUGGGCGCUGAUGAGGGGCCACCGUUGGAUUGGGCAUAUCGUCCACAUCGAUGAACUGAAUGGGCGAUGUGCUCCCGCCCAAC---GA .....---------.....(((.((.....)).)))..((((((...((((((((((((((..(((......))).))))))))))))))....)))))---). ( -39.10) >DroPse_CAF1 2800 104 + 1 GGGUGGGUGUGCAUCGUCGGGCGUUGGGCGGCGACGACCGUCGGGUUGGGCACAUCGUCCACAUCGAUGAACUGAAUGGGGGAUGUGCUCCCUCCCAACGUCGA .((((......))))....(((((((((.((((((....))))....(((((((((.((((..(((......))).)))).))))))))))).))))))))).. ( -47.80) >DroSim_CAF1 815 92 + 1 GGAUG---------CAUUGGGCGCUGAUGCGGGGCCACCGUCGGAUUGGGCAUAUCGUCCAUGUCGAUGAACUGAAUGGGCGAUGUGCUCCCGCCCAAC---GA .....---------..(((((((((((((.(......)))))))...(((((((((((((((.(((......)))))))))))))))))).))))))).---.. ( -43.00) >DroEre_CAF1 6797 92 + 1 GGAUG---------CAUUGGGCGCUGAUGCGGGGCCACCGUCGGAUUGGGCAUAUCGUCCACGUCGAUGAACUGAAUGGGCGAUGUGCUCCCGCCCAAC---GA .....---------..(((((((((((((.(......)))))))...((((((((((((((..(((......))).)))))))))))))).))))))).---.. ( -41.70) >DroYak_CAF1 2589 92 + 1 GGAUG---------CAUAGGGCGCUGAUGCGGGGCCACCGUCGGAUUGGGCAUAUCGUCCACGUCGAUGAACUGAAUGGGCGAUGUGCUCCCGCCCAAC---GA .....---------....(((((((((((.(......)))))))...((((((((((((((..(((......))).)))))))))))))).)))))...---.. ( -40.30) >DroPer_CAF1 6687 104 + 1 GGGUGGGUGUGCAUCGUCGGGCGUUGGGCGGCGACGACCGUCGGGUUGGGCACAUCGUCCACAUCGAUGAACUGAAUGGGGGAUGUGCUCCCUCCCAACGUCGA .((((......))))....(((((((((.((((((....))))....(((((((((.((((..(((......))).)))).))))))))))).))))))))).. ( -47.80) >consensus GGAUG_________CAUUGGGCGCUGAUGCGGGGCCACCGUCGGAUUGGGCAUAUCGUCCACAUCGAUGAACUGAAUGGGCGAUGUGCUCCCGCCCAAC___GA ..................(((((((((((.(......)))))))...((((((((((((((..(((......))).)))))))))))))).)))))........ (-32.15 = -32.68 + 0.53)

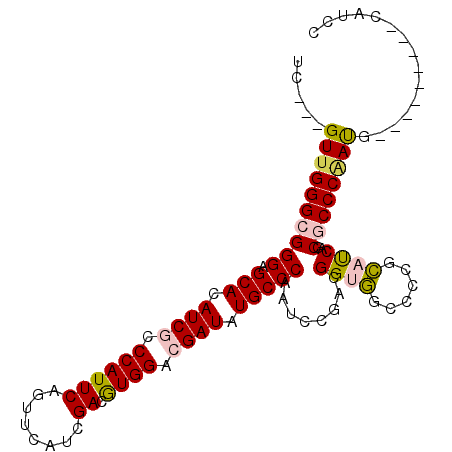
| Location | 4,830,893 – 4,830,985 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | reverse |
| Mean pairwise identity | 83.80 |
| Mean single sequence MFE | -36.37 |
| Consensus MFE | -29.62 |
| Energy contribution | -29.60 |
| Covariance contribution | -0.02 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.96 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.54 |
| SVM RNA-class probability | 0.775475 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
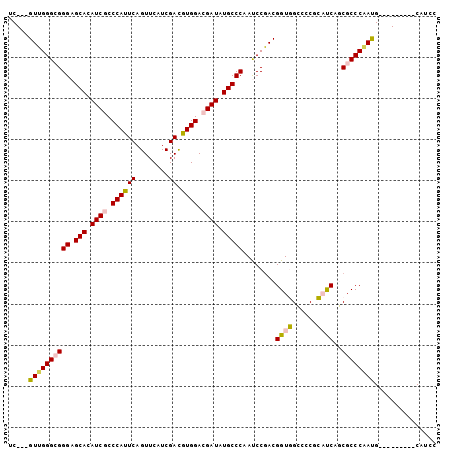
>2R_DroMel_CAF1 4830893 92 - 20766785 UC---GUUGGGCGGGAGCACAUCGCCCAUUCAGUUCAUCGAUGUGGACGAUAUGCCCAAUCCAACGGUGGCCCCUCAUCAGCGCCCAAUG---------CAUCC .(---((((((((((.(((.((((.((((((........)).)))).)))).)))))........(((((....)))))..)))))))))---------..... ( -34.40) >DroPse_CAF1 2800 104 - 1 UCGACGUUGGGAGGGAGCACAUCCCCCAUUCAGUUCAUCGAUGUGGACGAUGUGCCCAACCCGACGGUCGUCGCCGCCCAACGCCCGACGAUGCACACCCACCC ....(((((((..((.(((((((..((((((........)).))))..)))))))))..)))))))(((((((..((.....)).)))))))............ ( -41.80) >DroSim_CAF1 815 92 - 1 UC---GUUGGGCGGGAGCACAUCGCCCAUUCAGUUCAUCGACAUGGACGAUAUGCCCAAUCCGACGGUGGCCCCGCAUCAGCGCCCAAUG---------CAUCC .(---((((((((((.(((.((((.((((((........)).)))).)))).))))).....((..(((....))).))..)))))))))---------..... ( -34.50) >DroEre_CAF1 6797 92 - 1 UC---GUUGGGCGGGAGCACAUCGCCCAUUCAGUUCAUCGACGUGGACGAUAUGCCCAAUCCGACGGUGGCCCCGCAUCAGCGCCCAAUG---------CAUCC .(---((((((((((.(((.((((.((((((........)).)))).)))).))))).....((..(((....))).))..)))))))))---------..... ( -34.50) >DroYak_CAF1 2589 92 - 1 UC---GUUGGGCGGGAGCACAUCGCCCAUUCAGUUCAUCGACGUGGACGAUAUGCCCAAUCCGACGGUGGCCCCGCAUCAGCGCCCUAUG---------CAUCC .(---((((((..((.(((.((((.((((((........)).)))).)))).)))))..)))))))((((...(((....)))..)))).---------..... ( -31.20) >DroPer_CAF1 6687 104 - 1 UCGACGUUGGGAGGGAGCACAUCCCCCAUUCAGUUCAUCGAUGUGGACGAUGUGCCCAACCCGACGGUCGUCGCCGCCCAACGCCCGACGAUGCACACCCACCC ....(((((((..((.(((((((..((((((........)).))))..)))))))))..)))))))(((((((..((.....)).)))))))............ ( -41.80) >consensus UC___GUUGGGCGGGAGCACAUCGCCCAUUCAGUUCAUCGACGUGGACGAUAUGCCCAAUCCGACGGUGGCCCCGCAUCAGCGCCCAAUG_________CAUCC .....((((((((((.(((.((((.((((((........)).)))).)))).)))))........((((......))))..))))))))............... (-29.62 = -29.60 + -0.02)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:59:21 2006