
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 4,807,346 – 4,807,516 |
| Length | 170 |
| Max. P | 0.987161 |

| Location | 4,807,346 – 4,807,439 |
|---|---|
| Length | 93 |
| Sequences | 4 |
| Columns | 93 |
| Reading direction | forward |
| Mean pairwise identity | 75.46 |
| Mean single sequence MFE | -28.35 |
| Consensus MFE | -19.10 |
| Energy contribution | -19.16 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.45 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.52 |
| SVM RNA-class probability | 0.767668 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
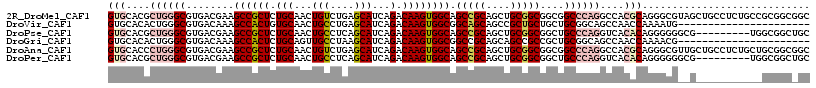
>2R_DroMel_CAF1 4807346 93 + 20766785 AGCAGCAGCACCACCAACAGCACCACCAGCACCACCAUGUGGUCCAACGCCCUGUGCACGCUGGGCGUGACGAAGCCGCUCUGCAACUGUCUG .((((.(((...................(.(((((...))))).).((((((.((....)).)))))).........)))))))......... ( -28.70) >DroVir_CAF1 7302 90 + 1 AAAGGUGAAACG---AGCACUUGCAACACCACCACCAUGUGGUCCAAUGCUCUGUGCACACUGGGCGUGACAAAGCCACUGUGCAACUGCCUG ..((((.....(---((((.(((....(((((......))))).))))))))..((((((...(((........)))..))))))...)))). ( -31.00) >DroGri_CAF1 6719 81 + 1 AAAGGUGAAACA---AGCAGUUGC---------ACCAUGUGGUCCAAUGCUCUGUGCACACUGGGCGUGACAAAGCCACUCUGCAGUUGCCUA ............---.(((..(((---------(....(((((...((((((.((....)).))))))......)))))..))))..)))... ( -24.30) >DroMoj_CAF1 5727 87 + 1 AAAGGUGAAGCG---AGCAUUUGUAACGCC---ACCAUGUGGUCCAAUGCUCUGUGCACACUGGGCGUGACAAAGCCUUUGUGCAACUGUUUG .......(((((---((((((.(....(((---((...)))))).)))))))..((((((..((((........)))).))))))...)))). ( -29.40) >consensus AAAGGUGAAACG___AGCAGUUGCAACACC___ACCAUGUGGUCCAAUGCUCUGUGCACACUGGGCGUGACAAAGCCACUCUGCAACUGCCUG ................(((((((((.............(((((...((((((.((....)).))))))......)))))..)))))))))... (-19.10 = -19.16 + 0.06)


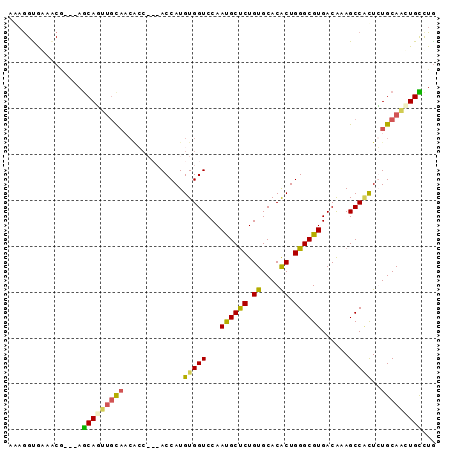
| Location | 4,807,346 – 4,807,439 |
|---|---|
| Length | 93 |
| Sequences | 4 |
| Columns | 93 |
| Reading direction | reverse |
| Mean pairwise identity | 75.46 |
| Mean single sequence MFE | -31.23 |
| Consensus MFE | -20.56 |
| Energy contribution | -20.25 |
| Covariance contribution | -0.31 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.53 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.07 |
| SVM RNA-class probability | 0.569127 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4807346 93 - 20766785 CAGACAGUUGCAGAGCGGCUUCGUCACGCCCAGCGUGCACAGGGCGUUGGACCACAUGGUGGUGCUGGUGGUGCUGUUGGUGGUGCUGCUGCU (((.((((.((..((((((.((..((.(((((((((.(...).)))))))(((((...)))))))))..)).)))))).))...))))))).. ( -36.00) >DroVir_CAF1 7302 90 - 1 CAGGCAGUUGCACAGUGGCUUUGUCACGCCCAGUGUGCACAGAGCAUUGGACCACAUGGUGGUGGUGUUGCAAGUGCU---CGUUUCACCUUU ..((((.((((((((((.(((((((((((...))))).))))))))))).(((((......)))))..))))).))))---............ ( -35.40) >DroGri_CAF1 6719 81 - 1 UAGGCAACUGCAGAGUGGCUUUGUCACGCCCAGUGUGCACAGAGCAUUGGACCACAUGGU---------GCAACUGCU---UGUUUCACCUUU .(((.(((.((((....((((((((((((...))))).))))))).(((.(((....)))---------.))))))).---.)))...))).. ( -28.80) >DroMoj_CAF1 5727 87 - 1 CAAACAGUUGCACAAAGGCUUUGUCACGCCCAGUGUGCACAGAGCAUUGGACCACAUGGU---GGCGUUACAAAUGCU---CGCUUCACCUUU ......((.(((((..(((........)))...))))))).(((((((.((((((...))---))..))...))))))---)........... ( -24.70) >consensus CAGACAGUUGCACAGUGGCUUUGUCACGCCCAGUGUGCACAGAGCAUUGGACCACAUGGU___GGUGUUGCAACUGCU___CGUUUCACCUUU .........((((....((((((((((((...))))).))))))).....(((....))).............))))................ (-20.56 = -20.25 + -0.31)


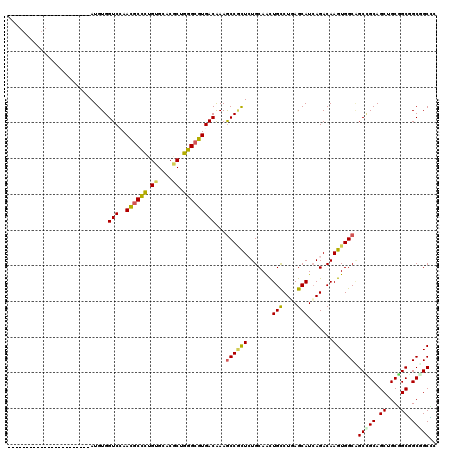
| Location | 4,807,359 – 4,807,479 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 75.56 |
| Mean single sequence MFE | -46.17 |
| Consensus MFE | -31.94 |
| Energy contribution | -31.44 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.29 |
| Structure conservation index | 0.69 |
| SVM decision value | 2.07 |
| SVM RNA-class probability | 0.987161 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4807359 120 + 20766785 CCAACAGCACCACCAGCACCACCAUGUGGUCCAACGCCCUGUGCACGCUGGGCGUGACGAAGCCGCUCUGCAACUGUCUGAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCGGCCC ......((((..((((((((((...))))).....((.....))..)))))..))).)...((((((.(((.((((((((.....))))).))).)))(((((....))))))))))).. ( -52.20) >DroVir_CAF1 7314 118 + 1 --AGCACUUGCAACACCACCACCAUGUGGUCCAAUGCUCUGUGCACACUGGGCGUGACAAAGCCACUGUGCAACUGCCUGAGCAUCAGACAAGUGGCGGCAGCAGCCGCUGCUGCUGCGG --..(((((((...(((((......)))))...((((((..((((((...(((........)))..)))))).......))))))..).))))))((((((((((...)))))))))).. ( -51.50) >DroGri_CAF1 6731 109 + 1 --AGCAGUUGC---------ACCAUGUGGUCCAAUGCUCUGUGCACACUGGGCGUGACAAAGCCACUCUGCAGUUGCCUAAGCAUCAGACAAGUGGCGGCCGCAGCAGCCGCCGCUGCGG --.((....))---------.((.(((((((..((((((.((....)).))))))......(((((((((....(((....))).))))...))))))))))))(((((....))))))) ( -42.40) >DroWil_CAF1 1 97 + 1 -----------------------AUGUGGUCAAACACCUUGUGUACUCUGGGAGUGACAAAACCUUUGUGCAACUGCCUUAGCAUAAGACAAGUGGCCGCUGCAGCAGCAGCUGCGGCAC -----------------------....(((((.....(((((((.....((.(((.((((.....))))...))).))...))))))).....)))))((((((((....)))))))).. ( -37.00) >DroYak_CAF1 1 97 + 1 -----------------------AUGUGGUCCAACGCCCUGUGCACGCUGGGCGUGACGAAGCCGCUCUGCAACUGUCUGAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCGGCCC -----------------------.....(((..((((((.((....)).)))))))))...((((((.(((.((((((((.....))))).))).)))(((((....))))))))))).. ( -49.50) >DroPer_CAF1 1 97 + 1 -----------------------AUGUGGUCGAACGCCCUGUGCACGCUGGGCGUGACGAAGCCGCUCUGCAACUGCCUCAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCUGCCC -----------------------..(..(((..((((((.((....)).))))))......(((((.((((..(((((.(............).)))))..))))..))))))))..).. ( -44.40) >consensus _______________________AUGUGGUCCAACGCCCUGUGCACGCUGGGCGUGACAAAGCCGCUCUGCAACUGCCUGAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCGGCCC ............................(((..((((((.((....)).)))))))))...((((((.......(((....))).......)))))).(((((.((....)).))))).. (-31.94 = -31.44 + -0.50)

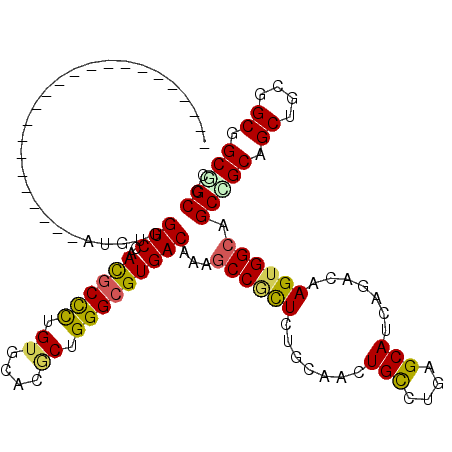
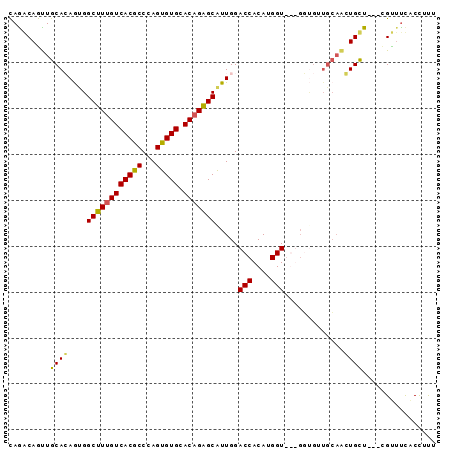
| Location | 4,807,359 – 4,807,479 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 75.56 |
| Mean single sequence MFE | -48.48 |
| Consensus MFE | -31.87 |
| Energy contribution | -30.65 |
| Covariance contribution | -1.22 |
| Combinations/Pair | 1.42 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.66 |
| SVM decision value | 1.84 |
| SVM RNA-class probability | 0.979462 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4807359 120 - 20766785 GGGCCGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUCAGACAGUUGCAGAGCGGCUUCGUCACGCCCAGCGUGCACAGGGCGUUGGACCACAUGGUGGUGCUGGUGGUGCUGUUGG (.(((((((((((....))))).((((((.(((..((((((.((.((((((...))))))))(((((((...))))).)).))))))..))).....))))))...)))))).)...... ( -59.10) >DroVir_CAF1 7314 118 - 1 CCGCAGCAGCAGCGGCUGCUGCCGCCACUUGUCUGAUGCUCAGGCAGUUGCACAGUGGCUUUGUCACGCCCAGUGUGCACAGAGCAUUGGACCACAUGGUGGUGGUGUUGCAAGUGCU-- ..((((((((((...)))))(((((((((.(((..((((((.(.....((((((.((((........).))).))))))).))))))..))).....)))))))))))))).......-- ( -54.70) >DroGri_CAF1 6731 109 - 1 CCGCAGCGGCGGCUGCUGCGGCCGCCACUUGUCUGAUGCUUAGGCAACUGCAGAGUGGCUUUGUCACGCCCAGUGUGCACAGAGCAUUGGACCACAUGGU---------GCAACUGCU-- ..(((.(((((((((...))))))))....(((..((((((.((((((((....(((((...)))))...)))).))).).))))))..))).....).)---------)).......-- ( -48.50) >DroWil_CAF1 1 97 - 1 GUGCCGCAGCUGCUGCUGCAGCGGCCACUUGUCUUAUGCUAAGGCAGUUGCACAAAGGUUUUGUCACUCCCAGAGUACACAAGGUGUUUGACCACAU----------------------- (((..(((((((((...(((..(((.....)))...)))...))))))))).((((.(..((((.((((...))))..))))..).))))..)))..----------------------- ( -33.80) >DroYak_CAF1 1 97 - 1 GGGCCGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUCAGACAGUUGCAGAGCGGCUUCGUCACGCCCAGCGUGCACAGGGCGUUGGACCACAU----------------------- .(((((.(((((.((((((..((((.(((.(((((.....)))))))).)))).)))))).)(((((((...))))).))..)))).))).))....----------------------- ( -48.90) >DroPer_CAF1 1 97 - 1 GGGCAGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUGAGGCAGUUGCAGAGCGGCUUCGUCACGCCCAGCGUGCACAGGGCGUUCGACCACAU----------------------- ((((.((((((((..(((((((((((.(..((.....)).).))))))))))).)))))...(((((((...))))).))..)))))))........----------------------- ( -45.90) >consensus GGGCAGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUCAGGCAGUUGCAGAGCGGCUUCGUCACGCCCAGCGUGCACAGGGCGUUGGACCACAU_______________________ .....((.(((((....))))).)).....(((..((((((.((.((((((...))))))))(((((((...))))).)).))))))..)))............................ (-31.87 = -30.65 + -1.22)

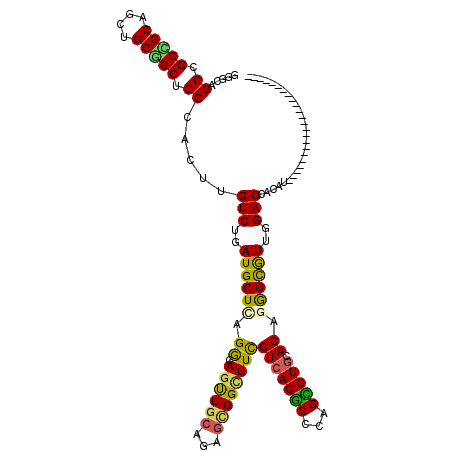
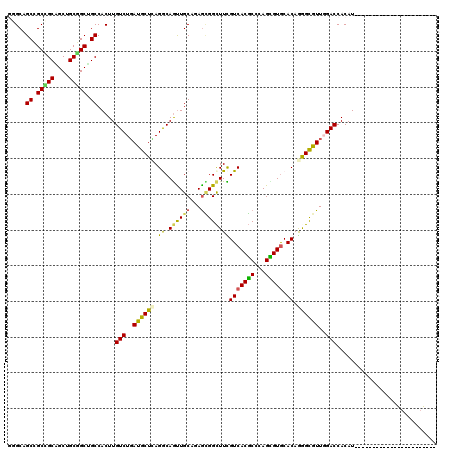
| Location | 4,807,399 – 4,807,516 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 76.18 |
| Mean single sequence MFE | -51.59 |
| Consensus MFE | -28.49 |
| Energy contribution | -29.88 |
| Covariance contribution | 1.39 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.52 |
| SVM RNA-class probability | 0.766398 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4807399 117 + 20766785 GUGCACGCUGGGCGUGACGAAGCCGCUCUGCAACUGUCUGAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCGGCCCAGGCCACGCAGGGCGUAGCUGCCUCUGCCGCGGCGGC ..((.(((((((((.((.(.(((((((((((...((((((.....)))))).(((((.(((((.((....)).)))))....)))))))))))))..))).).)))))).))))))) ( -65.60) >DroVir_CAF1 7352 95 + 1 GUGCACACUGGGCGUGACAAAGCCACUGUGCAACUGCCUGAGCAUCAGACAAGUGGCGGCAGCAGCCGCUGCUGCUGCGGCAGCCAACCAAAAUG---------------------- .((((((...(((........)))..)))))).(((((((.....)).....(..((((((((....))))))))..))))))............---------------------- ( -41.60) >DroPse_CAF1 18 108 + 1 GUGCACGCUGGGCGUGACGAAGCCGCUCUGCAACUGCCUCAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCUGCCCAGGUCACACAGGGGGGCG---------UGGCGGCUGC ((.(((((...)))))))..(((((((.(((..((((....))....(((..(.(((((((((.......))))))))))..)))......))..)))---------.))))))).. ( -52.00) >DroGri_CAF1 6760 95 + 1 GUGCACACUGGGCGUGACAAAGCCACUCUGCAGUUGCCUAAGCAUCAGACAAGUGGCGGCCGCAGCAGCCGCCGCUGCGGCAGCCAACCAAAACG---------------------- ((........(((........(((((((((....(((....))).))))...))))).((((((((.......)))))))).))).......)).---------------------- ( -36.76) >DroAna_CAF1 169 117 + 1 GUGCACCCUGGGCGUGACGAAGCCGCUCUGCAACUGUCUGAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCGGCCCAGGCCACGCAGGGCGUUGCUGCCUCUGCUGCGGCGGC ((.((((....).)))))...(((((.((((.((((((((.....))))).))).)))).((((((.(.(((((((((((..((...))..)).))))))))).).))))))))))) ( -61.60) >DroPer_CAF1 18 108 + 1 GUGCACGCUGGGCGUGACGAAGCCGCUCUGCAACUGCCUCAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCUGCCCAGGUCACACAGGGGGGCG---------UGGCGGCUGC ((.(((((...)))))))..(((((((.(((..((((....))....(((..(.(((((((((.......))))))))))..)))......))..)))---------.))))))).. ( -52.00) >consensus GUGCACGCUGGGCGUGACGAAGCCGCUCUGCAACUGCCUGAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCUGCCCAGGCCACACAGGGCG__G___________GCGGC_GC (((....((((((........((((((.(((...(((....)))...).)))))))).(((((....)))))....))))))....)))............................ (-28.49 = -29.88 + 1.39)
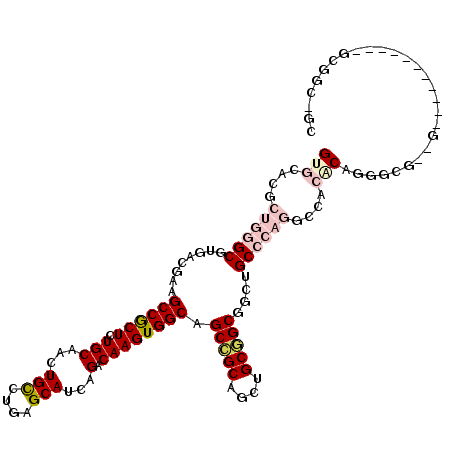
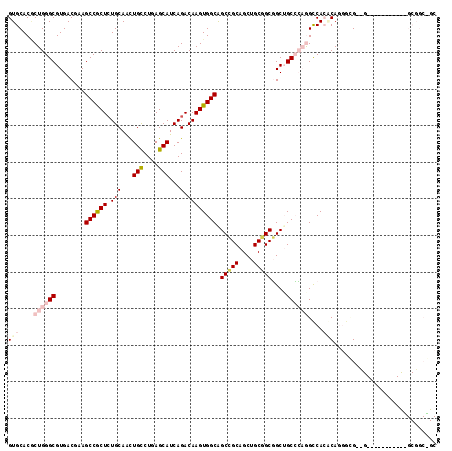
| Location | 4,807,399 – 4,807,516 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 76.18 |
| Mean single sequence MFE | -51.32 |
| Consensus MFE | -30.73 |
| Energy contribution | -31.48 |
| Covariance contribution | 0.76 |
| Combinations/Pair | 1.28 |
| Mean z-score | -1.70 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.72 |
| SVM RNA-class probability | 0.832660 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4807399 117 - 20766785 GCCGCCGCGGCAGAGGCAGCUACGCCCUGCGUGGCCUGGGCCGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUCAGACAGUUGCAGAGCGGCUUCGUCACGCCCAGCGUGCAC (((((.((.((((.(((......)))))))(((((..(((((((((((((....)))))(((.(((.(((((.....)))))))).)))).))))))).)))))))...))).)).. ( -62.20) >DroVir_CAF1 7352 95 - 1 ----------------------CAUUUUGGUUGGCUGCCGCAGCAGCAGCGGCUGCUGCCGCCACUUGUCUGAUGCUCAGGCAGUUGCACAGUGGCUUUGUCACGCCCAGUGUGCAC ----------------------.........((((....((((((((....)))))))).)))).(((((((.....))))))).((((((.((((........).))).)))))). ( -41.00) >DroPse_CAF1 18 108 - 1 GCAGCCGCCA---------CGCCCCCCUGUGUGACCUGGGCAGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUGAGGCAGUUGCAGAGCGGCUUCGUCACGCCCAGCGUGCAC ...(((((((---------(((......)))))...(((((.((.(((((..(((((((((((.(..((.....)).).))))))))))).)))))...))...)))))))).)).. ( -49.50) >DroGri_CAF1 6760 95 - 1 ----------------------CGUUUUGGUUGGCUGCCGCAGCGGCGGCUGCUGCGGCCGCCACUUGUCUGAUGCUUAGGCAACUGCAGAGUGGCUUUGUCACGCCCAGUGUGCAC ----------------------..........((((((.(((((....))))).))))))(((((((....(.(((....))).)....)))))))..((.(((((...))))))). ( -41.60) >DroAna_CAF1 169 117 - 1 GCCGCCGCAGCAGAGGCAGCAACGCCCUGCGUGGCCUGGGCCGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUCAGACAGUUGCAGAGCGGCUUCGUCACGCCCAGGGUGCAC ((.(((........))).))..(((((((((((((..(((((((((((((....)))))(((.(((.(((((.....)))))))).)))).))))))).)))))))..))))))... ( -64.10) >DroPer_CAF1 18 108 - 1 GCAGCCGCCA---------CGCCCCCCUGUGUGACCUGGGCAGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUGAGGCAGUUGCAGAGCGGCUUCGUCACGCCCAGCGUGCAC ...(((((((---------(((......)))))...(((((.((.(((((..(((((((((((.(..((.....)).).))))))))))).)))))...))...)))))))).)).. ( -49.50) >consensus GC_GCCGC___________C__CGCCCUGCGUGGCCUGGGCAGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUCAGGCAGUUGCAGAGCGGCUUCGUCACGCCCAGCGUGCAC ............................(((((((...(((....)))(.((((((..((((.(((.(((((.....)))))))).)))).)))))).))))))))........... (-30.73 = -31.48 + 0.76)
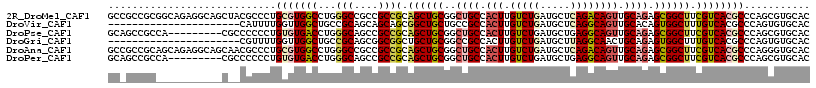
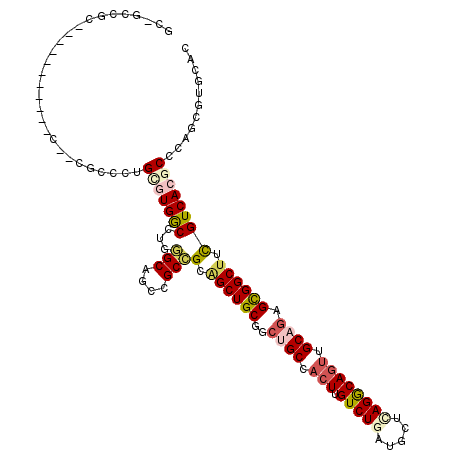

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:59:08 2006