
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 4,532,533 – 4,532,623 |
| Length | 90 |
| Max. P | 0.728421 |

| Location | 4,532,533 – 4,532,623 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | forward |
| Mean pairwise identity | 81.44 |
| Mean single sequence MFE | -20.85 |
| Consensus MFE | -16.84 |
| Energy contribution | -18.20 |
| Covariance contribution | 1.36 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.52 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.42 |
| SVM RNA-class probability | 0.728421 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4532533 90 + 20766785 CGUUAAGCUAUGUG---UAA--UAUAUGGCUUAAUUAUCAUUAGCGUGCUGUGGUAAUUAACAUGCUCAUGCCGGGCAAUUAAACUUAUUU----U----------AUU .(((((((((((((---...--))))))))))(((((((((.((....)))))))))))))).(((((.....))))).............----.----------... ( -21.80) >DroPse_CAF1 6206 100 + 1 CGUUAAGCUAUGUG---UAA--U--AUGGCUUAAUUAUCAUUAGCGUGCUGUGGUAAUUAACAUGCUCAUGCUUGGCAAUUAAACUCUAUAUCUAUAUCUUUCAUU--G .(((((((.(((.(---((.--.--...(.(((((((((((.((....)))))))))))))).))).)))))))))).............................--. ( -21.40) >DroEre_CAF1 6086 100 + 1 CGUUAAGCUAUGUG---UAA--UAUAUGGCUUAAUUAUCAUUAGCGUGCUGUGGUAAUUAACAUGCUCAUGCCGCGCAAUUAAACAUAUUU----UAUUUUUCAUAGUA ......((((((.(---(((--.(((((..(((((((((((.((....)))))))))))))..(((.(.....).)))......))))).)----)))....)))))). ( -22.90) >DroYak_CAF1 6058 95 + 1 CGUUAAACUAUGUGGCUUAAUAUAUAUGGCUUAAUUAUCAUUAGCGUGCUGUGGUAAUAAACAUGCUCAUGCCGGGCAAUUAAACUUAUUU----C----------GUA .(((...(((((((........)))))))....((((((((.((....)))))))))).))).(((((.....))))).............----.----------... ( -16.70) >DroAna_CAF1 7608 96 + 1 CGUUAAGUUAUGUG---UAA--UAUAUGGCUUAAUUAUCAUUAGCGUGCUGUGGUAAUUAACAUGCUCAUGCCGGGCAAUUAAACUUAUAU-AU-UAUUU------UUC .............(---(((--(((((((.(((((((((((.((....)))))))))))))..(((((.....))))).......))))))-))-)))..------... ( -20.90) >DroPer_CAF1 6425 100 + 1 CGUUAAGCUAUGUG---UAA--U--AUGGCUUAAUUAUCAUUAGCGUGCUGUGGUAAUUAACAUGCUCAUGCUUGGCAAUUAAACUCUAUAUCUAUAUCUUUCAUU--G .(((((((.(((.(---((.--.--...(.(((((((((((.((....)))))))))))))).))).)))))))))).............................--. ( -21.40) >consensus CGUUAAGCUAUGUG___UAA__UAUAUGGCUUAAUUAUCAUUAGCGUGCUGUGGUAAUUAACAUGCUCAUGCCGGGCAAUUAAACUUAUUU____UAU_U_______UA .(((((((((((((........))))))))))(((((((((.((....)))))))))))))).(((((.....)))))............................... (-16.84 = -18.20 + 1.36)

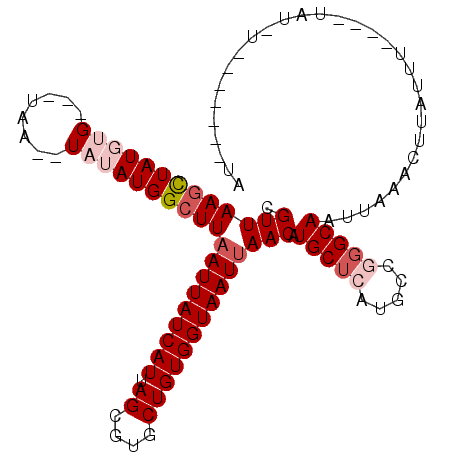
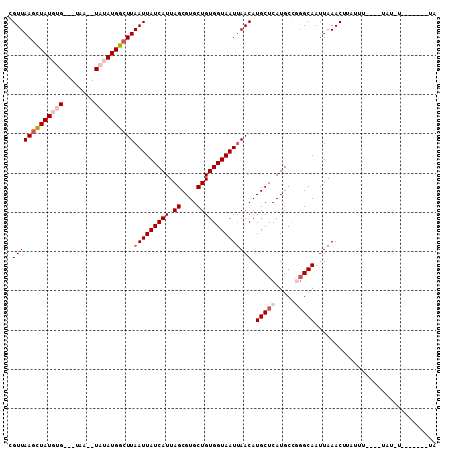
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:57:20 2006