| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 4,523,053 – 4,523,213 |
| Length | 160 |
| Max. P | 0.555049 |

| Location | 4,523,053 – 4,523,173 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 86.67 |
| Mean single sequence MFE | -51.18 |
| Consensus MFE | -39.27 |
| Energy contribution | -39.17 |
| Covariance contribution | -0.11 |
| Combinations/Pair | 1.28 |
| Mean z-score | -2.12 |
| Structure conservation index | 0.77 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.513175 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
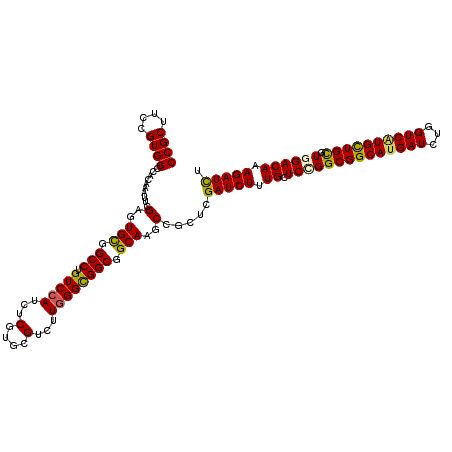
>2R_DroMel_CAF1 4523053 120 - 20766785 UCCUGUGUCUUGGGCGGCGGCAAGCCGCUGGAUCUUUGCUCUGGCGGCAUGAUCUGGUCUUGUUGCGUGGACAAAGAUCUCGACGCCGAGGAGAGUCCGCACGCCGCCCCGGUGAACAAC ...(((..((.(((((((((((((((((..((.......))..)))))..(((...)))...))))((((((.....(((((....)))))...)))))).)))))))).))...))).. ( -55.50) >DroVir_CAF1 70140 120 - 1 UCGUGCGUCUUGGGCGGCGGCAAGCCGCUGGAUCUAUGCUCGGGCGGCAUGAUCUGGUCAUGCUGUGUGGACAAAGAUCUCGAUGCCGAGGAGAGUCCACAUGCGGCGCCGGUCAACAAU (((.(((((...((((((.....))))))((((((.((..((.((((((((((...)))))))))).))..)).)))))).))))))))((...((((....).))).)).......... ( -52.20) >DroGri_CAF1 63680 120 - 1 UCGUGCGUCUUGGGCGGCGGCAAGCCGCUCGAUCUAUGCUCCGGCGGCAUGAUCUGGUCAUGCUGUGUGGACAAAGAUCUCGAUGCCGAGGAGAGUCCGCAUGCGGCACCGAUUAACAAC (((.(((((..(((((((.....)))))))(((((.((.((((((((((((((...)))))))))).)))))).)))))..))))))))...(.(((((....))).))).......... ( -53.60) >DroWil_CAF1 80603 120 - 1 UCAUGUGUCUUAGGUGGCGGCAAACCGCUCGAUCUUUGCUCAGGCGGCAUGAUUUGGUCAUGUUGCGUGGACAAAGAUUUGGAUGCCGAGGAGAACUCACAUGCAACGCCGGUUAAUAAC .........((((.((((((((..(((...((((((((..((.((((((((((...)))))))))).))..))))))))))).))))(((.....))).........)))).)))).... ( -46.40) >DroMoj_CAF1 73890 120 - 1 UCGUGCGUCUUGGGCGGCGGCAAACCUCUCGAUCUCUGCUCCGGCGGCAUGAUCUGGUCAUGCUGUGUGGACAAAGAUCUCGACGCCGAGGAGAGUCCCCAUGCGGCGCCAAUCAACAAU ..(.(((((.((((.(((((....))(((((((((.((.((((((((((((((...)))))))))).)))))).)))))(((....))).)))))))))))...)))))).......... ( -48.30) >DroAna_CAF1 56179 120 - 1 UCAUGUGUCUUGGGCGGCGGCAAGCCCCUGGAUCUUUGCUCCGGCGGCAUGAUCUGGUCGUGUUGCGUGGACAAAGAUCUGGAUGCCGAAGAGAGUCCACACGCUGCUCCAGUCAACAAC ...((((.((.(((((((((((.....(..((((((((.((((((((((((((...)))))))))).))))))))))))..).)))).......((....))))))))).)).).))).. ( -51.10) >consensus UCGUGCGUCUUGGGCGGCGGCAAGCCGCUCGAUCUUUGCUCCGGCGGCAUGAUCUGGUCAUGCUGCGUGGACAAAGAUCUCGAUGCCGAGGAGAGUCCACAUGCGGCGCCGGUCAACAAC .((((((((((.((((((((....))))..(((((.((.((((((((((((((...)))))))))).)))))).)))))....)))).)))).....))))))................. (-39.27 = -39.17 + -0.11)


| Location | 4,523,093 – 4,523,213 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.28 |
| Mean single sequence MFE | -48.53 |
| Consensus MFE | -40.39 |
| Energy contribution | -39.95 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.18 |
| Mean z-score | -1.74 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.04 |
| SVM RNA-class probability | 0.555049 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4523093 120 - 20766785 CCGCUUUCGUGGCCACAAGUUCGAGUGCGGCCUGUCCAUCUCCUGUGUCUUGGGCGGCGGCAAGCCGCUGGAUCUUUGCUCUGGCGGCAUGAUCUGGUCUUGUUGCGUGGACAAAGAUCU ((((.((((.(((.....))))))).))))..((((((((.((((.....)))).)((((((((..((..((((..((((.....)))).))))..)))))))))))))))))....... ( -45.10) >DroVir_CAF1 70180 120 - 1 CCGCUUCCGUGGCCACAAAUUCGAAUGUGGCCUGUCCAUCUCGUGCGUCUUGGGCGGCGGCAAGCCGCUGGAUCUAUGCUCGGGCGGCAUGAUCUGGUCAUGCUGUGUGGACAAAGAUCU ..(((((((((((((((........)))))))((((((...(....)...)))))))))).))))....((((((.((..((.((((((((((...)))))))))).))..)).)))))) ( -54.70) >DroGri_CAF1 63720 120 - 1 CCGCUUCCGUGGACACAAAUUCGAGUGCGGCCUGUCCAUCUCGUGCGUCUUGGGCGGCGGCAAGCCGCUCGAUCUAUGCUCCGGCGGCAUGAUCUGGUCAUGCUGUGUGGACAAAGAUCU .(((....(((((((.....(((....)))..))))))).....)))....(((((((.....)))))))(((((.((.((((((((((((((...)))))))))).)))))).))))). ( -50.60) >DroWil_CAF1 80643 120 - 1 CCGCUUUCGUGGCCACAAAUUCGAAUGCGGCCUGUCCAUCUCAUGUGUCUUAGGUGGCGGCAAACCGCUCGAUCUUUGCUCAGGCGGCAUGAUUUGGUCAUGUUGCGUGGACAAAGAUUU ((((....))))........((((..((((.(((.((((((..........)))))))))....))))))))((((((..((.((((((((((...)))))))))).))..))))))... ( -44.50) >DroMoj_CAF1 73930 120 - 1 CCGCUUCCGUGGCCACAAAUUCGAGUGCGGCCUGUCCAUCUCGUGCGUCUUGGGCGGCGGCAAACCUCUCGAUCUCUGCUCCGGCGGCAUGAUCUGGUCAUGCUGUGUGGACAAAGAUCU ((((....))))..........((((((.(((.(((((...(....)...)))))))).)))...)))..(((((.((.((((((((((((((...)))))))))).)))))).))))). ( -46.80) >DroAna_CAF1 56219 120 - 1 CCGCUUUCGUGGUCACAAAUUCGAGUGCGGCCUGUCCAUUUCAUGUGUCUUGGGCGGCGGCAAGCCCCUGGAUCUUUGCUCCGGCGGCAUGAUCUGGUCGUGUUGCGUGGACAAAGAUCU ((((....))))..........(..(((.(((.(((((...(....)...)))))))).)))..)....(((((((((.((((((((((((((...)))))))))).))))))))))))) ( -49.50) >consensus CCGCUUCCGUGGCCACAAAUUCGAGUGCGGCCUGUCCAUCUCGUGCGUCUUGGGCGGCGGCAAGCCGCUCGAUCUUUGCUCCGGCGGCAUGAUCUGGUCAUGCUGCGUGGACAAAGAUCU ((((....))))..........(..(((.(((.(((((...(....)...)))))))).)))..).....(((((.((.((((((((((((((...)))))))))).)))))).))))). (-40.39 = -39.95 + -0.44)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:57:17 2006