
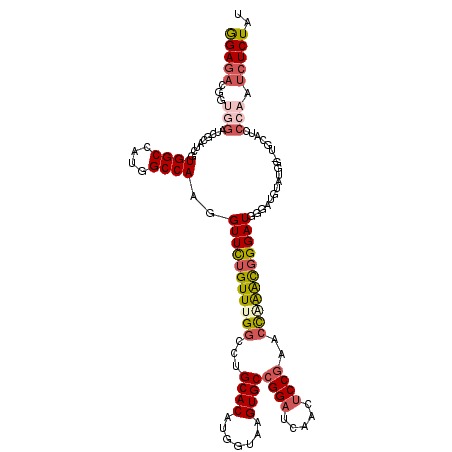
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 4,393,712 – 4,393,830 |
| Length | 118 |
| Max. P | 0.565209 |

| Location | 4,393,712 – 4,393,830 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 82.85 |
| Mean single sequence MFE | -42.77 |
| Consensus MFE | -27.96 |
| Energy contribution | -29.13 |
| Covariance contribution | 1.17 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.74 |
| Structure conservation index | 0.65 |
| SVM decision value | -0.03 |
| SVM RNA-class probability | 0.516441 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4393712 118 + 20766785 GGAGACAGUGGAUCGCAUCGUGGCGAUGGCCAAGGUUCUGUUUGGCCUGCACAUGGUAAGUGCCGGAUCAACUCCGAACCAAACGGGAUGGGAUGUACGGAUGCAUACCAAUCUCUAU (((((...(((((.(((((.((((....))))..(((((((((((...((((.......))))((((.....))))..)))))))))))..........))))))).))).))))).. ( -43.30) >DroSec_CAF1 3717 118 + 1 GGAGACAGUGGAUCGCAUCGUGGCGAUGGCCAAGGUUCUGUUUGGCCUGCACAUGGUAAGUGCCGGAUCAACUCCGAACCGAACGGGAUGGGAUGUAUGGGUGCAUCCCAAUCUCUAU ((((((..((((((((......)))))..)))..)((((((((((...((((.......))))((((.....))))..))))))))))(((((((((....))))))))).))))).. ( -46.90) >DroSim_CAF1 3620 118 + 1 GGAGACGGUGGAUCGCAUCGUGGCGAUGGCCAAGGUUCUGUUUGGCCUGCACAUGGUAAGUGCCGGAUCAACUCCGUACCAAACGGGAUGGGAUGUAUGGGUGCAUCCCAAUCUCUAU (((((.(((.(.((((......))))).)))...(((((((((((...((((.......))))((((.....))))..)))))))))))((((((((....))))))))..))))).. ( -48.70) >DroEre_CAF1 3721 102 + 1 GGAGACGGUGGAUCGCAUCGUGGCCAUGGCCAAGGUUCUGUUUGGCCUGCACAUGGUAAGUGCCGGAUCAACUCCGCCCCCGACGGGAUGGGACGUA----------------UCUAU (....).((((((.((((((((((...(((((((......))))))).)).))))))..((.(((((.....)))..(((....)))..)).)))))----------------))))) ( -35.40) >DroYak_CAF1 3740 117 + 1 GGAGACGGUGGAUCGCAUAGUGGCCAUGGCCAAGGUUCUGUUUGGCCUGCACAUGGUAAGUGCCGGAUCAACUCCGCU-UCGGCGGGAUGGGAUGUAUGAAUGCAACCCAAUCUCUAU (((((.(((.((((((((....((((((((..(((((......))))))).))))))..))))..)))).)))((((.-...))))..((((.((((....)))).)))).))))).. ( -46.20) >DroAna_CAF1 3502 104 + 1 AGAGACGGUGGACCGUAUUGUGGCCAUGGCCAAGGUUUUGUUUGGUCUGCACAUGGUAAGUGCCGGUUAGA-UCCCAUCCAAAU-GGAUGUGAC------------CCGUAUCUCUAU ((((((((.((((((((((...((((((((((((......)))))).....)))))).)))).)))))...-((.(((((....-))))).)))------------)))..))))).. ( -36.10) >consensus GGAGACGGUGGAUCGCAUCGUGGCCAUGGCCAAGGUUCUGUUUGGCCUGCACAUGGUAAGUGCCGGAUCAACUCCGAACCAAACGGGAUGGGAUGUAUGG_UGCAUCCCAAUCUCUAU (((((...(((.........((((....))))..(((((((((((...((((.......))))((((.....))))..)))))))))))..................))).))))).. (-27.96 = -29.13 + 1.17)


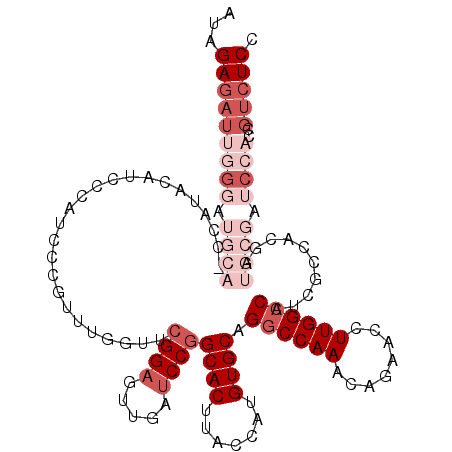
| Location | 4,393,712 – 4,393,830 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 82.85 |
| Mean single sequence MFE | -39.13 |
| Consensus MFE | -21.63 |
| Energy contribution | -25.13 |
| Covariance contribution | 3.50 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.59 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.565209 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4393712 118 - 20766785 AUAGAGAUUGGUAUGCAUCCGUACAUCCCAUCCCGUUUGGUUCGGAGUUGAUCCGGCACUUACCAUGUGCAGGCCAAACAGAACCUUGGCCAUCGCCACGAUGCGAUCCACUGUCUCC ...((((((((..((((((...............((((((((((((.....))))((((.......)))).)))))))).......((((....)))).))))))..)))..))))). ( -40.20) >DroSec_CAF1 3717 118 - 1 AUAGAGAUUGGGAUGCACCCAUACAUCCCAUCCCGUUCGGUUCGGAGUUGAUCCGGCACUUACCAUGUGCAGGCCAAACAGAACCUUGGCCAUCGCCACGAUGCGAUCCACUGUCUCC ...((((((((((((........)))))))........((..((((.....))))((((.......)))).((((((........))))))(((((......)))))))...))))). ( -40.10) >DroSim_CAF1 3620 118 - 1 AUAGAGAUUGGGAUGCACCCAUACAUCCCAUCCCGUUUGGUACGGAGUUGAUCCGGCACUUACCAUGUGCAGGCCAAACAGAACCUUGGCCAUCGCCACGAUGCGAUCCACCGUCUCC ...((((((((((((........)))))))........(((.((((.....))))((((.......)))).((((((........))))))(((((......)))))..)))))))). ( -42.30) >DroEre_CAF1 3721 102 - 1 AUAGA----------------UACGUCCCAUCCCGUCGGGGGCGGAGUUGAUCCGGCACUUACCAUGUGCAGGCCAAACAGAACCUUGGCCAUGGCCACGAUGCGAUCCACCGUCUCC ...((----------------.(((....(((.(((((..((((((.....))).((((.......)))).((((((........))))))...))).))))).)))....))).)). ( -35.40) >DroYak_CAF1 3740 117 - 1 AUAGAGAUUGGGUUGCAUUCAUACAUCCCAUCCCGCCGA-AGCGGAGUUGAUCCGGCACUUACCAUGUGCAGGCCAAACAGAACCUUGGCCAUGGCCACUAUGCGAUCCACCGUCUCC ...(((((((((((((((..............((((...-.))))(((....(((((((.......)))).((((((........)))))).)))..)))))))))))))..))))). ( -41.40) >DroAna_CAF1 3502 104 - 1 AUAGAGAUACGG------------GUCACAUCC-AUUUGGAUGGGA-UCUAACCGGCACUUACCAUGUGCAGACCAAACAAAACCUUGGCCAUGGCCACAAUACGGUCCACCGUCUCU ..((((((..((------------(((.(((((-....))))).))-))).((((((((.......))))................((((....)))).....)))).....)))))) ( -35.40) >consensus AUAGAGAUUGGGAUGCA_CCAUACAUCCCAUCCCGUUUGGUUCGGAGUUGAUCCGGCACUUACCAUGUGCAGGCCAAACAGAACCUUGGCCAUCGCCACGAUGCGAUCCACCGUCUCC ...(((((((((.((((.........................((((.....))))((((.......)))).((((((........))))))..........)))).))))..))))). (-21.63 = -25.13 + 3.50)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:56:15 2006