
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
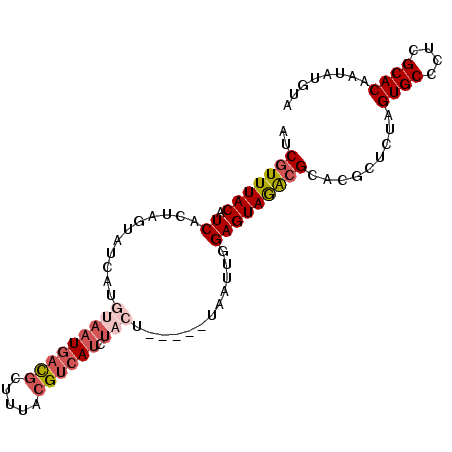
| Location | 4,240,507 – 4,240,599 |
| Length | 92 |
| Max. P | 0.811275 |

| Location | 4,240,507 – 4,240,599 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 97 |
| Reading direction | forward |
| Mean pairwise identity | 82.61 |
| Mean single sequence MFE | -19.48 |
| Consensus MFE | -13.97 |
| Energy contribution | -15.22 |
| Covariance contribution | 1.25 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.21 |
| SVM RNA-class probability | 0.637532 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
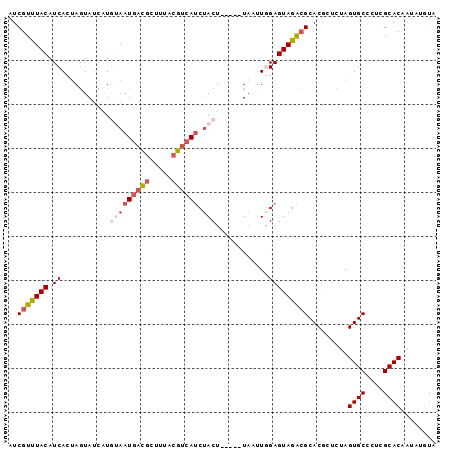
>2R_DroMel_CAF1 4240507 92 + 20766785 AUCGUUUACAUCACUAGUAUCAUGUAAUGACGCUUUACGUCAUCUACU-----UAAAUGGAGUAGACGCACACUCUAGUGCCCUCGCACAAUAUGUA ......(((((((((((.....(((..(((((.....)))))((((((-----(.....))))))).)))....))))))............))))) ( -19.90) >DroSec_CAF1 2055 92 + 1 AUCGUUUACAUCACUAGUAUCAUUGAAUGACGAGUUACGUCAUCUACU-----UAAUUGGAGUAGACGCACGCUCUAGUGCCCUUGCACAAUAUCUA ..(((((((..((..((((.....((.(((((.....)))))))))))-----....))..))))))).........((((....))))........ ( -19.60) >DroSim_CAF1 3560 92 + 1 AUCGUUUACAUCACUAGUAUCAUUGAAUGACGCUUUACGUCAUCUACU-----UAAUUGGAGUAGACGCACGCUCUAGUGCCCUCGCACAAUAUCUA ..(((((((..((..((((.....((.(((((.....)))))))))))-----....))..))))))).........((((....))))........ ( -19.70) >DroEre_CAF1 2070 90 + 1 AUCGUUUACAUCACAAGUAUCAUGUAAUGACGCUUUACGUCAUCUACU-----UAUUUAGAGUAGACGCACGACCUAGUGCCCUCGCACAAU--GUA ..(((((((.((..(((((....(((((((((.....)))))).))).-----))))).))))))))).........((((....))))...--... ( -21.00) >DroYak_CAF1 2051 90 + 1 AUCGUUUACAUCACUAGUAUCAUGUAAUGACGCUUUACGUCAUCUACU-----UAUUUGGAGUAGACGCACGACCUAGUGCCCUCGCACAAU--GUA ..(((((((.((...((((....(((((((((.....)))))).))).-----))))..))))))))).........((((....))))...--... ( -21.90) >DroAna_CAF1 2049 90 + 1 AUCGUUUACAUCAUUAUUAGAUUGUAAUAAUG------GCCAACCACUAUCAAUUCUUAGAGUAAGAGCACACUCUAGUGCCCUCGCACC-UACGUA ......(((.(((((((((.....))))))))------).......(((........))).(..((.((((......)))).))..)...-...))) ( -14.80) >consensus AUCGUUUACAUCACUAGUAUCAUGUAAUGACGCUUUACGUCAUCUACU_____UAAUUGGAGUAGACGCACGCUCUAGUGCCCUCGCACAAUAUGUA ..(((((((.((...........(((((((((.....)))))).)))............))))))))).........((((....))))........ (-13.97 = -15.22 + 1.25)

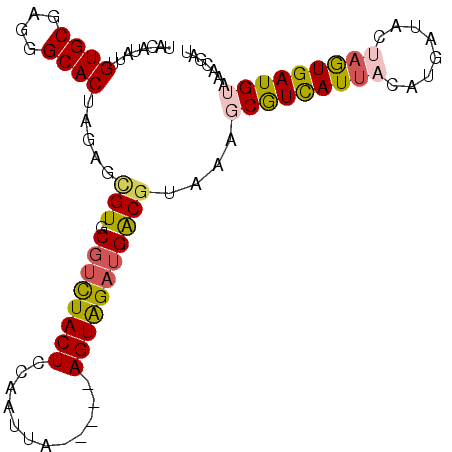
| Location | 4,240,507 – 4,240,599 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 97 |
| Reading direction | reverse |
| Mean pairwise identity | 82.61 |
| Mean single sequence MFE | -26.33 |
| Consensus MFE | -18.39 |
| Energy contribution | -18.78 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.46 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.65 |
| SVM RNA-class probability | 0.811275 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
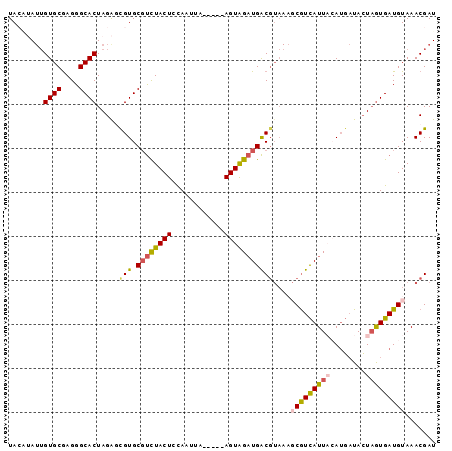
>2R_DroMel_CAF1 4240507 92 - 20766785 UACAUAUUGUGCGAGGGCACUAGAGUGUGCGUCUACUCCAUUUA-----AGUAGAUGACGUAAAGCGUCAUUACAUGAUACUAGUGAUGUAAACGAU (((((...((((....))))....)))))((((((((.......-----)))))))).(((...(((((((((........)))))))))..))).. ( -26.40) >DroSec_CAF1 2055 92 - 1 UAGAUAUUGUGCAAGGGCACUAGAGCGUGCGUCUACUCCAAUUA-----AGUAGAUGACGUAACUCGUCAUUCAAUGAUACUAGUGAUGUAAACGAU .....(((((.......((((((.((((.((((((((.......-----)))))))))))).....(((((...))))).))))))......))))) ( -25.42) >DroSim_CAF1 3560 92 - 1 UAGAUAUUGUGCGAGGGCACUAGAGCGUGCGUCUACUCCAAUUA-----AGUAGAUGACGUAAAGCGUCAUUCAAUGAUACUAGUGAUGUAAACGAU .....(((((.......((((((.((((.((((((((.......-----)))))))))))).....(((((...))))).))))))......))))) ( -25.42) >DroEre_CAF1 2070 90 - 1 UAC--AUUGUGCGAGGGCACUAGGUCGUGCGUCUACUCUAAAUA-----AGUAGAUGACGUAAAGCGUCAUUACAUGAUACUUGUGAUGUAAACGAU (((--(((((((....))))...((((((.(((((((.......-----)))))))((((.....))))....))))))......))))))...... ( -26.20) >DroYak_CAF1 2051 90 - 1 UAC--AUUGUGCGAGGGCACUAGGUCGUGCGUCUACUCCAAAUA-----AGUAGAUGACGUAAAGCGUCAUUACAUGAUACUAGUGAUGUAAACGAU ...--(((((.......((((((((((((.(((((((.......-----)))))))((((.....))))....)))))).))))))......))))) ( -28.12) >DroAna_CAF1 2049 90 - 1 UACGUA-GGUGCGAGGGCACUAGAGUGUGCUCUUACUCUAAGAAUUGAUAGUGGUUGGC------CAUUAUUACAAUCUAAUAAUGAUGUAAACGAU (((((.-((.(..(((((((......)))))))..).)).(((..(((((((((....)------))))))))...))).......)))))...... ( -26.40) >consensus UACAUAUUGUGCGAGGGCACUAGAGCGUGCGUCUACUCCAAUUA_____AGUAGAUGACGUAAAGCGUCAUUACAUGAUACUAGUGAUGUAAACGAU ........((((....)))).....(((.((((((((............)))))))))))....(((((((((........)))))))))....... (-18.39 = -18.78 + 0.39)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:54:45 2006