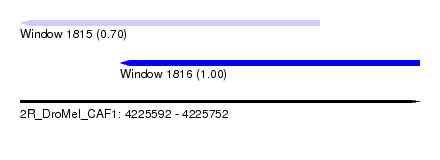
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 4,225,592 – 4,225,752 |
| Length | 160 |
| Max. P | 0.997791 |

| Location | 4,225,592 – 4,225,712 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.67 |
| Mean single sequence MFE | -53.28 |
| Consensus MFE | -50.42 |
| Energy contribution | -50.90 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.19 |
| Structure conservation index | 0.95 |
| SVM decision value | 0.36 |
| SVM RNA-class probability | 0.704941 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4225592 120 - 20766785 GCAGCUGCUGGGCGGCGGAGAUCAUGUUCGGCUCCAGGGUGUCCCAGUUGUAGUUGCCCAGGAAGCUGAUGCCGAUGCUGUAGGGGUUCCACUCGGCGGCGUGGGCGCCCAUGUUGUUCC .(((((.(((((((((((((.((......)))))).(((...))).......)))))))))..)))))..(((.(((((((.(((......))).))))))).))).............. ( -55.30) >DroSec_CAF1 37463 120 - 1 GCAGCUGCUGGGCGGCGGAGAUCAUGUUCGGCUCCAAGGUGUUCCAGUUGUAGUUGCCCAGGAAGCUAAUGCCGAUGCUGUAGGGGUUCCACUCGGCGGCGUGGGCACCCAUGUUGUUCC ..((((.(((((((((((((.((......))))))..((....)).......)))))))))..))))..((((.(((((((.(((......))).))))))).))))............. ( -51.40) >DroSim_CAF1 66853 120 - 1 GCAGCUGCUGGGCGGCGGAGAUCAUGUUCGGCUCCAGGGUGUCCCAGUUGUAGUUGCCCAGGAAGCUGAUGCCGAUGCUGUAGGGGUUCCACUCGGCGGCGUGGGCGCCCAUGUUGUUCC .(((((.(((((((((((((.((......)))))).(((...))).......)))))))))..)))))..(((.(((((((.(((......))).))))))).))).............. ( -55.30) >DroEre_CAF1 38976 120 - 1 GCAGCUGCUGGGCGGCGGAGAUCAUGUUUGGCUCCAGGGUGUCCCAGUUGAAGUUGCCCAGGAAGCUGAUGCCGAUGCUGUAGGCAUUCCAUUCGGCGGCGUGGGCGCCCAUGUUGUUCC .(((((.(((((((((((((.(((....))))))).(((...))).......)))))))))..)))))..((((((((.....)))......)))))(((((((....)))))))..... ( -54.20) >DroYak_CAF1 40104 120 - 1 GCAGCUGCUGUGCGGCGGAGAUCAUGUUCGGCUCCAGGGUGUCCCAGUUGUAGUUGCCCAGGAAGCUGAUGCCGAUGCUGUAGGGGUUCCACUCGGCGGCGUGGGCGCCCAUGUUGUUCC ...(((((...)))))((((..((((.((((((((.(((((.............))))).)).)))))).(((.(((((((.(((......))).))))))).)))...))))...)))) ( -50.22) >consensus GCAGCUGCUGGGCGGCGGAGAUCAUGUUCGGCUCCAGGGUGUCCCAGUUGUAGUUGCCCAGGAAGCUGAUGCCGAUGCUGUAGGGGUUCCACUCGGCGGCGUGGGCGCCCAUGUUGUUCC .(((((.(((((((((((((.((......)))))).(((...))).......)))))))))..))))).((((.(((((((.(((......))).))))))).))))............. (-50.42 = -50.90 + 0.48)

| Location | 4,225,632 – 4,225,752 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.67 |
| Mean single sequence MFE | -55.22 |
| Consensus MFE | -53.38 |
| Energy contribution | -53.82 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.02 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.97 |
| SVM decision value | 2.93 |
| SVM RNA-class probability | 0.997791 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4225632 120 - 20766785 UAGCCGGAGCUGAGCUGGCCACGGUUGACGGCGUCGUUGAGCAGCUGCUGGGCGGCGGAGAUCAUGUUCGGCUCCAGGGUGUCCCAGUUGUAGUUGCCCAGGAAGCUGAUGCCGAUGCUG ((((((..(((.....)))..)))))).((((((((.....(((((.(((((((((((((.((......)))))).(((...))).......)))))))))..)))))....)))))))) ( -57.50) >DroSec_CAF1 37503 120 - 1 UAGCCGGAGCUGAGCUGGCCACGGUUGACGGCGUCGUUGAGCAGCUGCUGGGCGGCGGAGAUCAUGUUCGGCUCCAAGGUGUUCCAGUUGUAGUUGCCCAGGAAGCUAAUGCCGAUGCUG ((((((..(((.....)))..)))))).((((((((......((((.(((((((((((((.((......))))))..((....)).......)))))))))..)))).....)))))))) ( -51.70) >DroSim_CAF1 66893 120 - 1 UAGCCGGAGCUGAGCUGGCCACGGUUGACGGCGUCGUUGAGCAGCUGCUGGGCGGCGGAGAUCAUGUUCGGCUCCAGGGUGUCCCAGUUGUAGUUGCCCAGGAAGCUGAUGCCGAUGCUG ((((((..(((.....)))..)))))).((((((((.....(((((.(((((((((((((.((......)))))).(((...))).......)))))))))..)))))....)))))))) ( -57.50) >DroEre_CAF1 39016 120 - 1 UAGCCGGAGCUGAGUUGGCCACGGUUGACGGCGUCGUUGAGCAGCUGCUGGGCGGCGGAGAUCAUGUUUGGCUCCAGGGUGUCCCAGUUGAAGUUGCCCAGGAAGCUGAUGCCGAUGCUG ((((((..(((.....)))..)))))).((((((((.....(((((.(((((((((((((.(((....))))))).(((...))).......)))))))))..)))))....)))))))) ( -59.00) >DroYak_CAF1 40144 120 - 1 UAGCCGGAGCUGAGCUGGCCACGGUUGACGGCGUCGUUGAGCAGCUGCUGUGCGGCGGAGAUCAUGUUCGGCUCCAGGGUGUCCCAGUUGUAGUUGCCCAGGAAGCUGAUGCCGAUGCUG ((((((..(((.....)))..)))))).((((((((.....(((((.(((.(((((((((.((......)))))).(((...))).......))))).)))..)))))....)))))))) ( -50.40) >consensus UAGCCGGAGCUGAGCUGGCCACGGUUGACGGCGUCGUUGAGCAGCUGCUGGGCGGCGGAGAUCAUGUUCGGCUCCAGGGUGUCCCAGUUGUAGUUGCCCAGGAAGCUGAUGCCGAUGCUG ((((((..(((.....)))..)))))).((((((((.....(((((.(((((((((((((.((......)))))).(((...))).......)))))))))..)))))....)))))))) (-53.38 = -53.82 + 0.44)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:54:30 2006