
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
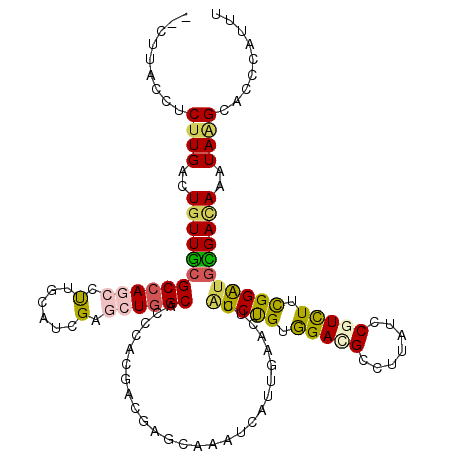
| Location | 870,010 – 870,128 |
| Length | 118 |
| Max. P | 0.849891 |

| Location | 870,010 – 870,128 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.89 |
| Mean single sequence MFE | -36.30 |
| Consensus MFE | -20.78 |
| Energy contribution | -20.65 |
| Covariance contribution | -0.13 |
| Combinations/Pair | 1.44 |
| Mean z-score | -2.09 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.54 |
| SVM RNA-class probability | 0.773689 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
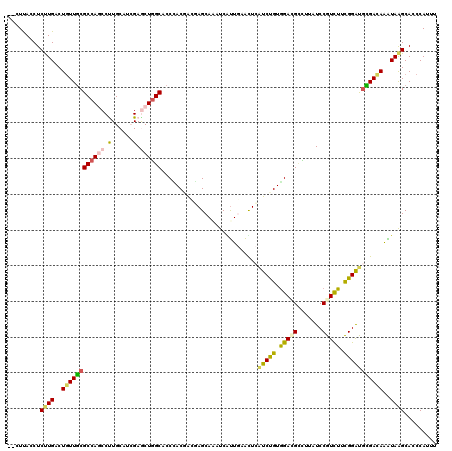
>2R_DroMel_CAF1 870010 118 + 20766785 --UUUACCUCUUGGCUGUUCCGCCAUCCUUGCAUAGAGCUAGCUCCAACGGCUAGCAAUUCAUUAAACUGAUCUGUGGAUGUUUUAUCCGUUUUCGGAUGGGACAAAUAGGCACCCAUAU --....((....)).......(((.......(((((((((((((.....)))))))...(((......)))))))))..(((((((((((....)))))))))))....)))........ ( -36.90) >DroVir_CAF1 7413 118 + 1 --CUUACCUCUUGACUGUUGCGCCAGCCCUGCAUCGAACUGGCACCCACGACGAGCAUAUCAUUGAAUUCAUCUGUGGAAGCUUUUUCCGUCUUCGGAUGCGACAAGUAAGCACCCAUUU --.......((((..(((((((((((..(......)..)))))..((((((.((.((......))...)).)).))))........((((....)))).))))))..))))......... ( -29.30) >DroPse_CAF1 2200 118 + 1 --CUUACCUCUUGACUGUUGCGCCAGCCCUGCAUCGAGCUGGCACCUACUACCAGCAAAUCGUUCAAUUCAUCCGUAGACGAUUUUUCCGUCUUCGGAUGUGACAAAUAAGCACCCAUUU --.......((((..(((..(((((((.(......).))))))...........................(((((.(((((.......))))).))))))..)))..))))......... ( -32.50) >DroGri_CAF1 10060 118 + 1 --AUUACCUCUUGACUGUUGCGCCAGCCUUGCAUGGAACUGGCUCCCACAAUGAGCAGCUCGCUGCGCUCGUCUGUGGAGGCCUUUUCCGUCUUUGGAUGCGAUAGGUAGGCACCCAUUG --.......((((.((((((((((((....(.((((((..(((..(((((((((((.((.....)))))))).)))))..)))..))))))).)))).)))))))).))))......... ( -47.80) >DroWil_CAF1 16744 120 + 1 UGCUCACCUCCUGGCUAUUGCGCCAACCUUGCAUCGAGCUGGCCCCUACCACAAGCAAGUCGUUGAACUCAUCUGUGGACGCCUUAUCCGUCUUCGGGCUCGACAAAUAAGCACCCAUGU ((((.......((((......))))........((((((.(((.....(((((....(((......)))....)))))..)))....(((....)))))))))......))))....... ( -30.00) >DroMoj_CAF1 6758 118 + 1 --CUCACCUCUUGGCUGUUGCGCCAGCCUUGCAUCGAGCUGGCACCCACGACGAGCAUCUCAUUGAAUUCGUCUGUGGACGCCUUAUCCGUCUUUGGAUGCGAUAGGUAAGCACCCAUUU --.......((((.(((((((((((((.(((...)))))))))..((((((((((..((.....)).)))))).)))).......(((((....)))))))))))).))))......... ( -41.30) >consensus __CUUACCUCUUGACUGUUGCGCCAGCCUUGCAUCGAGCUGGCACCCACGACGAGCAAAUCAUUGAACUCAUCUGUGGACGCCUUAUCCGUCUUCGGAUGCGACAAAUAAGCACCCAUUU .........((((..((((((((((((.(......).))))))...........................(((((.(((((.......))))).)))))))))))..))))......... (-20.78 = -20.65 + -0.13)

| Location | 870,010 – 870,128 |
|---|---|
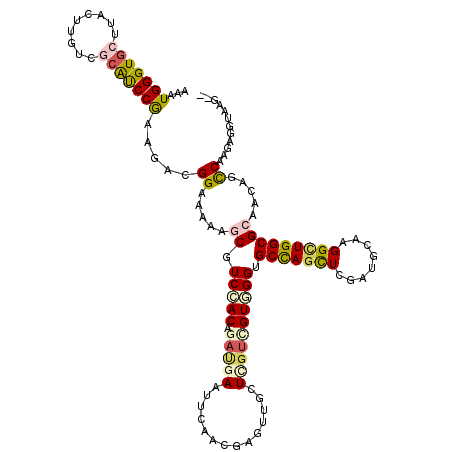
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.89 |
| Mean single sequence MFE | -42.02 |
| Consensus MFE | -22.28 |
| Energy contribution | -22.96 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.39 |
| Mean z-score | -2.46 |
| Structure conservation index | 0.53 |
| SVM decision value | 0.78 |
| SVM RNA-class probability | 0.849891 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
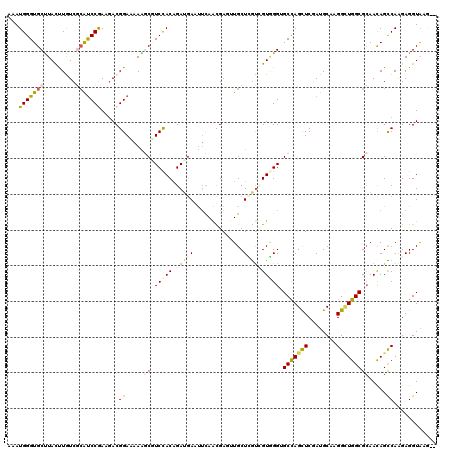
>2R_DroMel_CAF1 870010 118 - 20766785 AUAUGGGUGCCUAUUUGUCCCAUCCGAAAACGGAUAAAACAUCCACAGAUCAGUUUAAUGAAUUGCUAGCCGUUGGAGCUAGCUCUAUGCAAGGAUGGCGGAACAGCCAAGAGGUAAA-- .......(((((..((((.(((((((....)))))....(((((.....(((......)))...(((((((....).)))))).........)))))..)).)))).....)))))..-- ( -32.90) >DroVir_CAF1 7413 118 - 1 AAAUGGGUGCUUACUUGUCGCAUCCGAAGACGGAAAAAGCUUCCACAGAUGAAUUCAAUGAUAUGCUCGUCGUGGGUGCCAGUUCGAUGCAGGGCUGGCGCAACAGUCAAGAGGUAAG-- ...(((((((.........)))))))..(((...........((((.(((((..............)))))))))((((((((((......))))))))))....)))..........-- ( -39.14) >DroPse_CAF1 2200 118 - 1 AAAUGGGUGCUUAUUUGUCACAUCCGAAGACGGAAAAAUCGUCUACGGAUGAAUUGAACGAUUUGCUGGUAGUAGGUGCCAGCUCGAUGCAGGGCUGGCGCAACAGUCAAGAGGUAAG-- .......(((((..((((((((((((.((((((.....)))))).))))))...)).))))(((((((.......((((((((((......))))))))))..))).)))))))))..-- ( -43.80) >DroGri_CAF1 10060 118 - 1 CAAUGGGUGCCUACCUAUCGCAUCCAAAGACGGAAAAGGCCUCCACAGACGAGCGCAGCGAGCUGCUCAUUGUGGGAGCCAGUUCCAUGCAAGGCUGGCGCAACAGUCAAGAGGUAAU-- ...(((((((.........))))))).....((((..(((..((((((..((((((.....)).)))).))))))..)))..))))......(((((......)))))..........-- ( -45.10) >DroWil_CAF1 16744 120 - 1 ACAUGGGUGCUUAUUUGUCGAGCCCGAAGACGGAUAAGGCGUCCACAGAUGAGUUCAACGACUUGCUUGUGGUAGGGGCCAGCUCGAUGCAAGGUUGGCGCAAUAGCCAGGAGGUGAGCA .......(((((((((((((((((((....)))....(((.(((((((..(((((....)))))..))))))...).))).)))))))......(((((......))))).))))))))) ( -44.80) >DroMoj_CAF1 6758 118 - 1 AAAUGGGUGCUUACCUAUCGCAUCCAAAGACGGAUAAGGCGUCCACAGACGAAUUCAAUGAGAUGCUCGUCGUGGGUGCCAGCUCGAUGCAAGGCUGGCGCAACAGCCAAGAGGUGAG-- .........(((((((.....((((......))))..(((..((((.(((((..((.....))...)))))))))(((((((((........)))))))))....)))...)))))))-- ( -46.40) >consensus AAAUGGGUGCUUACUUGUCGCAUCCGAAGACGGAAAAAGCGUCCACAGAUGAAUUCAACGAGUUGCUCGUCGUGGGUGCCAGCUCGAUGCAAGGCUGGCGCAACAGCCAAGAGGUAAG__ ...(((((((.........))))))).....((.....((.(((((.(((((..............)))))))))).(((((((........))))))))).....))............ (-22.28 = -22.96 + 0.67)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:23:20 2006