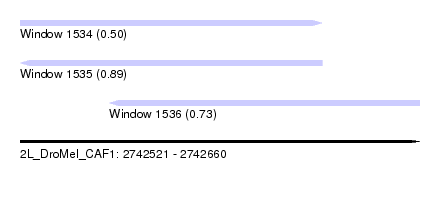
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 2,742,521 – 2,742,660 |
| Length | 139 |
| Max. P | 0.894119 |

| Location | 2,742,521 – 2,742,626 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 74.46 |
| Mean single sequence MFE | -34.17 |
| Consensus MFE | -10.53 |
| Energy contribution | -10.12 |
| Covariance contribution | -0.42 |
| Combinations/Pair | 1.38 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.31 |
| SVM decision value | -0.07 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
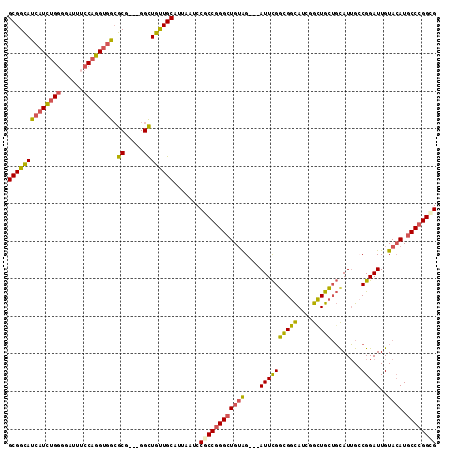
>2L_DroMel_CAF1 2742521 105 + 22407834 CCCAACGGACAUGGCCAGUUUCAGGCCAUUCCGCCGGGCAUGUACAAUCCGGCAAUACAGCAGCCGAUGCCGCCGAAU---CUCCAGCCCGGCGGAUUAAUGCAACAG .((...(((.((((((.......)))))))))(((((((.((.......((((......(((.....))).))))...---...)))))))))))............. ( -39.32) >DroPse_CAF1 198947 89 + 1 CCCAAUGGCCACGGACAGUUCCAGGGCAUUCCGCCGGGCAUGUACAACCCGGCCAUUGCCCAGCCA---CCGACAAAU-----GCAGCCCCAGGAAU----------- .((...(((...(((....))).(((((....((((((.........))))))...))))).....---.........-----...)))...))...----------- ( -29.80) >DroEre_CAF1 179420 105 + 1 CCCAACGGACAUGGCCAGUUCCAGGCCAUUCCACCGGGCAUGUACAAUCCGUCAAUGCAGCAACCGUUGCCGCCGAAU---CUACAGCCCGGGGGAUUAAUGCAACAA (((...(((.((((((.......))))))))).((((((.((((.(.((.((....(((((....))))).)).)).)---.)))))))))))))............. ( -41.40) >DroYak_CAF1 170999 105 + 1 CCCAACGGACAUGGUCAGUUCCAGGCCAUUCCGCCGGGCAUGUACAAUCCGUCAAUGCAGCAGCCGAUGCCGCCGAAU---CUACAGCCCGGUGGAUUAAUGCAACAA ......((...(((......)))..))..((((((((((.((((.......((...((.(((.....))).)).))..---.))))))))))))))............ ( -34.40) >DroAna_CAF1 265860 108 + 1 CCCAAUGGUCAUGGACAGUUCCAGGGUAUUCCACCUGGUAUUUACAAUCCGGCCAUGACCCAGCAAAUACCAGCAAAUAUUCCACAGCCUGGUGGAUUGAUGCAGCAA ......((((((((..(((.(((((........))))).)))..........))))))))............(((.....(((((......)))))....)))..... ( -30.30) >DroPer_CAF1 197631 89 + 1 CCCAAUGGCCACGGACAGUUCCAGGGCAUUCCGCCGGGCAUGUACAACCCGGCCAUUGCCCAGCCA---CCGACAAAU-----GCAGCCCCAGGAAU----------- .((...(((...(((....))).(((((....((((((.........))))))...))))).....---.........-----...)))...))...----------- ( -29.80) >consensus CCCAACGGACAUGGACAGUUCCAGGCCAUUCCGCCGGGCAUGUACAAUCCGGCAAUGCACCAGCCAAUGCCGCCAAAU___CUACAGCCCGGGGGAUUAAUGCAACAA .....(((....((......))..((......((((((.........))))))......))........))).................................... (-10.53 = -10.12 + -0.42)

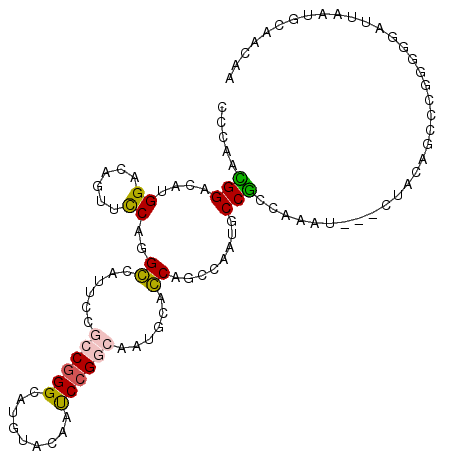
| Location | 2,742,521 – 2,742,626 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 74.46 |
| Mean single sequence MFE | -41.47 |
| Consensus MFE | -22.40 |
| Energy contribution | -21.51 |
| Covariance contribution | -0.89 |
| Combinations/Pair | 1.44 |
| Mean z-score | -1.90 |
| Structure conservation index | 0.54 |
| SVM decision value | 0.98 |
| SVM RNA-class probability | 0.894119 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2742521 105 - 22407834 CUGUUGCAUUAAUCCGCCGGGCUGGAG---AUUCGGCGGCAUCGGCUGCUGUAUUGCCGGAUUGUACAUGCCCGGCGGAAUGGCCUGAAACUGGCCAUGUCCGUUGGG ............((((((((((((..(---(((((((((((.....)))).....))))))))...)).))))))))))((((((.......)))))).......... ( -49.30) >DroPse_CAF1 198947 89 - 1 -----------AUUCCUGGGGCUGC-----AUUUGUCGG---UGGCUGGGCAAUGGCCGGGUUGUACAUGCCCGGCGGAAUGCCCUGGAACUGUCCGUGGCCAUUGGG -----------...((..((((..(-----....(.(((---(..(.(((((.(.(((((((.......))))))).)..))))).)..)))).).)..))).)..)) ( -40.60) >DroEre_CAF1 179420 105 - 1 UUGUUGCAUUAAUCCCCCGGGCUGUAG---AUUCGGCGGCAACGGUUGCUGCAUUGACGGAUUGUACAUGCCCGGUGGAAUGGCCUGGAACUGGCCAUGUCCGUUGGG .............((((((((((((((---(((((((((((.....)))))).....)))))).)))).)))))).(((((((((.......)))))).)))...))) ( -44.70) >DroYak_CAF1 170999 105 - 1 UUGUUGCAUUAAUCCACCGGGCUGUAG---AUUCGGCGGCAUCGGCUGCUGCAUUGACGGAUUGUACAUGCCCGGCGGAAUGGCCUGGAACUGACCAUGUCCGUUGGG ............(((.(((((((((((---(((((((((((.....)))))).....)))))).)))).)))))).)))(((((.(((......))).).)))).... ( -39.40) >DroAna_CAF1 265860 108 - 1 UUGCUGCAUCAAUCCACCAGGCUGUGGAAUAUUUGCUGGUAUUUGCUGGGUCAUGGCCGGAUUGUAAAUACCAGGUGGAAUACCCUGGAACUGUCCAUGACCAUUGGG .(((((((....(((((......))))).....))).)))).......((((((((.(((((.....)).(((((.(.....))))))..))).))))))))...... ( -34.20) >DroPer_CAF1 197631 89 - 1 -----------AUUCCUGGGGCUGC-----AUUUGUCGG---UGGCUGGGCAAUGGCCGGGUUGUACAUGCCCGGCGGAAUGCCCUGGAACUGUCCGUGGCCAUUGGG -----------...((..((((..(-----....(.(((---(..(.(((((.(.(((((((.......))))))).)..))))).)..)))).).)..))).)..)) ( -40.60) >consensus UUGUUGCAUUAAUCCCCCGGGCUGUAG___AUUCGGCGGCAUCGGCUGCGGCAUGGCCGGAUUGUACAUGCCCGGCGGAAUGCCCUGGAACUGUCCAUGUCCAUUGGG ............(((.((((((........((((((((((....)))))........))))).......)))))).)))..(((.(((......))).)))....... (-22.40 = -21.51 + -0.89)

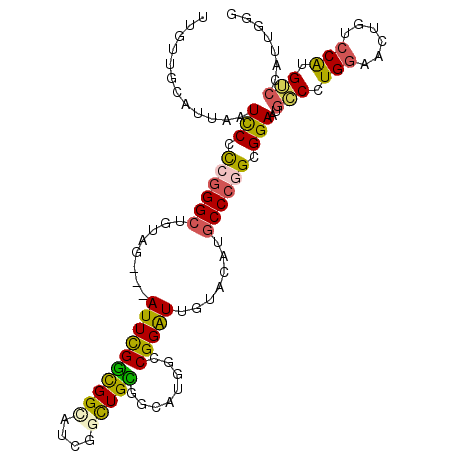
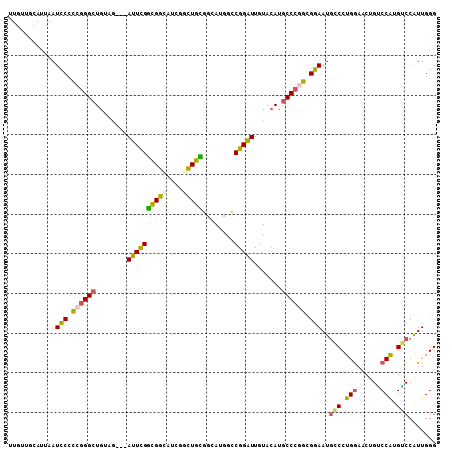
| Location | 2,742,552 – 2,742,660 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 85.58 |
| Mean single sequence MFE | -49.58 |
| Consensus MFE | -38.32 |
| Energy contribution | -38.05 |
| Covariance contribution | -0.27 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.46 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.42 |
| SVM RNA-class probability | 0.729117 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
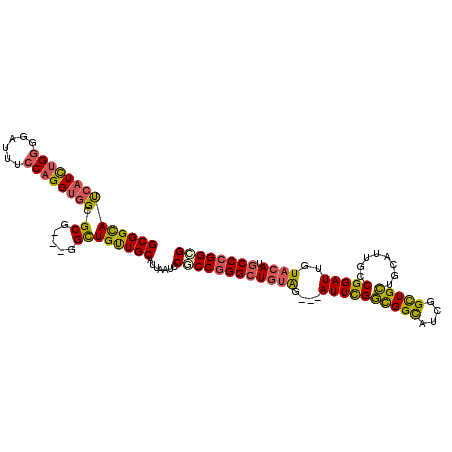
>2L_DroMel_CAF1 2742552 108 - 22407834 GCGGCAUCAUCUGGGGAUUUCCAGGUGGCGCG---GGCUGUUGCAUUAAUCCGCCGGGCUGGAG---AUUCGGCGGCAUCGGCUGCUGUAUUGCCGGAUUGUACAUGCCCGGCG ((((((.((((((((....))))))))((...---.)))))))).......((((((((((..(---(((((((((((.....)))).....))))))))...)).)))))))) ( -51.10) >DroSec_CAF1 161046 108 - 1 GCGGCAUCAUCUGGGGAUUUCCAGGUGGCGCG---GGCUGUUGCAUUAAUCCGCCGGGCUGUAG---AUUCGGCGGCAUUGGCUGCUGCAUUGCCGGAUUGUACAUGCCCGGCG ((((((.((((((((....))))))))((...---.)))))))).......(((((((((((((---(((((((.(((........)))...)))))))).)))).)))))))) ( -53.00) >DroSim_CAF1 167158 108 - 1 GCGGCAUCAUCUGGGGAUUUCCAGGUGGCGCG---GGCUGUUGCAUUAAUCCGCCGGGCUGUAG---AUUCGGCGGCAUUGGCUGCUGCAUUGCCGGAUUGUACAUGCCCGGCG ((((((.((((((((....))))))))((...---.)))))))).......(((((((((((((---(((((((.(((........)))...)))))))).)))).)))))))) ( -53.00) >DroEre_CAF1 179451 108 - 1 GCGGCAUCAUCUGGGGAUUUCCAGGUGGCGCG---GGUUGUUGCAUUAAUCCCCCGGGCUGUAG---AUUCGGCGGCAACGGUUGCUGCAUUGACGGAUUGUACAUGCCCGGUG ...((..((((((((....))))))))..))(---(((((......)))))).(((((((((((---(((((((((((.....)))))).....)))))).)))).)))))).. ( -47.00) >DroYak_CAF1 171030 108 - 1 GCGGCAUCAUCUGGGGAUUACCAGGUGGCGCG---GGUUGUUGCAUUAAUCCACCGGGCUGUAG---AUUCGGCGGCAUCGGCUGCUGCAUUGACGGAUUGUACAUGCCCGGCG ...((..(((((((......)))))))..))(---(((((......)))))).(((((((((((---(((((((((((.....)))))).....)))))).)))).)))))).. ( -49.00) >DroAna_CAF1 265891 114 - 1 GCGGUACCAUUUGUCCAUUACCCGGCUGGGCCGGUGGUUGCUGCAUCAAUCCACCAGGCUGUGGAAUAUUUGCUGGUAUUUGCUGGGUCAUGGCCGGAUUGUAAAUACCAGGUG ((((((((((..(((((.........)))))..)))).)))))).....(((((......))))).......((((((((((((.(((....))).)...)))))))))))... ( -44.40) >consensus GCGGCAUCAUCUGGGGAUUUCCAGGUGGCGCG___GGCUGUUGCAUUAAUCCGCCGGGCUGUAG___AUUCGGCGGCAUCGGCUGCUGCAUUGCCGGAUUGUACAUGCCCGGCG ((((((((((((((......)))))))).((.....)))))))).......((((((((((((....((((((((((....)))))........)))))..)))).)))))))) (-38.32 = -38.05 + -0.27)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:49:36 2006