
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 2,722,369 – 2,722,507 |
| Length | 138 |
| Max. P | 0.968476 |

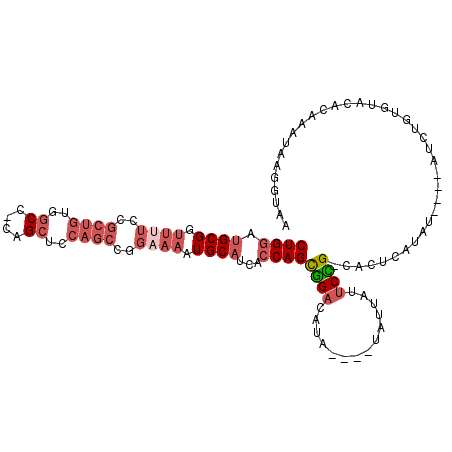
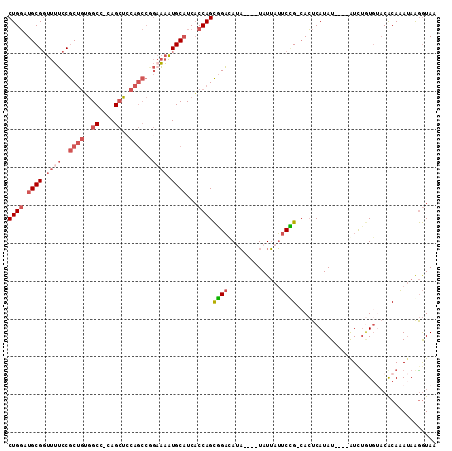
| Location | 2,722,369 – 2,722,472 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 77.42 |
| Mean single sequence MFE | -33.09 |
| Consensus MFE | -13.33 |
| Energy contribution | -14.97 |
| Covariance contribution | 1.64 |
| Combinations/Pair | 1.14 |
| Mean z-score | -3.00 |
| Structure conservation index | 0.40 |
| SVM decision value | 1.63 |
| SVM RNA-class probability | 0.968476 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2722369 103 - 22407834 CUGGAUGCGGUUUUCCGCUGUGGCC-CAGCUCCAGCCGGAAAAUGCAUCACCAGCGGACAUA----UAUUAUUCCG-CACUCAUAU----GUUUGUGUACACAAAUAGGGUAA ...((((((.((((((((((..((.-..))..))).)))))))))))))(((.(((((....----......))))-).......(----((((((....))))))).))).. ( -38.50) >DroVir_CAF1 205753 100 - 1 CUGUCUGCGGUUGGCCACA-AAGCCACAGAUUUC-----AUUGUGCCUCUCCAGUUGCUACU---GUAUA-GACAGUAAAUCAUAUA---AUCUAUGCACGCAUAAAAAAUAU ..(((((.((((.......-.)))).)))))...-----.((((((......(((....)))---(((((-((..(((.....))).---.)))))))..))))))....... ( -23.30) >DroSec_CAF1 141142 103 - 1 CUGGAUGCGGUUUUCCGCUGUGGCC-CAGCUCCAGCCGGAAAAUGCAUCACCAGCGGACAUA----UAUUAUUCCG-CACUCAUAU----GUUUGUGUACACAAAUAGGGUAA ...((((((.((((((((((..((.-..))..))).)))))))))))))(((.(((((....----......))))-).......(----((((((....))))))).))).. ( -38.50) >DroSim_CAF1 144118 102 - 1 CUGGAUGCG-UUUUCCGCUGUGGCC-CAGCUCCAGCCGGAAAAUGCAUCACCAGCGGACAUA----UAUUAUUCCG-CACUCAUAU----GUUUGUGUACACAAAUAGGGUAA ...((((((-((((((((((..((.-..))..))).)))))))))))))(((.(((((....----......))))-).......(----((((((....))))))).))).. ( -42.30) >DroEre_CAF1 159409 107 - 1 CUGGCUGCGGUUUUCCGCUGUGGCC-CAGCUCCAGCCAGAAAAUGCAUCACCAGCGGACACAUUUGUAUUAUUCCG-CACUCAUAU----AUCCUUCAUCACAAAUAAGGUAA (((((((.((....))((((.....-))))..)))))))..............(((((..............))))-)........----..((((..........))))... ( -25.24) >DroYak_CAF1 149219 107 - 1 CUGGUUGCGGUUUUCCGCUGUGGCC-CAGCUGCAGCCAGCAAAUGCAUCACCAGCGGACAUA----UAUUAUUCUG-CACUCAUAUACAUAUCUGUGUACGCAAAUAAGGUAA ((((((((((....))((((.....-)))).))))))))....(((.......(((((....----......))))-).....((((((....)))))).))).......... ( -30.70) >consensus CUGGAUGCGGUUUUCCGCUGUGGCC_CAGCUCCAGCCGGAAAAUGCAUCACCAGCGGACAUA____UAUUAUUCCG_CACUCAUAU____AUCUGUGUACACAAAUAAGGUAA ((((.((((.((((..((((..((....))..))))..)))).))))...))))((((..............))))..................................... (-13.33 = -14.97 + 1.64)

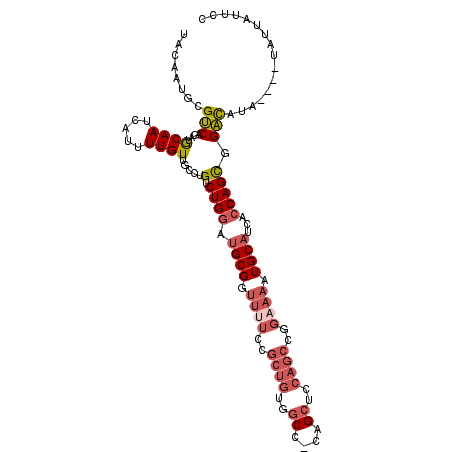
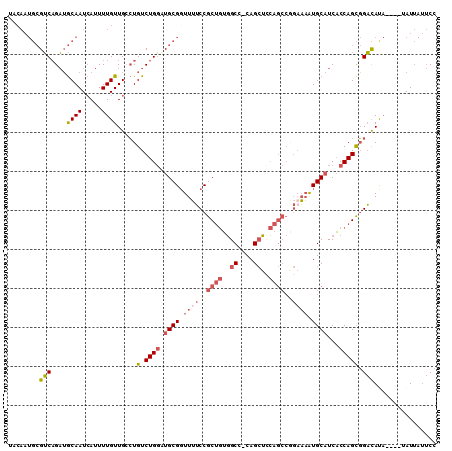
| Location | 2,722,402 – 2,722,507 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 80.24 |
| Mean single sequence MFE | -35.53 |
| Consensus MFE | -17.94 |
| Energy contribution | -19.25 |
| Covariance contribution | 1.31 |
| Combinations/Pair | 1.15 |
| Mean z-score | -3.02 |
| Structure conservation index | 0.50 |
| SVM decision value | 1.21 |
| SVM RNA-class probability | 0.931449 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2722402 105 - 22407834 UACAAUGCGUCAGAUGCAAUCAUUUUGUUGCCUGUCUGGAUGCGGUUUUCCGCUGUGGCC-CAGCUCCAGCCGGAAAAUGCAUCACCAGCGGACAUA----UAUUAUUCC .....(((..((((((((((......)))))..)))))((((((.((((((((((..((.-..))..))).)))))))))))))....)))......----......... ( -34.80) >DroVir_CAF1 205788 93 - 1 U-------GGCUGCAACAAUAAUUUUGUUGCAUGUCUGUCUGCGGUUGGCCACA-AAGCCACAGAUUUC-----AUUGUGCCUCUCCAGUUGCUACU---GUAUA-GACA .-------(((.(((((((.....)))))))(((...(((((.((((.......-.)))).)))))..)-----))...)))(((.((((....)))---)...)-)).. ( -28.20) >DroSec_CAF1 141175 105 - 1 UACAAUGCGUCAGAUGCAAUCAUUUUGUUGCCUGUCUGGAUGCGGUUUUCCGCUGUGGCC-CAGCUCCAGCCGGAAAAUGCAUCACCAGCGGACAUA----UAUUAUUCC .....(((..((((((((((......)))))..)))))((((((.((((((((((..((.-..))..))).)))))))))))))....)))......----......... ( -34.80) >DroSim_CAF1 144151 104 - 1 CACAAUGCGUCAGAUGCAAUCAUUUUGUUGCCUGUCUGGAUGCG-UUUUCCGCUGUGGCC-CAGCUCCAGCCGGAAAAUGCAUCACCAGCGGACAUA----UAUUAUUCC .....(((..((((((((((......)))))..)))))((((((-((((((((((..((.-..))..))).)))))))))))))....)))......----......... ( -38.60) >DroEre_CAF1 159442 109 - 1 UACAAUGCGUCAGAUGCAAUCAUUUUGUUGGCUGUCUGGCUGCGGUUUUCCGCUGUGGCC-CAGCUCCAGCCAGAAAAUGCAUCACCAGCGGACACAUUUGUAUUAUUCC (((((((.(((.((((((....((((.(((((((.(((((..((((.....))))..).)-)))...))))))))))))))))).(....)))).)).)))))....... ( -36.70) >DroYak_CAF1 149256 105 - 1 CACAAUGCGUCAGAUGCAAUCAUUUUGUUGGCUGUCUGGUUGCGGUUUUCCGCUGUGGCC-CAGCUGCAGCCAGCAAAUGCAUCACCAGCGGACAUA----UAUUAUUCU .....(((....((((((.....(((((((((((((((((..((((.....))))..).)-)))..))))))))))))))))))....)))......----......... ( -40.10) >consensus UACAAUGCGUCAGAUGCAAUCAUUUUGUUGCCUGUCUGGAUGCGGUUUUCCGCUGUGGCC_CAGCUCCAGCCGGAAAAUGCAUCACCAGCGGACAUA____UAUUAUUCC ........(((....((((.....)))).....(.((((.((((.((((..((((..((....))..))))..)))).))))...))))).)))................ (-17.94 = -19.25 + 1.31)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:49:25 2006