
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
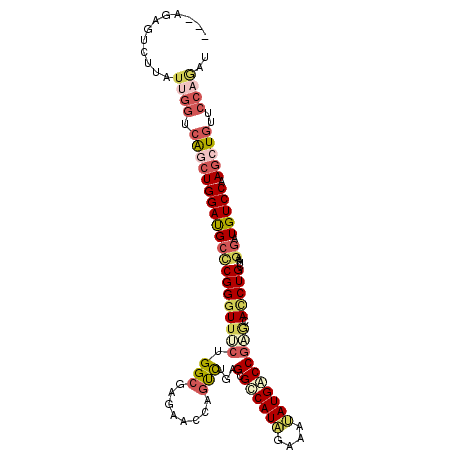
| Location | 2,654,422 – 2,654,528 |
| Length | 106 |
| Max. P | 0.672146 |

| Location | 2,654,422 – 2,654,528 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | forward |
| Mean pairwise identity | 77.63 |
| Mean single sequence MFE | -41.13 |
| Consensus MFE | -19.35 |
| Energy contribution | -22.13 |
| Covariance contribution | 2.78 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.47 |
| SVM decision value | 0.12 |
| SVM RNA-class probability | 0.593578 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2654422 106 + 22407834 ---AGAGUCUUAUUGGUCAGCUGGAUGCCCGGGUUUCUGGCGAGAGCCAGUCUGAGCGUCAUAGAAAUAUGACCGAGCACCUGCUAAGGAUGUCCAAGCUGUUCCAGAU ---.........((((.(((((((((((.((((((((((((....)))))...)))).(((((....)))))))).)).(((....)))..)))).)))))..)))).. ( -43.30) >DroSec_CAF1 75267 106 + 1 ---AGAGUCUUAUUGGUCAGCUGGAUGCCCGGGUUUCUGGCGAGAGCCAGUCUGAGCGUCAUAGAAAUAUGACCGAGCACCUGCUUCGGAUGUCCAAGCUGUUCCAGAU ---.........((((.(((((((((..(((.(((((((((....)))))...))))((((((....)))))).((((....)))))))..)))).)))))..)))).. ( -44.80) >DroSim_CAF1 76308 106 + 1 ---AGAGUCUUAUCAGUCAGCUGGAUGCCCGGGUUUCUGGCGAGAGCCAGUCUGAGCGUCAUAGAAAUAUGACCGAGCACCUGCUGCGGAUGUCCAAGCUGUUCCAGAU ---...((((...((((((((.((.(((.((((((((((((....)))))...)))).(((((....)))))))).))))).)))).((....))..))))....)))) ( -40.80) >DroEre_CAF1 83640 106 + 1 ---AGAGCCUCAUUGGCCAACUGGAUGCUCGGGUUUCCGGCGAGAAUCAGUCUGUGCGCCAUAGAGAGAUGACCGCGCACCUGCACAGGAUGUCCAAGCUGUUCCAGAU ---.(.(((.....))))..(((((.(((.(((..(((.(((...(((..((((((...))))))..)))...)))((....))...)))..))).)))...))))).. ( -38.10) >DroYak_CAF1 78689 106 + 1 ---AGCGUCUCAUUGGUCAACUGGAUGCCCGGGUUGCUGGCGAGAAUCAGUCUGAGCGCCAUAGGAAUAUGACCGCACAUCUGCAAAGGAUGUCCAAGCUGUUCCAAAU ---(((........(((((.((((....))))(((.((((((....((.....)).))))..)).))).)))))(.((((((.....)))))).)..)))......... ( -29.80) >DroPer_CAF1 94721 109 + 1 UGGAGAAGGUGAUCGCCAGGCUGGACGACCGGGUGGAGGGGGCGGAGCAGCUGCGGUGCCAUCGCCAGAUGGCCAAGAAGCUGCGGCACCUGUCCAACCUGUUCCUCUA (((((..(((....))).........((.((((((((.(((((...((((((...(.((((((....)))))))....)))))).)).))).))).))))).))))))) ( -50.00) >consensus ___AGAGUCUUAUUGGUCAGCUGGAUGCCCGGGUUUCUGGCGAGAACCAGUCUGAGCGCCAUAGAAAUAUGACCGAGCACCUGCUAAGGAUGUCCAAGCUGUUCCAGAU ............((((.((((((((((((((((((((.(((........)))...(.((((((....)))))))))).)))))....)).))))).)))))..)))).. (-19.35 = -22.13 + 2.78)

| Location | 2,654,422 – 2,654,528 |
|---|---|
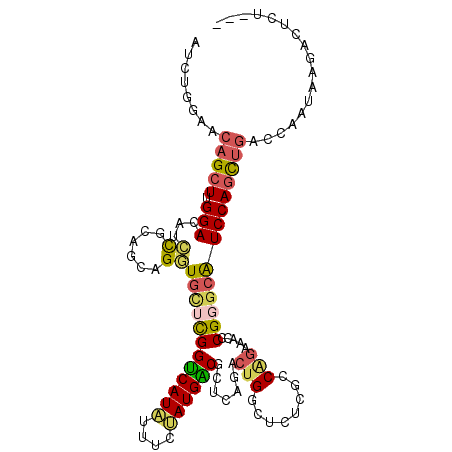
| Length | 106 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 77.63 |
| Mean single sequence MFE | -36.95 |
| Consensus MFE | -20.20 |
| Energy contribution | -20.35 |
| Covariance contribution | 0.15 |
| Combinations/Pair | 1.40 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.29 |
| SVM RNA-class probability | 0.672146 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2654422 106 - 22407834 AUCUGGAACAGCUUGGACAUCCUUAGCAGGUGCUCGGUCAUAUUUCUAUGACGCUCAGACUGGCUCUCGCCAGAAACCCGGGCAUCCAGCUGACCAAUAAGACUCU--- ...(((..(((((.(((...(((....)))((((((((((((....)))))).......(((((....))))).....)))))))))))))).)))..........--- ( -36.00) >DroSec_CAF1 75267 106 - 1 AUCUGGAACAGCUUGGACAUCCGAAGCAGGUGCUCGGUCAUAUUUCUAUGACGCUCAGACUGGCUCUCGCCAGAAACCCGGGCAUCCAGCUGACCAAUAAGACUCU--- ...(((..((((((((....))))....((((((((((((((....)))))).......(((((....))))).....))))))))..)))).)))..........--- ( -36.90) >DroSim_CAF1 76308 106 - 1 AUCUGGAACAGCUUGGACAUCCGCAGCAGGUGCUCGGUCAUAUUUCUAUGACGCUCAGACUGGCUCUCGCCAGAAACCCGGGCAUCCAGCUGACUGAUAAGACUCU--- .(((....(((...((....)).((((.((((((((((((((....)))))).......(((((....))))).....))))))))..)))).)))...)))....--- ( -36.50) >DroEre_CAF1 83640 106 - 1 AUCUGGAACAGCUUGGACAUCCUGUGCAGGUGCGCGGUCAUCUCUCUAUGGCGCACAGACUGAUUCUCGCCGGAAACCCGAGCAUCCAGUUGGCCAAUGAGGCUCU--- ..(((((...((((((...(((.((((((((((((.((.........)).))))))...))).....))).)))...)))))).)))))..((((.....))))..--- ( -36.20) >DroYak_CAF1 78689 106 - 1 AUUUGGAACAGCUUGGACAUCCUUUGCAGAUGUGCGGUCAUAUUCCUAUGGCGCUCAGACUGAUUCUCGCCAGCAACCCGGGCAUCCAGUUGACCAAUGAGACGCU--- ..((((..(((((.(((..(((.((((.((((((....))))))....(((((...(((.....))))))))))))...)))..)))))))).)))).........--- ( -27.20) >DroPer_CAF1 94721 109 - 1 UAGAGGAACAGGUUGGACAGGUGCCGCAGCUUCUUGGCCAUCUGGCGAUGGCACCGCAGCUGCUCCGCCCCCUCCACCCGGUCGUCCAGCCUGGCGAUCACCUUCUCCA ....(((.((((((((((.((((..((((((.(...((((((....))))))...).))))))..))))....((....))..))))))))))..((......))))). ( -48.90) >consensus AUCUGGAACAGCUUGGACAUCCGCAGCAGGUGCUCGGUCAUAUUUCUAUGACGCUCAGACUGGCUCUCGCCAGAAACCCGGGCAUCCAGCUGACCAAUAAGACUCU___ ........(((((.(((...((......))((((((((((((....)))))).......(((........))).....))))))))))))))................. (-20.20 = -20.35 + 0.15)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:48:51 2006