
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 2,556,142 – 2,556,267 |
| Length | 125 |
| Max. P | 0.985233 |

| Location | 2,556,142 – 2,556,250 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 80.43 |
| Mean single sequence MFE | -40.35 |
| Consensus MFE | -25.74 |
| Energy contribution | -27.78 |
| Covariance contribution | 2.05 |
| Combinations/Pair | 1.16 |
| Mean z-score | -4.03 |
| Structure conservation index | 0.64 |
| SVM decision value | 1.87 |
| SVM RNA-class probability | 0.980591 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
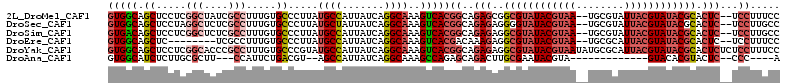
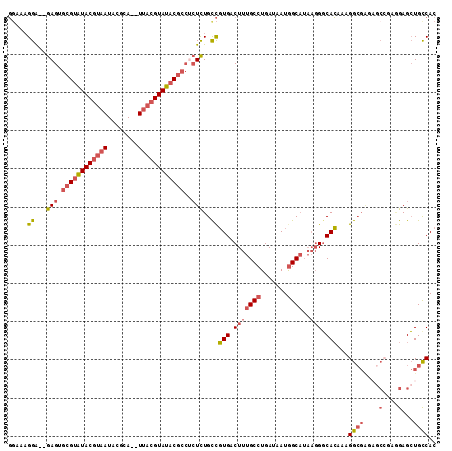
>2L_DroMel_CAF1 2556142 108 + 22407834 GUGGCAGCUCCUCGGCUAUCGCCUUUGUGCCCUUAUGCCAUUAUCAGGCAAAGUCACGGCAGAGCGGCGUAUACGUAA--UGCGUAUUACGUAUACGCACUC--UCCUUUCC (((((.((.(...(((....)))...).)).....((((.......))))..)))))((.((((..((((((((((((--(....))))))))))))).)))--)))..... ( -42.30) >DroSec_CAF1 135569 108 + 1 GUGGCAGCUCCUAGGCUCUCGCCUUUGUGCCCUUAUGCUAUUAUCAGGCAAAGUCACGGCAGAGAGGGGUAUACGUAA--UGCGUAUUACGUAUACGCACUC--UCCUUGCC (((((.((.(..((((....))))..).)).....((((.......))))..)))))((((((((((.((((((((((--(....))))))))))).).)))--))..)))) ( -44.80) >DroSim_CAF1 139596 108 + 1 GUGACAGCUCCUCGGCUCUCGCCUUUGUGCCCUUAUGCCAUUAUCAGGCAAAGUCACGGCAGAGAGGCGUAUACGUAA--UGCGUAUUACGUAUACGCACUC--UCCUUGCC (((((.((.(...(((....)))...).)).....((((.......))))..)))))(((((((((((((((((((((--(....))))))))))))).)))--))..)))) ( -48.80) >DroEre_CAF1 139785 100 + 1 GUGGCAGCUC--------UCGCCUUUGUGCCCUUAUGCCAUUAUCAGGCAAAGUCACGACAAAGAGGCGUAUACGUAA--UGCGCAUUACGUAUACGCACUC--UCCUUUCC (((((.((.(--------........).)).....((((.......))))..))))).....((((((((((((((((--(....))))))))))))).)))--)....... ( -36.80) >DroYak_CAF1 139530 112 + 1 GUGGCAGCUCCUCGGCACCCGCCUUUGUGCCCGUAUGCCAUUAUCAGGCAAAGUCACGGCAGAGAGGCGUAUACGUAAUAUGCGCAUUACGUAUACGCACUCUCUCCUUUCC (((((........(((((........)))))....((((.......))))..)))))((.(((((((((((((((((((......))))))))))))).))))))))..... ( -50.30) >DroAna_CAF1 194327 88 + 1 GUGGCAUCUCUUGCGCUU---CCAUUCUGACGU--AGCCAUUAUCAGGCAAAGCCAGAGCAGACUUGCGAAUACGUA-------------GUACACGUACUC--CCC----A ...(((..(((.(((...---.)..((((.(..--.(((.......)))...).)))))))))..)))((.(((((.-------------....))))).))--...----. ( -19.10) >consensus GUGGCAGCUCCUCGGCUCUCGCCUUUGUGCCCUUAUGCCAUUAUCAGGCAAAGUCACGGCAGAGAGGCGUAUACGUAA__UGCGUAUUACGUAUACGCACUC__UCCUUUCC (((((.((.....(((....))).....)).....((((.......))))..)))))((..(((..((((((((((((........)))))))))))).)))...))..... (-25.74 = -27.78 + 2.05)
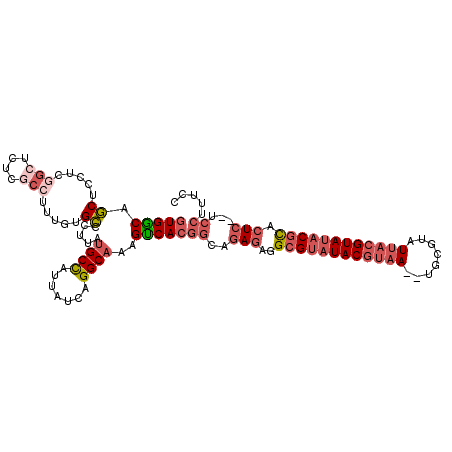
| Location | 2,556,142 – 2,556,250 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 80.43 |
| Mean single sequence MFE | -44.37 |
| Consensus MFE | -25.96 |
| Energy contribution | -27.37 |
| Covariance contribution | 1.41 |
| Combinations/Pair | 1.19 |
| Mean z-score | -4.22 |
| Structure conservation index | 0.59 |
| SVM decision value | 2.00 |
| SVM RNA-class probability | 0.985233 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
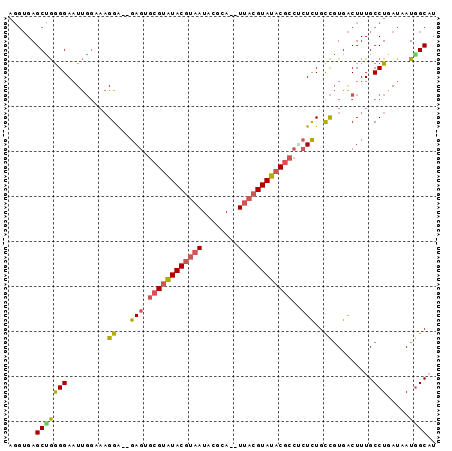
>2L_DroMel_CAF1 2556142 108 - 22407834 GGAAAGGA--GAGUGCGUAUACGUAAUACGCA--UUACGUAUACGCCGCUCUGCCGUGACUUUGCCUGAUAAUGGCAUAAGGGCACAAAGGCGAUAGCCGAGGAGCUGCCAC ((...(((--(((((((((((((((((....)--))))))))))).)))))).(((((.(((((((.......)))).)))..)))...(((....)))..))..)).)).. ( -44.40) >DroSec_CAF1 135569 108 - 1 GGCAAGGA--GAGUGCGUAUACGUAAUACGCA--UUACGUAUACCCCUCUCUGCCGUGACUUUGCCUGAUAAUAGCAUAAGGGCACAAAGGCGAGAGCCUAGGAGCUGCCAC ((((.(((--(((.(.(((((((((((....)--)))))))))).))))))).(((((.((((((.........))).)))..)))..((((....)))).))...)))).. ( -43.50) >DroSim_CAF1 139596 108 - 1 GGCAAGGA--GAGUGCGUAUACGUAAUACGCA--UUACGUAUACGCCUCUCUGCCGUGACUUUGCCUGAUAAUGGCAUAAGGGCACAAAGGCGAGAGCCGAGGAGCUGUCAC ((((..((--(((.(((((((((((((....)--)))))))))))))))))))))(((((..((((.......))))....(((.(...(((....)))...).)))))))) ( -54.40) >DroEre_CAF1 139785 100 - 1 GGAAAGGA--GAGUGCGUAUACGUAAUGCGCA--UUACGUAUACGCCUCUUUGUCGUGACUUUGCCUGAUAAUGGCAUAAGGGCACAAAGGCGA--------GAGCUGCCAC ((...(((--(((.(((((((((((((....)--))))))))))))))))))...(((.(((((((.......)))).)))..)))...(((..--------..))).)).. ( -42.60) >DroYak_CAF1 139530 112 - 1 GGAAAGGAGAGAGUGCGUAUACGUAAUGCGCAUAUUACGUAUACGCCUCUCUGCCGUGACUUUGCCUGAUAAUGGCAUACGGGCACAAAGGCGGGUGCCGAGGAGCUGCCAC ((...((((((((.((((((((((((((....)))))))))))))))))))).))((..(((((((.......))))....(((((........))))))))..))..)).. ( -50.70) >DroAna_CAF1 194327 88 - 1 U----GGG--GAGUACGUGUAC-------------UACGUAUUCGCAAGUCUGCUCUGGCUUUGCCUGAUAAUGGCU--ACGUCAGAAUGG---AAGCGCAAGAGAUGCCAC (----((.--(((((((((...-------------)))))))))((...(((..((((((...(((.......))).--..))))))..))---)...))........))). ( -30.60) >consensus GGAAAGGA__GAGUGCGUAUACGUAAUACGCA__UUACGUAUACGCCUCUCUGCCGUGACUUUGCCUGAUAAUGGCAUAAGGGCACAAAGGCGAGAGCCGAGGAGCUGCCAC .....((...(((.((((((((((((........))))))))))))..)))..))(((.(((((((.......))))..))).)))...((((....(....)...)))).. (-25.96 = -27.37 + 1.41)

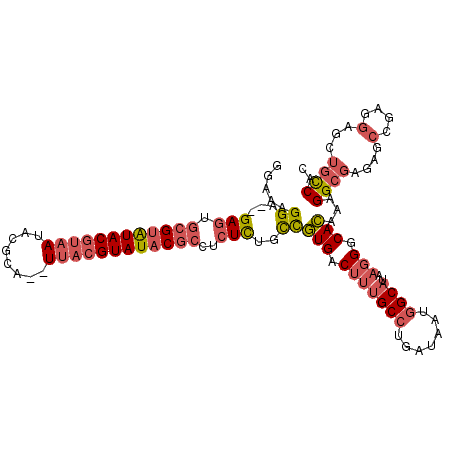
| Location | 2,556,176 – 2,556,267 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 95 |
| Reading direction | forward |
| Mean pairwise identity | 82.17 |
| Mean single sequence MFE | -30.22 |
| Consensus MFE | -17.76 |
| Energy contribution | -18.75 |
| Covariance contribution | 0.99 |
| Combinations/Pair | 1.19 |
| Mean z-score | -4.29 |
| Structure conservation index | 0.59 |
| SVM decision value | 1.58 |
| SVM RNA-class probability | 0.965487 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
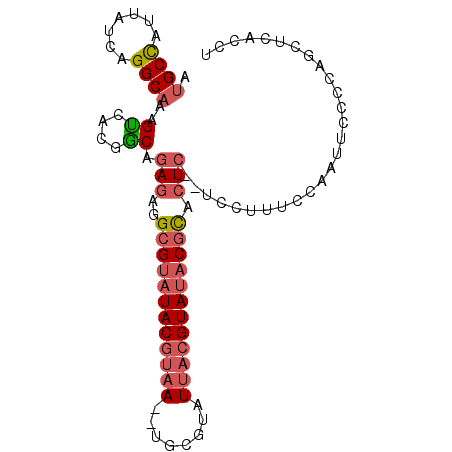
>2L_DroMel_CAF1 2556176 91 + 22407834 AUGCCAUUAUCAGGCAAAGUCACGGCAGAGCGGCGUAUACGUAA--UGCGUAUUACGUAUACGCACUC--UCCUUUCCAAUUCCCCAGCUCACCU .((((.......)))).......((.((((..((((((((((((--(....))))))))))))).)))--)))...................... ( -29.80) >DroSec_CAF1 135603 91 + 1 AUGCUAUUAUCAGGCAAAGUCACGGCAGAGAGGGGUAUACGUAA--UGCGUAUUACGUAUACGCACUC--UCCUUGCCAAUUCCCCAGCUCACCU .((((.......)))).......((((((((((.((((((((((--(....))))))))))).).)))--))..))))................. ( -31.10) >DroSim_CAF1 139630 91 + 1 AUGCCAUUAUCAGGCAAAGUCACGGCAGAGAGGCGUAUACGUAA--UGCGUAUUACGUAUACGCACUC--UCCUUGCCAAUUCCCCAGCUCACCU .((((.......)))).......(((((((((((((((((((((--(....))))))))))))).)))--))..))))................. ( -37.40) >DroEre_CAF1 139811 91 + 1 AUGCCAUUAUCAGGCAAAGUCACGACAAAGAGGCGUAUACGUAA--UGCGCAUUACGUAUACGCACUC--UCCUUUCCAAUUCCCCGGCUCACCU ..(((.......(((...))).......((((((((((((((((--(....))))))))))))).)))--)...............)))...... ( -28.90) >DroYak_CAF1 139564 95 + 1 AUGCCAUUAUCAGGCAAAGUCACGGCAGAGAGGCGUAUACGUAAUAUGCGCAUUACGUAUACGCACUCUCUCCUUUCCAAUUCCCCGGCUCACCU ..(((.......(((...)))..((.(((((((((((((((((((......))))))))))))).)))))))).............)))...... ( -36.20) >DroAna_CAF1 194357 71 + 1 -AGCCAUUAUCAGGCAAAGCCAGAGCAGACUUGCGAAUACGUA-------------GUACACGUACUC--CCC----AGAGUCCUUCGCUC---- -.(((.......))).......((((.(((((..((.(((((.-------------....))))).))--...----.)))))....))))---- ( -17.90) >consensus AUGCCAUUAUCAGGCAAAGUCACGGCAGAGAGGCGUAUACGUAA__UGCGUAUUACGUAUACGCACUC__UCCUUUCCAAUUCCCCAGCUCACCU .((((.......))))..((....)).(((..((((((((((((........)))))))))))).)))........................... (-17.76 = -18.75 + 0.99)

| Location | 2,556,176 – 2,556,267 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 95 |
| Reading direction | reverse |
| Mean pairwise identity | 82.17 |
| Mean single sequence MFE | -33.90 |
| Consensus MFE | -21.75 |
| Energy contribution | -22.02 |
| Covariance contribution | 0.27 |
| Combinations/Pair | 1.33 |
| Mean z-score | -3.36 |
| Structure conservation index | 0.64 |
| SVM decision value | 1.13 |
| SVM RNA-class probability | 0.920498 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
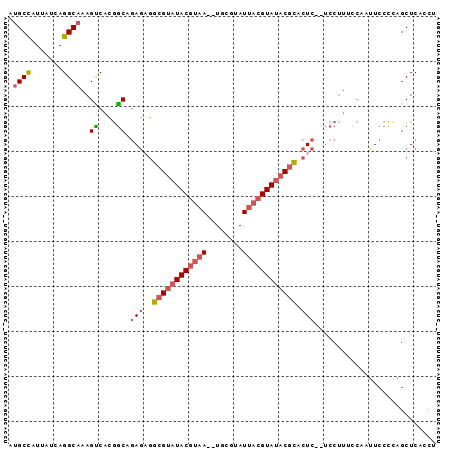
>2L_DroMel_CAF1 2556176 91 - 22407834 AGGUGAGCUGGGGAAUUGGAAAGGA--GAGUGCGUAUACGUAAUACGCA--UUACGUAUACGCCGCUCUGCCGUGACUUUGCCUGAUAAUGGCAU ((((.(.(..(....)..)...(((--(((((((((((((((((....)--))))))))))).)))))).)).).))))((((.......)))). ( -33.10) >DroSec_CAF1 135603 91 - 1 AGGUGAGCUGGGGAAUUGGCAAGGA--GAGUGCGUAUACGUAAUACGCA--UUACGUAUACCCCUCUCUGCCGUGACUUUGCCUGAUAAUAGCAU (((..(....((..((.((((..((--(((.(.(((((((((((....)--)))))))))).))))))))))))..)))..)))........... ( -31.10) >DroSim_CAF1 139630 91 - 1 AGGUGAGCUGGGGAAUUGGCAAGGA--GAGUGCGUAUACGUAAUACGCA--UUACGUAUACGCCUCUCUGCCGUGACUUUGCCUGAUAAUGGCAU .(((..((..(....)..))..(((--(((.(((((((((((((....)--))))))))))))))))))))).......((((.......)))). ( -40.40) >DroEre_CAF1 139811 91 - 1 AGGUGAGCCGGGGAAUUGGAAAGGA--GAGUGCGUAUACGUAAUGCGCA--UUACGUAUACGCCUCUUUGUCGUGACUUUGCCUGAUAAUGGCAU .((....))(((..((.((...(((--(((.(((((((((((((....)--)))))))))))))))))).))))..)))((((.......)))). ( -34.30) >DroYak_CAF1 139564 95 - 1 AGGUGAGCCGGGGAAUUGGAAAGGAGAGAGUGCGUAUACGUAAUGCGCAUAUUACGUAUACGCCUCUCUGCCGUGACUUUGCCUGAUAAUGGCAU ......(((((((....(....((((((((.((((((((((((((....)))))))))))))))))))).))....)....))).....)))).. ( -39.60) >DroAna_CAF1 194357 71 - 1 ----GAGCGAAGGACUCU----GGG--GAGUACGUGUAC-------------UACGUAUUCGCAAGUCUGCUCUGGCUUUGCCUGAUAAUGGCU- ----((((...(((((..----.(.--(((((((((...-------------))))))))).).))))))))).......(((.......))).- ( -24.90) >consensus AGGUGAGCUGGGGAAUUGGAAAGGA__GAGUGCGUAUACGUAAUACGCA__UUACGUAUACGCCUCUCUGCCGUGACUUUGCCUGAUAAUGGCAU ......(((((((.........((...(((.((((((((((((........))))))))))))..)))..)).........))).....)))).. (-21.75 = -22.02 + 0.27)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:48:13 2006