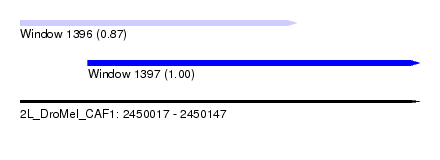
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 2,450,017 – 2,450,147 |
| Length | 130 |
| Max. P | 0.996936 |

| Location | 2,450,017 – 2,450,107 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | forward |
| Mean pairwise identity | 80.83 |
| Mean single sequence MFE | -31.28 |
| Consensus MFE | -19.69 |
| Energy contribution | -20.25 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.36 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.86 |
| SVM RNA-class probability | 0.868746 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2450017 90 + 22407834 CGCUCGCACUUGU-----GC-------AAU------CCAUAUAACAUGGCAUGUG-CGGUGGAUCGGUGAUCAUGAUCGUGAUCAUCGUCAUUAUACCAUCGCUGCCUG .....(((....)-----))-------...------...........((((...(-((((((((((((((((((....)))))))))).....)).))))))))))).. ( -31.40) >DroSec_CAF1 29194 90 + 1 CGCUCUCACUUGU-----GC-------AAU------CCAUAUAGCAUGGCAUGUG-CGGUGGAUCGGUGAUCAUGAUCGUGAUCAUCGUCAUUAUACCAUCGCUGCCUG .((.(......).-----))-------...------...........((((...(-((((((((((((((((((....)))))))))).....)).))))))))))).. ( -30.30) >DroSim_CAF1 30573 90 + 1 CGCUCGCACUUGU-----GC-------AAU------CCAUAUAGCAUGGCAUGUG-CGGUGGAUCGGUGAUCAUGAUCGUGAUCAUCGUCAUUAUACCAUCGCUGCCUG .....(((....)-----))-------...------...........((((...(-((((((((((((((((((....)))))))))).....)).))))))))))).. ( -31.40) >DroEre_CAF1 31278 90 + 1 CGCUCGCACUUGU-----GC-------CAU------CCAUAUUGCAUGGCAUGUG-CGGUGGAUCGGUGAUCAUGAUCGUGAUCAUCGUCAUUAUACCACCGCUGCCUG .((..((((..((-----((-------(((------.(.....).))))))))))-)(((((((((((((((((....)))))))))).....)).)))))))...... ( -37.20) >DroWil_CAF1 45357 98 + 1 CGCUCACUCUAGUCUUGGGCUUAUCCGGAU------CCAUACC--GUGUC-UGUGUCUGUAGAUCUGUAUAGAUUCUUAUGAUCAU--UUAUUUGGCUGGCGCUGACUG .........(((((..(.(((...((((((------(.((((.--(.(((-((......)))))).)))).)))))..((((....--))))..))..))).).))))) ( -20.60) >DroYak_CAF1 29921 96 + 1 CGCUCGCACUUGU-----GC-------CAUCAUAUGCCAUAUAGCAUGGCAUGUG-CGGUGGAUCGGUGAUCAUGAUCGUGAUCAUCGUCAUUAUACCAUCGCUGCCUG .((..(((....)-----))-------....(((((((((.....)))))))))(-((((((((((((((((((....)))))))))).....)).))))))).))... ( -36.80) >consensus CGCUCGCACUUGU_____GC_______AAU______CCAUAUAGCAUGGCAUGUG_CGGUGGAUCGGUGAUCAUGAUCGUGAUCAUCGUCAUUAUACCAUCGCUGCCUG ...............................................((((...(.((((((..((((((((((....))))))))))........))))))))))).. (-19.69 = -20.25 + 0.56)

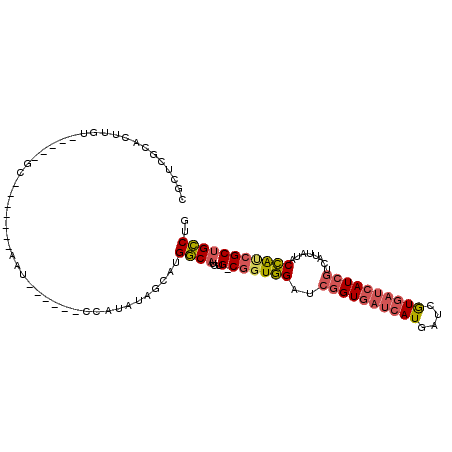
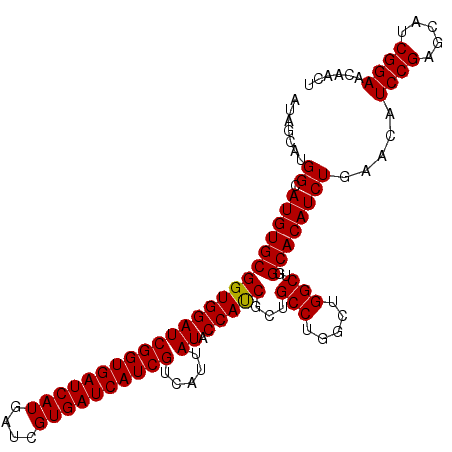
| Location | 2,450,039 – 2,450,147 |
|---|---|
| Length | 108 |
| Sequences | 5 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 98.15 |
| Mean single sequence MFE | -38.66 |
| Consensus MFE | -37.14 |
| Energy contribution | -36.98 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.02 |
| Structure conservation index | 0.96 |
| SVM decision value | 2.77 |
| SVM RNA-class probability | 0.996936 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2450039 108 + 22407834 AUAACAUGGCAUGUGCGGUGGAUCGGUGAUCAUGAUCGUGAUCAUCGUCAUUAUACCAUCGCUGCCUGGCUGGCUGGCACAUCUGAACAUCCGAGCAUCGGAACAACU .......((.((((((((((...((((((((((....))))))))))......))))......(((.....)))..)))))))).....((((.....))))...... ( -36.70) >DroSec_CAF1 29216 108 + 1 AUAGCAUGGCAUGUGCGGUGGAUCGGUGAUCAUGAUCGUGAUCAUCGUCAUUAUACCAUCGCUGCCUGGCUGGCUGGCACAUCUGAACAUCCGAGCAUCGGAACAACU .(((.(((((....(((((((((((((((((((....)))))))))).....)).))))))).(((.....)))..)).))))))....((((.....))))...... ( -37.90) >DroSim_CAF1 30595 108 + 1 AUAGCAUGGCAUGUGCGGUGGAUCGGUGAUCAUGAUCGUGAUCAUCGUCAUUAUACCAUCGCUGCCUGGCUGGCUGGCACAUCUGAACAUCCGAGCAUCGGAACAACU .(((.(((((....(((((((((((((((((((....)))))))))).....)).))))))).(((.....)))..)).))))))....((((.....))))...... ( -37.90) >DroEre_CAF1 31300 108 + 1 AUUGCAUGGCAUGUGCGGUGGAUCGGUGAUCAUGAUCGUGAUCAUCGUCAUUAUACCACCGCUGCCUGGCUGGCUGGCACAUCUGAACCUCCGAGCAUCGGAGCAACU .((((..((.(((((((((((((((((((((((....)))))))))).....)).)))))...(((.....)))..)))))))).....((((.....)))))))).. ( -42.90) >DroYak_CAF1 29949 108 + 1 AUAGCAUGGCAUGUGCGGUGGAUCGGUGAUCAUGAUCGUGAUCAUCGUCAUUAUACCAUCGCUGCCUGGCUGGCUGGCACAUCUGAACAUCCGAGCAUCGGAACAACU .(((.(((((....(((((((((((((((((((....)))))))))).....)).))))))).(((.....)))..)).))))))....((((.....))))...... ( -37.90) >consensus AUAGCAUGGCAUGUGCGGUGGAUCGGUGAUCAUGAUCGUGAUCAUCGUCAUUAUACCAUCGCUGCCUGGCUGGCUGGCACAUCUGAACAUCCGAGCAUCGGAACAACU .......((.(((((((((((((((((((((((....)))))))))).....)).)))))...(((.....)))..)))))))).....((((.....))))...... (-37.14 = -36.98 + -0.16)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:47:23 2006