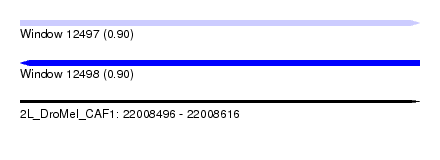
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 22,008,496 – 22,008,616 |
| Length | 120 |
| Max. P | 0.902855 |

| Location | 22,008,496 – 22,008,616 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.89 |
| Mean single sequence MFE | -55.03 |
| Consensus MFE | -33.25 |
| Energy contribution | -33.59 |
| Covariance contribution | 0.34 |
| Combinations/Pair | 1.41 |
| Mean z-score | -2.65 |
| Structure conservation index | 0.60 |
| SVM decision value | 1.00 |
| SVM RNA-class probability | 0.897131 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 22008496 120 + 22407834 UGCAGUUGCUCUGCUACCGCCCCAAUCCGCCGCCAUGGCCGCUUACCUAGCCGCUGCCCAACAAAAUCACCUUCUGCUAACAAAUCCAUUGGCGGCAGCAGCUUCACUGGUCCAACAUGC ((.(((((((..((....))........(((((((.(((.(((.....))).)))((..................))............)))))))))))))).)).............. ( -29.17) >DroVir_CAF1 40338 120 + 1 AGCUGCGGCUCUGCUGCCGCCGCAGUCGGCAGCGAUGGCCGCUUACCUGGCGGCCGCGCAACAGAAUCACCUGCUGCUGACCAAUCCGUUGGCGGCGGCCGCUUCGCUGGUGCAGCAGGC .((((((((...((.((((((((.((((((((((.((((((((.....)))))))))))..(((......))).)))))))(((....))))))))))).))...)))...))))).... ( -67.20) >DroGri_CAF1 49860 120 + 1 GGCUGCUGCGCUGCUGCCGCCGCAGUCGGCAGCGAUGGCUGCCUAUUUGGCGGCCGCUCAACAGAAUCAUUUGCUGCUCACAAAUCCGUUGGCAGCGGCUGCCUCGCUGGUGCAGCAGGC (.(((((((((((((((....))))).((((((((((((((((.....)))))))).))............(((((((.((......)).))))))))))))).....)))))))))).) ( -65.50) >DroWil_CAF1 27502 120 + 1 AGCAGCGGCUUUGCUUCCUCCACAAUCUGCCGCGAUGGCGGCGUACUUGGCAGCCGCGCAGCAGAAUCACUUGCUGCUAACGAAUCCCUUGGCAGCAGCCGCUUCGCUGGUACAGCAGGC .(.((((((................(((((.(((.((((.((.......)).))))))).)))))......(((((((((.(....).))))))))))))))).)((((...)))).... ( -50.00) >DroAna_CAF1 50144 120 + 1 UGCCGCUGCCCUGUUGCCGCCCCAGUCCGCGGCAAUGGCUGCCUACCUGGCGGCCGCCCAGCAGAAUCACCUGCUGCUCACGAAUCCCCUGGCGGCGGCCGCCUCCCUGGUCCAGCAGGC .(((((((..((((((((((........))))))))))..(((.....((((((((((.(((((......)))))((.((.(.....).)))))))))))))).....))).)))).))) ( -60.70) >DroPer_CAF1 70229 120 + 1 UGCCGCGGCACUGCUGCCACCGCAGUCGGCAGCGAUGGCCGCAUACCUGGCGGCUGCACAACAGAAUCACCUGCUGCUGACGAAUCCUCUGGCGGCAGCGGCCUCACUGGUGCAGCAGGC .((((((((..(((((((((....)).)))))))...)))))..........(((((((..(((......(((((((((..((....))...))))))))).....)))))))))).))) ( -57.60) >consensus UGCAGCGGCUCUGCUGCCGCCGCAGUCGGCAGCGAUGGCCGCCUACCUGGCGGCCGCCCAACAGAAUCACCUGCUGCUAACGAAUCCCUUGGCGGCAGCCGCCUCGCUGGUGCAGCAGGC .(((((.((...(((((...........)))))...(((((((.....)))))))))....(((......((((((((((........))))))))))........))).....)).))) (-33.25 = -33.59 + 0.34)

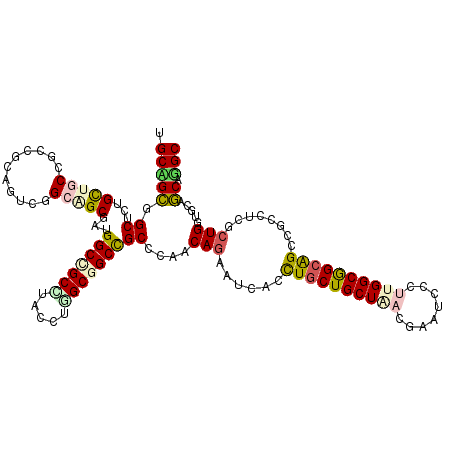
| Location | 22,008,496 – 22,008,616 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.89 |
| Mean single sequence MFE | -58.48 |
| Consensus MFE | -40.57 |
| Energy contribution | -41.02 |
| Covariance contribution | 0.45 |
| Combinations/Pair | 1.39 |
| Mean z-score | -2.37 |
| Structure conservation index | 0.69 |
| SVM decision value | 1.03 |
| SVM RNA-class probability | 0.902855 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 22008496 120 - 22407834 GCAUGUUGGACCAGUGAAGCUGCUGCCGCCAAUGGAUUUGUUAGCAGAAGGUGAUUUUGUUGGGCAGCGGCUAGGUAAGCGGCCAUGGCGGCGGAUUGGGGCGGUAGCAGAGCAACUGCA ...........((((....(((((((((((........(((((((((((.....))))))).))))((.(((((((.....))).)))).)).......)))))))))))....)))).. ( -47.60) >DroVir_CAF1 40338 120 - 1 GCCUGCUGCACCAGCGAAGCGGCCGCCGCCAACGGAUUGGUCAGCAGCAGGUGAUUCUGUUGCGCGGCCGCCAGGUAAGCGGCCAUCGCUGCCGACUGCGGCGGCAGCAGAGCCGCAGCU ((((((((...))))...((((((((.(((((....)))))..((((((((....)))))))))))))))).))))..(((((..(((((((((.(....)))))))).))))))).... ( -66.20) >DroGri_CAF1 49860 120 - 1 GCCUGCUGCACCAGCGAGGCAGCCGCUGCCAACGGAUUUGUGAGCAGCAAAUGAUUCUGUUGAGCGGCCGCCAAAUAGGCAGCCAUCGCUGCCGACUGCGGCGGCAGCAGCGCAGCAGCC (((((((((...((((((((.((((((..((((((((((((.....)))))....))))))))))))).(((.....))).))).)))))((((....)))).))))))).))....... ( -61.50) >DroWil_CAF1 27502 120 - 1 GCCUGCUGUACCAGCGAAGCGGCUGCUGCCAAGGGAUUCGUUAGCAGCAAGUGAUUCUGCUGCGCGGCUGCCAAGUACGCCGCCAUCGCGGCAGAUUGUGGAGGAAGCAAAGCCGCUGCU ((.(((((...))))).((((((((((((.((.(....).)).))))).......((((((((((((((((...))).)))))....))))))))((((.......))))))))))))). ( -49.50) >DroAna_CAF1 50144 120 - 1 GCCUGCUGGACCAGGGAGGCGGCCGCCGCCAGGGGAUUCGUGAGCAGCAGGUGAUUCUGCUGGGCGGCCGCCAGGUAGGCAGCCAUUGCCGCGGACUGGGGCGGCAACAGGGCAGCGGCA (((((((..(((.....((((((((((((((.(.....).)).))((((((....)))))).)))))))))).))).))))(((.(((((((........)))))))...)))...))). ( -66.80) >DroPer_CAF1 70229 120 - 1 GCCUGCUGCACCAGUGAGGCCGCUGCCGCCAGAGGAUUCGUCAGCAGCAGGUGAUUCUGUUGUGCAGCCGCCAGGUAUGCGGCCAUCGCUGCCGACUGCGGUGGCAGCAGUGCCGCGGCA (((((((((..(((((....)))))((......))........)))))))))..............(((((..(((((((.((((((((........)))))))).))).))))))))). ( -59.30) >consensus GCCUGCUGCACCAGCGAAGCGGCCGCCGCCAACGGAUUCGUCAGCAGCAGGUGAUUCUGUUGCGCGGCCGCCAGGUAAGCGGCCAUCGCUGCCGACUGCGGCGGCAGCAGAGCAGCAGCA (((((((((.((((...(((((((((..((...)).........(((((((....))))))).)))))))))......(((((....)))))...))).)...))))))).))....... (-40.57 = -41.02 + 0.45)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:45:05 2006