
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 21,669,010 – 21,669,165 |
| Length | 155 |
| Max. P | 0.986751 |

| Location | 21,669,010 – 21,669,130 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.61 |
| Mean single sequence MFE | -43.42 |
| Consensus MFE | -38.58 |
| Energy contribution | -37.33 |
| Covariance contribution | -1.25 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.54 |
| Structure conservation index | 0.89 |
| SVM decision value | 2.05 |
| SVM RNA-class probability | 0.986751 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
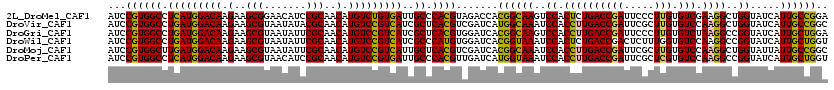
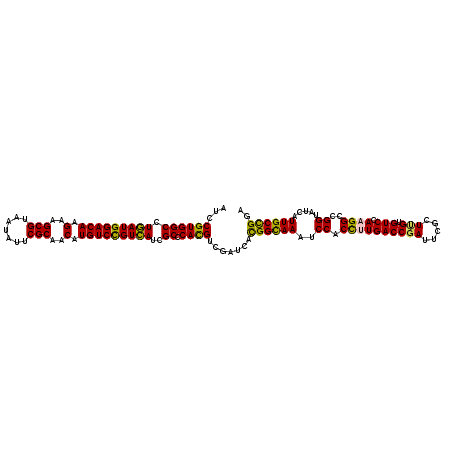
>2L_DroMel_CAF1 21669010 120 - 22407834 AUCCGUGGCCUCAUGGACAAGAAGCGGAACAUCCGCAACAUGUCUGUGAUUGCCCACGUAGACCACGGCAAGUCCACUCUGACCGAUUCCCUUGUGUCGAAGGCUGGUAUCAUUGCCGGA ...((((((.(((((((((.(..((((.....))))..).)))))))))..).))))).......((((((..(((..((...((((........)))).))..))).....)))))).. ( -43.70) >DroVir_CAF1 8174 120 - 1 AUCCGUGGCCUGAUGGACAAGAAGCGUAAUAUACGCAACAUGUCCGUCAUCGCUCACGUCGAUCAUGGCAAAUCCACCUUGACCGAUUCGCUUGUGUCCAAGGCUGGUAUCAUUGCCGGC ...((((((.(((((((((.(..(((((...)))))..).)))))))))..)).)))).......((((((..(((((((((((((.....))).)).))))).))).....)))))).. ( -44.50) >DroGri_CAF1 8329 120 - 1 AUCCGUGGCCUGAUGGACAAGAAGCGUAAUAUUCGCAACAUGUCCGUCAUCGCUCACGUGGAUCACGGCAAGUCCACCUUGACCGAUUCCCUUGUGUCUAAGGCCGGUAUCAUUGCUGGA .(((..(((((((((((((.(..(((.......)))..).))))))))......((((.(((...(((((((.....)))).)))..)))..))))....)))))((((....))))))) ( -44.10) >DroWil_CAF1 3395 120 - 1 AUCCGUGGCCUGAUGGACAAGAAGCGUAAUAUUCGCAACAUGUCCGUCAUCGCCCAUGUGGAUCACGGUAAAUCCACUCUGACCGACUCUUUGGUGUCCAAGGCCGGUAUCAUUGCUGGU ....(.(((.(((((((((.(..(((.......)))..).)))))))))..))))..((((((........))))))....(((((....))))).......(((((((....))))))) ( -41.10) >DroMoj_CAF1 8575 120 - 1 AUCCGUGGCUUGAUGGACAAGAAGCGUAAUAUUCGCAACAUGUCCGUCAUUGCUCACGUCGAUCACGGCAAAUCCACCUUGACCGAUUCGCUUGUGUCCAAGGCUGGUAUUAUUGCCGGC ...((((((.(((((((((.(..(((.......)))..).)))))))))..)).)))).......((((((..(((((((((((((.....))).)).))))).))).....)))))).. ( -45.70) >DroPer_CAF1 2973 120 - 1 AUCCGUGGCCUCAUGGACAAGAAGCGUAACAUCCGCAACAUGUCCGUGAUUGCCCACGUUGAUCAUGGUAAAUCCACCUUGACCGAUUCGCUCGUGUCCAAGGCCGGUAUCAUUGCUGGU ...((((((.(((((((((.(..(((.......)))..).)))))))))..).))))).......(((.....)))((((((((((.....))).)).)))))((((((....)))))). ( -41.40) >consensus AUCCGUGGCCUGAUGGACAAGAAGCGUAAUAUUCGCAACAUGUCCGUCAUCGCCCACGUCGAUCACGGCAAAUCCACCUUGACCGAUUCGCUUGUGUCCAAGGCCGGUAUCAUUGCCGGA ...((((((.(((((((((.(..(((.......)))..).)))))))))..)).)))).......((((((..((.((((((((((.....))).))).))))..)).....)))))).. (-38.58 = -37.33 + -1.25)
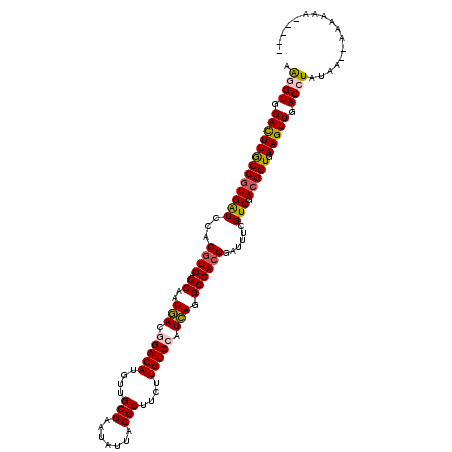
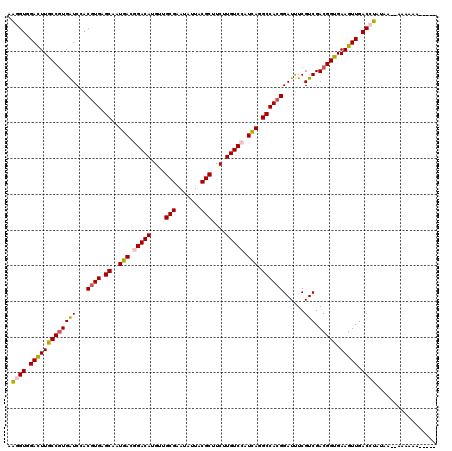
| Location | 21,669,050 – 21,669,165 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.85 |
| Mean single sequence MFE | -40.92 |
| Consensus MFE | -35.49 |
| Energy contribution | -35.18 |
| Covariance contribution | -0.30 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.68 |
| Structure conservation index | 0.87 |
| SVM decision value | 1.56 |
| SVM RNA-class probability | 0.963596 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
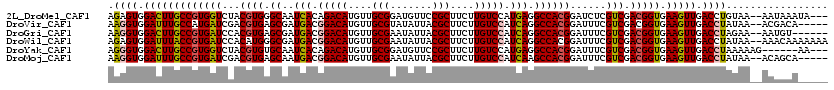
>2L_DroMel_CAF1 21669050 115 + 22407834 AGAGUGGACUUGCCGUGGUCUACGUGGGCAAUCACAGACAUGUUGCGGAUGUUCCGCUUCUUGUCCAUGAGGCCACGGAUCUCGUCGACGGUGAAGUUGACCUGUAA--AAUAAAUA--- .(((........(((((((((.(((((((((..(((....))).((((.....))))...))))))))))))))))))..)))((((((......))))))......--........--- ( -45.40) >DroVir_CAF1 8214 113 + 1 AAGGUGGAUUUGCCAUGAUCGACGUGAGCGAUGACGGACAUGUUGCGUAUAUUACGCUUCUUGUCCAUCAGGCCACGGAUUUCGUCGACGGUGAAGUUGACCUAUAA--ACGACA----- .((((.((((((((....((((((((.((..(((.(((((....(((((...)))))....))))).))).))))).(....)))))).))).))))).))))....--......----- ( -39.20) >DroGri_CAF1 8369 112 + 1 AAGGUGGACUUGCCGUGAUCCACGUGAGCGAUGACGGACAUGUUGCGAAUAUUACGCUUCUUGUCCAUCAGGCCACGGAUUUCGUCGACGGUGAAGUUGACCUAGAA--AAUGU------ .((((.(((((((((((((...((((.((..(((.(((((....(((.......)))....))))).))).))))))......))).))))).))))).))))....--.....------ ( -42.00) >DroWil_CAF1 3435 118 + 1 AGAGUGGAUUUACCGUGAUCCACAUGGGCGAUGACGGACAUGUUGCGAAUAUUACGCUUCUUGUCCAUCAGGCCACGGAUUUCGUCGACGGUGAAGUUGACCUAUAA--AAACAAAAAAA .(((((((((......)))))))...(((..(((.(((((....(((.......)))....))))).))).))).......))((((((......))))))......--........... ( -36.90) >DroYak_CAF1 3154 111 + 1 AGGGUGGACUUGCCGUGGUCUACGUGUGCAAUCACAGACAUGUUGCGGAUGUUCCGCUUCUUGUCCAUGAGGCCACGGAUUUCGUCGACGGUGAAGUUGACCUAAAAAG------AA--- .((((.(((((.(((((((((.((((..(((..(((....))).((((.....))))...)))..)))))))))))))...(((....)))..))))).))))......------..--- ( -42.70) >DroMoj_CAF1 8615 113 + 1 AAGGUGGAUUUGCCGUGAUCGACGUGAGCAAUGACGGACAUGUUGCGAAUAUUACGCUUCUUGUCCAUCAAGCCACGGAUUUCGUCGACGGUGAAGUUGACCUAUAA--ACAGCA----- .((((.((((((((((((.(((((((.((..(((.(((((....(((.......)))....))))).))).))))))....))))).))))).))))).))))....--......----- ( -39.30) >consensus AAGGUGGACUUGCCGUGAUCCACGUGAGCAAUGACGGACAUGUUGCGAAUAUUACGCUUCUUGUCCAUCAGGCCACGGAUUUCGUCGACGGUGAAGUUGACCUAUAA__AAAAAA_____ .((((.(((((((((((((...((((.((..(((.(((((....(((.......)))....))))).))).))))))......))).))))).))))).))))................. (-35.49 = -35.18 + -0.30)
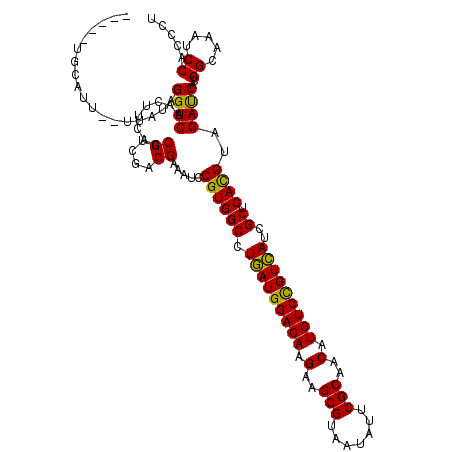
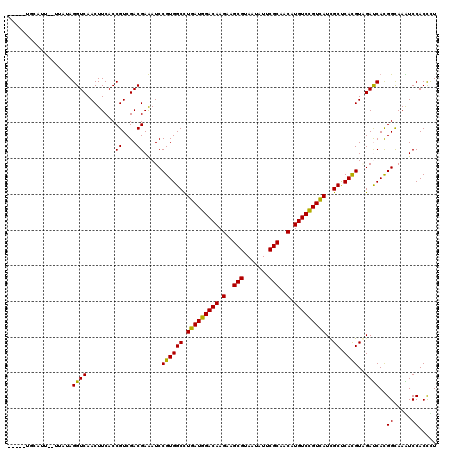
| Location | 21,669,050 – 21,669,165 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 84.85 |
| Mean single sequence MFE | -35.90 |
| Consensus MFE | -30.96 |
| Energy contribution | -29.93 |
| Covariance contribution | -1.03 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.66 |
| Structure conservation index | 0.86 |
| SVM decision value | 1.49 |
| SVM RNA-class probability | 0.958203 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 21669050 115 - 22407834 ---UAUUUAUU--UUACAGGUCAACUUCACCGUCGACGAGAUCCGUGGCCUCAUGGACAAGAAGCGGAACAUCCGCAACAUGUCUGUGAUUGCCCACGUAGACCACGGCAAGUCCACUCU ---........--.....(((.......)))((.(((.....((((((..(((((((((.(..((((.....))))..).)))))))))((((....)))).))))))...))).))... ( -34.80) >DroVir_CAF1 8214 113 - 1 -----UGUCGU--UUAUAGGUCAACUUCACCGUCGACGAAAUCCGUGGCCUGAUGGACAAGAAGCGUAAUAUACGCAACAUGUCCGUCAUCGCUCACGUCGAUCAUGGCAAAUCCACCUU -----((((((--.....(((.......)))(((((((.....)(((((.(((((((((.(..(((((...)))))..).)))))))))..)).))))))))).)))))).......... ( -37.40) >DroGri_CAF1 8369 112 - 1 ------ACAUU--UUCUAGGUCAACUUCACCGUCGACGAAAUCCGUGGCCUGAUGGACAAGAAGCGUAAUAUUCGCAACAUGUCCGUCAUCGCUCACGUGGAUCACGGCAAGUCCACCUU ------.....--....((((..((((..((((.((.....((((((((.(((((((((.(..(((.......)))..).)))))))))..)).)))).)).)))))).))))..)))). ( -35.50) >DroWil_CAF1 3435 118 - 1 UUUUUUUGUUU--UUAUAGGUCAACUUCACCGUCGACGAAAUCCGUGGCCUGAUGGACAAGAAGCGUAAUAUUCGCAACAUGUCCGUCAUCGCCCAUGUGGAUCACGGUAAAUCCACUCU ...(((((((.--.....(((.......)))...)))))))...(.(((.(((((((((.(..(((.......)))..).)))))))))..))))..((((((........))))))... ( -34.00) >DroYak_CAF1 3154 111 - 1 ---UU------CUUUUUAGGUCAACUUCACCGUCGACGAAAUCCGUGGCCUCAUGGACAAGAAGCGGAACAUCCGCAACAUGUCUGUGAUUGCACACGUAGACCACGGCAAGUCCACCCU ---..------.......(((.......)))((.(((.....((((((..(((((((((.(..((((.....))))..).)))))))))((((....)))).))))))...))).))... ( -35.00) >DroMoj_CAF1 8615 113 - 1 -----UGCUGU--UUAUAGGUCAACUUCACCGUCGACGAAAUCCGUGGCUUGAUGGACAAGAAGCGUAAUAUUCGCAACAUGUCCGUCAUUGCUCACGUCGAUCACGGCAAAUCCACCUU -----((((((--.....(((.......)))(((((((.....)(((((.(((((((((.(..(((.......)))..).)))))))))..)).))))))))).)))))).......... ( -38.70) >consensus _____UGCAUU__UUAUAGGUCAACUUCACCGUCGACGAAAUCCGUGGCCUGAUGGACAAGAAGCGUAAUAUUCGCAACAUGUCCGUCAUCGCUCACGUAGAUCACGGCAAAUCCACCCU ..................((((........((....)).....((((((.(((((((((.(..(((.......)))..).)))))))))..)).))))..))))..((.....))..... (-30.96 = -29.93 + -1.03)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:42:44 2006