
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 21,656,020 – 21,656,127 |
| Length | 107 |
| Max. P | 0.997204 |

| Location | 21,656,020 – 21,656,127 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 73.24 |
| Mean single sequence MFE | -50.68 |
| Consensus MFE | -24.85 |
| Energy contribution | -25.16 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.86 |
| Structure conservation index | 0.49 |
| SVM decision value | 2.82 |
| SVM RNA-class probability | 0.997204 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
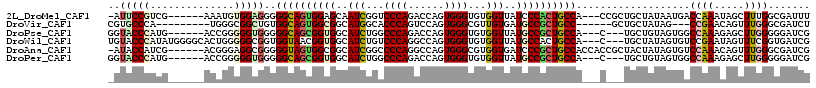
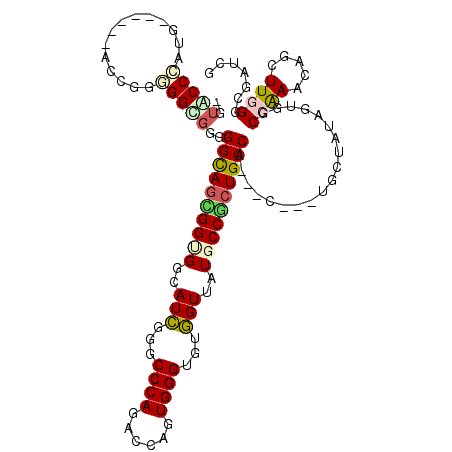
>2L_DroMel_CAF1 21656020 107 + 22407834 -AUUCCGUCG------AAAUGUGGAGGGGGCAGUGGAGCAAUCGGUCCCAGACCAGUGGGUGUGGUUAUCCCACUGCCA---CCGCUGCUAUAAUGACCAAAUAGCUUUGGCGAUUU -....(((((------((..((((....((((((((...(((((..((((......))))..)))))...)))))))).---)))).(((((.........)))))))))))).... ( -39.70) >DroVir_CAF1 49388 99 + 1 CGUGCCCA---------UGGGCGGCUGUGGCAGUGGCGGCAUGGCACCCAGUCCAGUGGGCGUUGUGAUGCCGCUGCC------GCUGCUAUAG---CCGAACAGUUUGGGCGAUCU ..((((((---------((..(((((((((((((((((((.((((.....).)))...(((((....)))))))))))------))))))))))---)))..))...)))))).... ( -60.60) >DroPse_CAF1 33212 105 + 1 GGUACCCAUG------ACCGGGGGUGGGGGCAGCGGUGGCAUCUGGCCCAGACCAGUGGGUGUGGUUAUGCCGCUGCCA---C---UGCUGUAGUGGCCAAAGAGCUUGGGGGAUCG (((.((((.(------..(...(((.(..((((((((((((..(((((((.(((....))).)))....)))).)))))---)---))))))..).)))...)..).))))..))). ( -51.50) >DroWil_CAF1 53170 111 + 1 UGUACCCAUAUGGGGCACUGGGGGCGGUGGUAACGGUGGCAUCUGUCCCAGGCCAGUGGGUGUGGUUAUGCCACUGCCA---C---UGCUAUAGUGUCCGAAUAGUUUCGGUGAUCG ..((((......((((((((..(((((((((...((((((((.....(((.(((....))).)))..))))))))))))---)---)))).))))))))(((....))))))).... ( -53.00) >DroAna_CAF1 32972 110 + 1 -AUACCAUCG------ACGGGAGGCGGGGGUAGUGGCGGCAUCGGCCCCAGGCCAGUGGGCGUGGUGAUCCCGCUGCCACCACCGCUACUAUAGUGUCCAAACAGUUUGGGCGAUCG -.....(((.------((.((.(((((.(((.(..((((.((((...(((.(((....))).))))))).))))..).))).))))).))...))((((((.....))))))))).. ( -47.80) >DroPer_CAF1 48211 105 + 1 GGUACCCAUG------ACCGGGGGUGGGGGCAGCGGUGGCAUCUGGCCCAGACCAGUGGGUGUGGUUAUGCCGCUGCCA---C---UGCUGUAGUGGCCAAAGAGCUUGGGGGAUCG (((.((((.(------..(...(((.(..((((((((((((..(((((((.(((....))).)))....)))).)))))---)---))))))..).)))...)..).))))..))). ( -51.50) >consensus _GUACCCAUG______ACCGGGGGCGGGGGCAGCGGUGGCAUCGGGCCCAGACCAGUGGGUGUGGUUAUGCCGCUGCCA___C___UGCUAUAGUGGCCAAACAGCUUGGGCGAUCG ..(((((..............)))))..((((((((((..(((...((((......))))...)))..))))))))))...................((((.....))))....... (-24.85 = -25.16 + 0.31)
| Location | 21,656,020 – 21,656,127 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 73.24 |
| Mean single sequence MFE | -38.02 |
| Consensus MFE | -18.84 |
| Energy contribution | -19.01 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.40 |
| Mean z-score | -1.81 |
| Structure conservation index | 0.50 |
| SVM decision value | 0.56 |
| SVM RNA-class probability | 0.781676 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
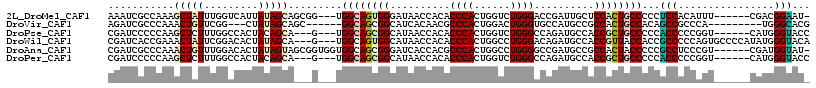
>2L_DroMel_CAF1 21656020 107 - 22407834 AAAUCGCCAAAGCUAUUUGGUCAUUAUAGCAGCGG---UGGCAGUGGGAUAACCACACCCACUGGUCUGGGACCGAUUGCUCCACUGCCCCCUCCACAUUU------CGACGGAAU- .(((.((((((....)))))).)))...((((.((---.((((((.((....(((.(((....))).)))..)).)))))))).)))).((.((.......------.)).))...- ( -33.70) >DroVir_CAF1 49388 99 - 1 AGAUCGCCCAAACUGUUCGG---CUAUAGCAGC------GGCAGCGGCAUCACAACGCCCACUGGACUGGGUGCCAUGCCGCCACUGCCACAGCCGCCCA---------UGGGCACG .....(((((...((..(((---((...(((((------((((..((((((.((...((....))..)))))))).))))))...)))...)))))..))---------)))))... ( -45.40) >DroPse_CAF1 33212 105 - 1 CGAUCCCCCAAGCUCUUUGGCCACUACAGCA---G---UGGCAGCGGCAUAACCACACCCACUGGUCUGGGCCAGAUGCCACCGCUGCCCCCACCCCCGGU------CAUGGGUACC ...........((((..((((((((.....)---)---)(((((((((((..(((.(((....))).))).....)))))...)))))).........)))------)).))))... ( -34.90) >DroWil_CAF1 53170 111 - 1 CGAUCACCGAAACUAUUCGGACACUAUAGCA---G---UGGCAGUGGCAUAACCACACCCACUGGCCUGGGACAGAUGCCACCGUUACCACCGCCCCCAGUGCCCCAUAUGGGUACA ......(((((....)))))......((((.---(---(((((((((.....)))).((((......)))).....)))))).))))............((((((.....)))))). ( -38.20) >DroAna_CAF1 32972 110 - 1 CGAUCGCCCAAACUGUUUGGACACUAUAGUAGCGGUGGUGGCAGCGGGAUCACCACGCCCACUGGCCUGGGGCCGAUGCCGCCACUACCCCCGCCUCCCGU------CGAUGGUAU- (.(((((((((.....))))........(..((((.(((((..((((.(((.(((.(((....))).)))....))).))))..))))).))))..)..).------)))).)...- ( -41.00) >DroPer_CAF1 48211 105 - 1 CGAUCCCCCAAGCUCUUUGGCCACUACAGCA---G---UGGCAGCGGCAUAACCACACCCACUGGUCUGGGCCAGAUGCCACCGCUGCCCCCACCCCCGGU------CAUGGGUACC ...........((((..((((((((.....)---)---)(((((((((((..(((.(((....))).))).....)))))...)))))).........)))------)).))))... ( -34.90) >consensus CGAUCGCCCAAACUAUUUGGACACUAUAGCA___G___UGGCAGCGGCAUAACCACACCCACUGGUCUGGGACAGAUGCCACCACUGCCCCCGCCCCCCGU______CAUGGGUAC_ ...........(((((.........))))).........((((((((..........((((......))))..........))))))))...(((................)))... (-18.84 = -19.01 + 0.17)
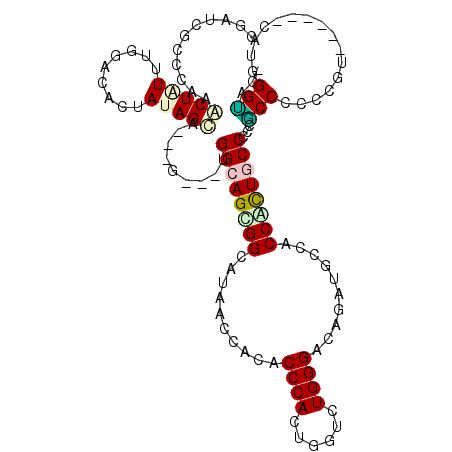
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:42:38 2006