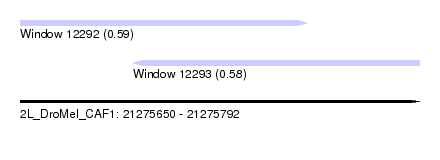
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 21,275,650 – 21,275,792 |
| Length | 142 |
| Max. P | 0.588858 |

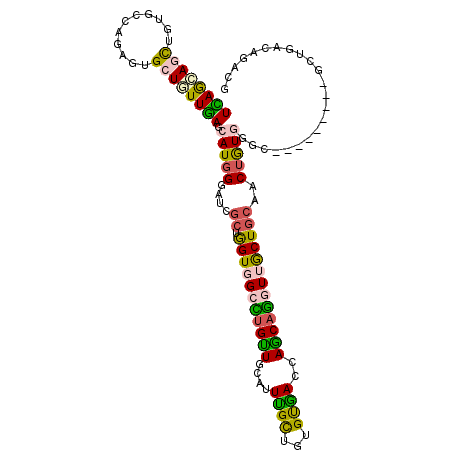
| Location | 21,275,650 – 21,275,752 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 75.81 |
| Mean single sequence MFE | -39.93 |
| Consensus MFE | -19.18 |
| Energy contribution | -18.80 |
| Covariance contribution | -0.38 |
| Combinations/Pair | 1.53 |
| Mean z-score | -1.83 |
| Structure conservation index | 0.48 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.588858 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
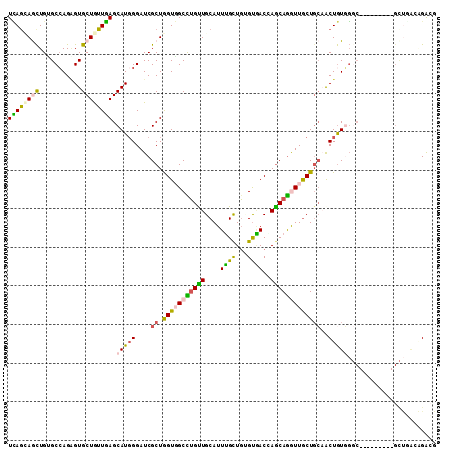
>2L_DroMel_CAF1 21275650 102 + 22407834 UGAGCAGUUGAGCCAGGGUGCUGUUUAGCAUGGGAUCGCUGGUUGCCUGCUGCAUUUGCUGAGUGACCAGCAGGUUGCUGCAACUAUGUGC---------GCCUAAUGAAG ((((((((...((....)))))))))).((((((..((((((((((..((...((((((((......)))))))).)).))))))).))).---------.))).)))... ( -35.80) >DroVir_CAF1 8618 111 + 1 UUAGCAGCUGGGCGAGCGUGCUGUUGAGCAUGGGAUCGCUGGUUGCCUGUUGCAUUUGUUGGGUGACAAGCAGGUUGCUGCUGCUGUUGCCGUGUCCAUUGUUGACGGGCG .(((((((..(((((.((((((....))))))...)))))(((.(((((((.((((.....))))...))))))).))))))))))..(((.((((.......))))))). ( -46.90) >DroPse_CAF1 1249 102 + 1 UCAGGAGCUGUGCCAGAGUGCUGUUGAGCAUGGGAUCGCUGGUGGCUUGUUGCAUUUGCUGUGUAACGAGCAGAUUGCUGCAACUGUGGGC---------GCUGGCAGGCG ......(((.((((((.((.(.(((((((((.((....)).)).(((((((((((.....))))))))))).....))).))))...).))---------.))))))))). ( -41.20) >DroWil_CAF1 3839 93 + 1 UCAGUAACUGUGCCAGGGUGCUAUUGAGCAUCGGAUCGCUAGUGGCCUGUUGCAUUUGCUGCGUGACCAACAGGUUGCUCACACUGUUG------------------GAAG .((((((.(((..((((.(((((.(((........))).))))).))))..))).)))))).....(((((((((.....)).))))))------------------)... ( -33.80) >DroAna_CAF1 3042 102 + 1 UCAACAACUGGGCCAGAGUGCUGUUGAGCAUGGGAUCGCUGGUGGCUGGCUGCAUUGGCUCUGCCACCAGCAAGUUACUGCAACUGUGGGC---------GCUCAGGGACG .......(((((((...(((((....)))))......(((((((((.((((.....))))..)))))))))......(..(....)..)).---------))))))..... ( -40.70) >DroPer_CAF1 1946 102 + 1 UCAGGAGCUGUGCCAGAGUGCUGUUGAGCAUGGGAUCGCUGGUGGCUUGUUGCAUUUGCUGUGUAACGAGCAGAUUGCUGCAACUGUGGGC---------GCUGGCAGGCG ......(((.((((((.((.(.(((((((((.((....)).)).(((((((((((.....))))))))))).....))).))))...).))---------.))))))))). ( -41.20) >consensus UCAGCAGCUGUGCCAGAGUGCUGUUGAGCAUGGGAUCGCUGGUGGCCUGUUGCAUUUGCUGUGUGACCAGCAGGUUGCUGCAACUGUGGGC_________GCUGACAGACG ((((((((...........)))))))).(((((....((.(((((((((((....((((...))))..)))))))))))))..)))))....................... (-19.18 = -18.80 + -0.38)

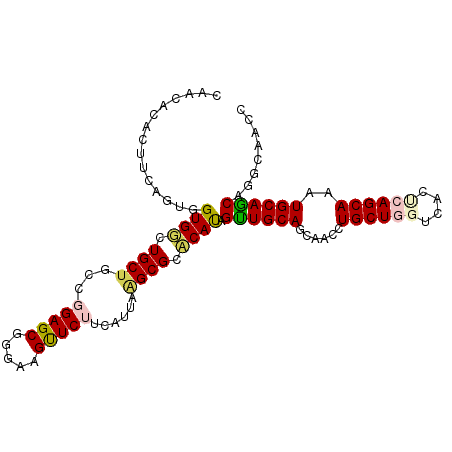
| Location | 21,275,690 – 21,275,792 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | reverse |
| Mean pairwise identity | 82.87 |
| Mean single sequence MFE | -35.61 |
| Consensus MFE | -24.95 |
| Energy contribution | -24.95 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.28 |
| Mean z-score | -1.69 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.581425 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 21275690 102 - 22407834 CAACACACUUCAGUGGUGUCUGCUGCUGGAGCGGGAAGUUCUUCAUUAGGCGCACAUAGUUGCAGCAACCUGCUGGUCACUCAGCAAAUGCAGCAGGCAACC .....(((....))).((((((((((.(((((.....))))).......(((((......))).))....((((((....))))))...))))))))))... ( -34.50) >DroPse_CAF1 1289 96 - 1 CAG------UCGGUGGUGGCUGCUGCCGGAGCAGGUAGCUCGCCUGCCAGCGCCCACAGUUGCAGCAAUCUGCUCGUUACACAGCAAAUGCAACAAGCCACC ...------..(((((((((.((((.....((((((.....)))))))))))))....((((((......((((.(.....)))))..))))))..)))))) ( -37.60) >DroSim_CAF1 1788 102 - 1 CAACACACUUCAGUGGUGUCUGCUGCUGGAGCUGGAAGUUCUUCAUUAAGCGCACAUAGUUGCAGCAACCUGCUGGUCACUCAGCAAAUGCAGCAGGCAACC .....(((....))).((((((((((.((.(((.(((....)))....)))(((......))).....))((((((....))))))...))))))))))... ( -35.20) >DroEre_CAF1 1570 102 - 1 CAACACACUUCUGUGGUGUCUGCAGCCGGAGCGGGAAGUUCUUCAUUAAGCGCACAUAGCUGCAGCAACCUGCUGGUCACCCAGCAAAUGCAGCAGGCAACC ...((((....)))).(((((((.((.(((((.....)))))......(((.......))))).(((...((((((....))))))..))).)))))))... ( -34.30) >DroYak_CAF1 1796 102 - 1 CAACACACUUCCGUGGUGGCUGCUGCCGGAGCGGGAAGUUCUUCAUUAAGCGCACAUAGUUGCAGCAGCCUGCUGGUCUCUCAGCAAAUGCAGCAGGCAACC ......((((((((..((((....))))..)).))))))..........(((((......))).)).((((((((...............)))))))).... ( -34.46) >DroPer_CAF1 1986 96 - 1 CAG------UCGGUGGUGGCUGCUGCCGGAGCAGGUAGCUCGCCUGCCAGCGCCCACAGUUGCAGCAAUCUGCUCGUUACACAGCAAAUGCAACAAGCCACC ...------..(((((((((.((((.....((((((.....)))))))))))))....((((((......((((.(.....)))))..))))))..)))))) ( -37.60) >consensus CAACACACUUCAGUGGUGGCUGCUGCCGGAGCGGGAAGUUCUUCAUUAAGCGCACAUAGUUGCAGCAACCUGCUGGUCACUCAGCAAAUGCAGCAGGCAACC ...............((((.((((...(((((.....)))))......)))).)))).((((((......((((((....))))))..))))))........ (-24.95 = -24.95 + 0.00)

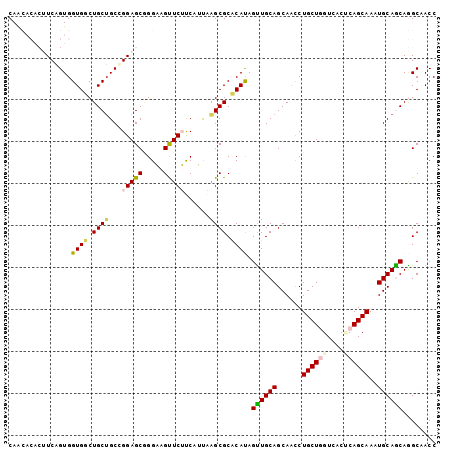
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:41:56 2006