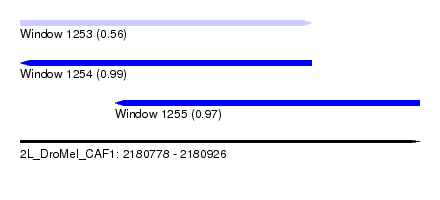
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 2,180,778 – 2,180,926 |
| Length | 148 |
| Max. P | 0.985913 |

| Location | 2,180,778 – 2,180,886 |
|---|---|
| Length | 108 |
| Sequences | 4 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 84.46 |
| Mean single sequence MFE | -23.77 |
| Consensus MFE | -14.88 |
| Energy contribution | -15.12 |
| Covariance contribution | 0.25 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.563307 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2180778 108 + 22407834 GCAGCCAAGUGGCAUCCACUUGUGCUCUAGAAUCAGACACCAAAACACAGAUAUGGCCGCACAUAUGUAAGCACGGCACACAUGG------UCACAUACAUAUGUAUGUGUACA ...(((((((((...))))))(((((((......)))...........(.(((((......))))).)..))))))).......(------((((((((....))))))).)). ( -30.30) >DroSec_CAF1 26853 100 + 1 GCAGCCAAGUGGCAUCCACUUGUGCUCUAGAAUCAGACACCAAAACGCAGAUAUGGCCGCACAU----AAGCACGGCACACAUGG------UCACAUACUA----AUAUGUACA ((.(((((((((...))))))).))(((......))).........)).(((.((((((..(..----..)..)))).))....)------))(((((...----.)))))... ( -22.70) >DroSim_CAF1 21407 100 + 1 GCAGCCAAGUGGCAUCCACUUGUGCUCUAGAAUCAGACACCAAAACACAGAUAUGGCCGCACAU----AAGCACGGCACACAUGG------UCACAUACUA----AUAUGUACA ..((((((((((...))))))).))).........(((.........((....))((((..(..----..)..)))).......)------))(((((...----.)))))... ( -22.30) >DroYak_CAF1 25573 110 + 1 GCAGCCAAGUGGCAUCCACUUGUGCUCUAGAAUCAGACACCAAAACACAGAUAUGCCCGCACAUAUGUAAGCACACCACACACACCAUACACCACACAUGG----GCACCUACA ..((((((((((...))))))).)))...........................((((((......((((..................)))).......)))----)))...... ( -19.79) >consensus GCAGCCAAGUGGCAUCCACUUGUGCUCUAGAAUCAGACACCAAAACACAGAUAUGGCCGCACAU____AAGCACGGCACACAUGG______UCACAUACUA____AUAUGUACA ((.(((((((((...))))))(((.(((......))))))..............))).))...................................................... (-14.88 = -15.12 + 0.25)

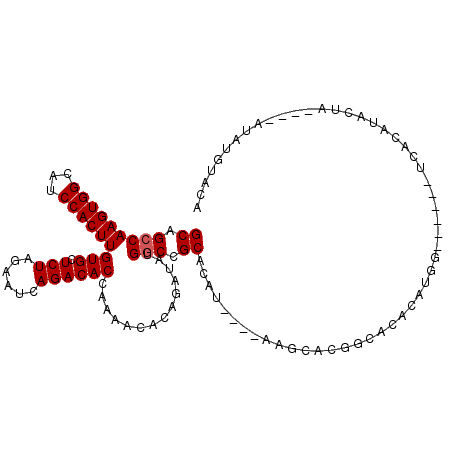
| Location | 2,180,778 – 2,180,886 |
|---|---|
| Length | 108 |
| Sequences | 4 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 84.46 |
| Mean single sequence MFE | -37.00 |
| Consensus MFE | -27.92 |
| Energy contribution | -27.18 |
| Covariance contribution | -0.75 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.78 |
| Structure conservation index | 0.75 |
| SVM decision value | 2.02 |
| SVM RNA-class probability | 0.985913 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2180778 108 - 22407834 UGUACACAUACAUAUGUAUGUGA------CCAUGUGUGCCGUGCUUACAUAUGUGCGGCCAUAUCUGUGUUUUGGUGUCUGAUUCUAGAGCACAAGUGGAUGCCACUUGGCUGC ....(((((((....))))))).------(((((((.((((..(........)..)))))))...((((((((((.........)))))))))).))))..(((....)))... ( -41.00) >DroSec_CAF1 26853 100 - 1 UGUACAUAU----UAGUAUGUGA------CCAUGUGUGCCGUGCUU----AUGUGCGGCCAUAUCUGCGUUUUGGUGUCUGAUUCUAGAGCACAAGUGGAUGCCACUUGGCUGC ..((((((.----...)))))).------..(((((.((((..(..----..)..)))))))))..(((((((((.........))))))).(((((((...)))))))))... ( -34.30) >DroSim_CAF1 21407 100 - 1 UGUACAUAU----UAGUAUGUGA------CCAUGUGUGCCGUGCUU----AUGUGCGGCCAUAUCUGUGUUUUGGUGUCUGAUUCUAGAGCACAAGUGGAUGCCACUUGGCUGC ..((((((.----...)))))).------(((((((.((((..(..----..)..)))))))...((((((((((.........)))))))))).))))..(((....)))... ( -35.40) >DroYak_CAF1 25573 110 - 1 UGUAGGUGC----CCAUGUGUGGUGUAUGGUGUGUGUGGUGUGCUUACAUAUGUGCGGGCAUAUCUGUGUUUUGGUGUCUGAUUCUAGAGCACAAGUGGAUGCCACUUGGCUGC ((((((..(----(((((..((.(....).))..))))).)..)))))).....((((.((....((((((((((.........))))))))))(((((...))))))).)))) ( -37.30) >consensus UGUACAUAU____UAGUAUGUGA______CCAUGUGUGCCGUGCUU____AUGUGCGGCCAUAUCUGUGUUUUGGUGUCUGAUUCUAGAGCACAAGUGGAUGCCACUUGGCUGC ....(((((........))))).........(((((.((((..(........)..)))))))))..(((((((((.........))))))).(((((((...)))))))))... (-27.92 = -27.18 + -0.75)

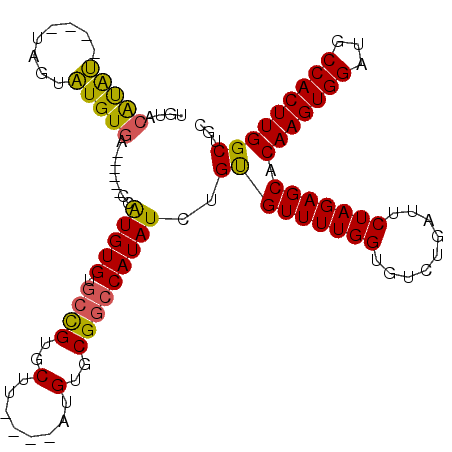
| Location | 2,180,813 – 2,180,926 |
|---|---|
| Length | 113 |
| Sequences | 4 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 81.62 |
| Mean single sequence MFE | -37.62 |
| Consensus MFE | -25.74 |
| Energy contribution | -26.30 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.47 |
| Structure conservation index | 0.68 |
| SVM decision value | 1.70 |
| SVM RNA-class probability | 0.972972 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2180813 113 - 22407834 UAUCAAAUCGCUGGCUGUCUGGCGGACCUCUGCUACUAUAUGUACACAUACAUAUGUAUGUGA------CCAUGUGUGCCGUGCUUACAUAUGUGCGGCCAUAUCUGUGUUUUGGUGUC (((((((.(((.((.....((((((....)))))).........(((((((....))))))).------))(((((.((((..(........)..)))))))))..))).))))))).. ( -39.50) >DroSec_CAF1 26888 105 - 1 UAUCAAAUCGCUGGCUGUCUGGCGGACCUCUGCUGCUAUGUGUACAUAU----UAGUAUGUGA------CCAUGUGUGCCGUGCUU----AUGUGCGGCCAUAUCUGCGUUUUGGUGUC (((((((.(((((((.....(((((....)))))))))((..((((((.----...)))))).------.))((((.((((..(..----..)..))))))))...))).))))))).. ( -35.90) >DroSim_CAF1 21442 105 - 1 UAUCAAAUCGCUGGCUGUCUGGCGGACCUCUGCUGCUAUAUGUACAUAU----UAGUAUGUGA------CCAUGUGUGCCGUGCUU----AUGUGCGGCCAUAUCUGUGUUUUGGUGUC (((((((.(((((((.....(((((....)))))))))(((((((....----..))))))).------..(((((.((((..(..----..)..)))))))))..))).))))))).. ( -33.80) >DroYak_CAF1 25608 115 - 1 UAUCAAAUCGCUGGCUGUCUGCCGUUCCUCCGACGCUAUAUGUAGGUGC----CCAUGUGUGGUGUAUGGUGUGUGUGGUGUGCUUACAUAUGUGCGGGCAUAUCUGUGUUUUGGUGUC (((((((.(((.(((.....))).....((((.(((.(((((((((..(----(((((..((.(....).))..))))).)..)))))))))))))))).......))).))))))).. ( -41.30) >consensus UAUCAAAUCGCUGGCUGUCUGGCGGACCUCUGCUGCUAUAUGUACAUAU____UAGUAUGUGA______CCAUGUGUGCCGUGCUU____AUGUGCGGCCAUAUCUGUGUUUUGGUGUC (((((((.(((((((.....(((((....)))))))))((..((((........))))..)).........(((((.((((..(........)..)))))))))..))).))))))).. (-25.74 = -26.30 + 0.56)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:45:03 2006