
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 20,923,730 – 20,923,889 |
| Length | 159 |
| Max. P | 0.999868 |

| Location | 20,923,730 – 20,923,850 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.25 |
| Mean single sequence MFE | -43.38 |
| Consensus MFE | -37.78 |
| Energy contribution | -38.70 |
| Covariance contribution | 0.92 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.87 |
| SVM decision value | 1.52 |
| SVM RNA-class probability | 0.961298 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20923730 120 + 22407834 CCAUGUUUUUGCCUUAUCAGCGGACGAGGACGCAGCGUGAGUGCGGCUGUCACUUGGGGCAAACAUAUGUUUCGCUACAAAUUGACGACCGCAACGCUCUUAUCAGCAGCGGCUGCUGAU ....(((((((((........)).)))))))(.(((((...(((((.(((((.(((..((((((....)))).))..)))..))))).)))))))))))..((((((((...)))))))) ( -44.00) >DroSec_CAF1 8935 120 + 1 CCAUGUUUUUGCCUUAUCAGCGGACGAGGACGCAGCGUGAGUGCGGCUGUCACUUGGGGCAAACAUAUGUUUCGCUACAAAUUGACGACAGCAACGCUCUUAUCAGCAGCGGCUGCUGAU ..(((((....(((..((....))..)))....)))))(((((..((((((..(((..((((((....)))).))..)))......))))))..)))))..((((((((...)))))))) ( -41.70) >DroSim_CAF1 8262 120 + 1 CCAUGUUUUUGCCUUAUCAGCGGACGAGGACGCAGCGUGAGUGCGGCUGUCACUUGGGGCAAACAUAUGUUUCGCUACAAAUUGACGACCGCAACGCUCUUAUCAGCAGCGGCUGCUGAU ....(((((((((........)).)))))))(.(((((...(((((.(((((.(((..((((((....)))).))..)))..))))).)))))))))))..((((((((...)))))))) ( -44.00) >DroEre_CAF1 7845 119 + 1 CCAUCUUUUGGCCUUAUCAGCCGACGAGGACGCAGCGUGAGUGCGGCUGUCACUUGGGGCAAACAUAUGUUUCGCUACAAAUUGACGACCGCAAGGCGCUUAUCAGCAGC-GCUGCUGAU ...(((((((((.......))))).))))..((((((((..(((((.(((((.(((..((((((....)))).))..)))..))))).)))))...)(((....))).))-))))).... ( -44.00) >DroYak_CAF1 8233 118 + 1 CCAUGUUUUCGCCUUAUCAGCGGACGAGAACGCAGCGUGAGUGCGGCUGUCACUUGGGGCAAACAUAUGUUUCGCUACAAAUUGACGACCGCAACGCUCUUAUCAGCCG--GCUGCUGAU ....(((((((((........)).)))))))(.(((((...(((((.(((((.(((..((((((....)))).))..)))..))))).)))))))))))..((((((..--...)))))) ( -43.20) >consensus CCAUGUUUUUGCCUUAUCAGCGGACGAGGACGCAGCGUGAGUGCGGCUGUCACUUGGGGCAAACAUAUGUUUCGCUACAAAUUGACGACCGCAACGCUCUUAUCAGCAGCGGCUGCUGAU ...((((((((((........)).))))))))..((((...(((((.(((((.(((..((((((....)))).))..)))..))))).)))))))))....((((((((...)))))))) (-37.78 = -38.70 + 0.92)

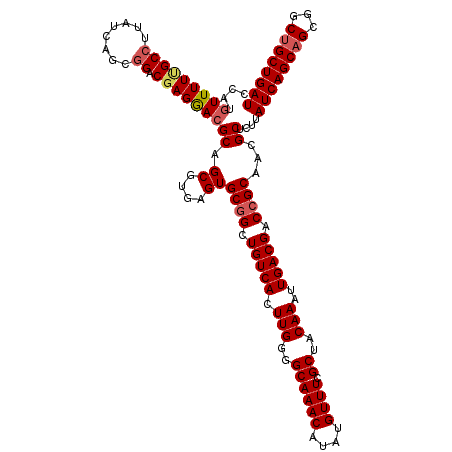
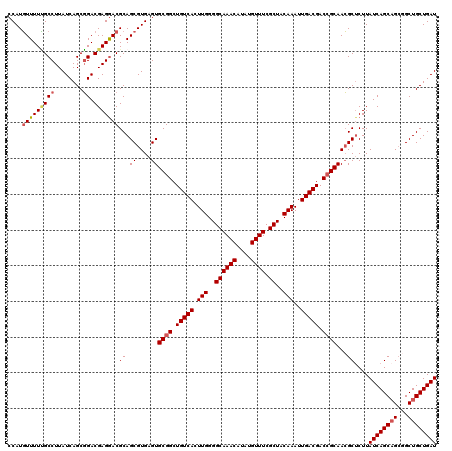
| Location | 20,923,730 – 20,923,850 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.25 |
| Mean single sequence MFE | -42.80 |
| Consensus MFE | -38.56 |
| Energy contribution | -39.20 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.08 |
| Structure conservation index | 0.90 |
| SVM decision value | 2.67 |
| SVM RNA-class probability | 0.996232 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20923730 120 - 22407834 AUCAGCAGCCGCUGCUGAUAAGAGCGUUGCGGUCGUCAAUUUGUAGCGAAACAUAUGUUUGCCCCAAGUGACAGCCGCACUCACGCUGCGUCCUCGUCCGCUGAUAAGGCAAAAACAUGG ((((((((...))))))))...(((((((((((.((((.((((..((.((((....))))))..)))))))).))))))...)))))..(.(((..((....))..)))).......... ( -43.40) >DroSec_CAF1 8935 120 - 1 AUCAGCAGCCGCUGCUGAUAAGAGCGUUGCUGUCGUCAAUUUGUAGCGAAACAUAUGUUUGCCCCAAGUGACAGCCGCACUCACGCUGCGUCCUCGUCCGCUGAUAAGGCAAAAACAUGG ((((((((...))))))))..(((.((.((((((....(((((..((.((((....))))))..))))))))))).)).)))..(((..(((..(....)..)))..))).......... ( -37.80) >DroSim_CAF1 8262 120 - 1 AUCAGCAGCCGCUGCUGAUAAGAGCGUUGCGGUCGUCAAUUUGUAGCGAAACAUAUGUUUGCCCCAAGUGACAGCCGCACUCACGCUGCGUCCUCGUCCGCUGAUAAGGCAAAAACAUGG ((((((((...))))))))...(((((((((((.((((.((((..((.((((....))))))..)))))))).))))))...)))))..(.(((..((....))..)))).......... ( -43.40) >DroEre_CAF1 7845 119 - 1 AUCAGCAGC-GCUGCUGAUAAGCGCCUUGCGGUCGUCAAUUUGUAGCGAAACAUAUGUUUGCCCCAAGUGACAGCCGCACUCACGCUGCGUCCUCGUCGGCUGAUAAGGCCAAAAGAUGG (((((((..-..)))))))..(.((((((((((.((((.((((..((.((((....))))))..)))))))).)))))..(((.((((((....)).))))))).))))))......... ( -45.70) >DroYak_CAF1 8233 118 - 1 AUCAGCAGC--CGGCUGAUAAGAGCGUUGCGGUCGUCAAUUUGUAGCGAAACAUAUGUUUGCCCCAAGUGACAGCCGCACUCACGCUGCGUUCUCGUCCGCUGAUAAGGCGAAAACAUGG ((((((...--..))))))...(((((((((((.((((.((((..((.((((....))))))..)))))))).))))))...)))))..(((.(((.((........))))).))).... ( -43.70) >consensus AUCAGCAGCCGCUGCUGAUAAGAGCGUUGCGGUCGUCAAUUUGUAGCGAAACAUAUGUUUGCCCCAAGUGACAGCCGCACUCACGCUGCGUCCUCGUCCGCUGAUAAGGCAAAAACAUGG ((((((((...))))))))...(((((((((((.((((.((((..((.((((....))))))..)))))))).))))))...)))))..(.(((..((....))..)))).......... (-38.56 = -39.20 + 0.64)

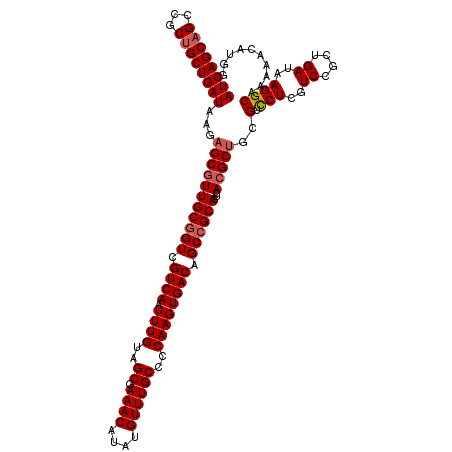
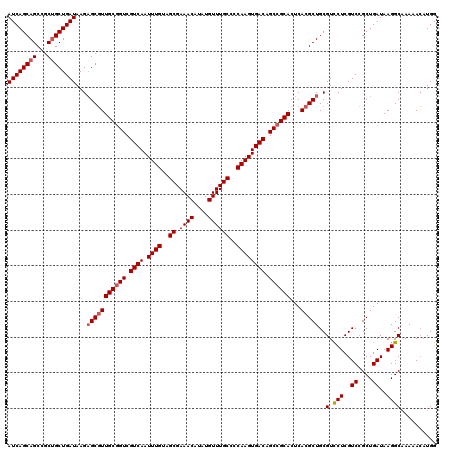
| Location | 20,923,770 – 20,923,889 |
|---|---|
| Length | 119 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.91 |
| Mean single sequence MFE | -39.12 |
| Consensus MFE | -36.34 |
| Energy contribution | -36.94 |
| Covariance contribution | 0.60 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.53 |
| Structure conservation index | 0.93 |
| SVM decision value | 4.31 |
| SVM RNA-class probability | 0.999868 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20923770 119 - 22407834 CCCCCAGUC-CCCCACUUAAAGUCUCUUGCUGUUGUUGUUAUCAGCAGCCGCUGCUGAUAAGAGCGUUGCGGUCGUCAAUUUGUAGCGAAACAUAUGUUUGCCCCAAGUGACAGCCGCAC ....((((.-....((.....)).....))))..(((.((((((((((...)))))))))).)))..((((((.((((.((((..((.((((....))))))..)))))))).)))))). ( -41.60) >DroSec_CAF1 8975 120 - 1 CCCCCAUUCAUCCCACUUAAAGUCCCUUGCUGUUGUUGUUAUCAGCAGCCGCUGCUGAUAAGAGCGUUGCUGUCGUCAAUUUGUAGCGAAACAUAUGUUUGCCCCAAGUGACAGCCGCAC ..................................(((.((((((((((...)))))))))).)))((.((((((....(((((..((.((((....))))))..))))))))))).)).. ( -35.40) >DroSim_CAF1 8302 120 - 1 CCCCCAUUCACCCCACUUAAAGUCACUUGCUGUUGUUGUUAUCAGCAGCCGCUGCUGAUAAGAGCGUUGCGGUCGUCAAUUUGUAGCGAAACAUAUGUUUGCCCCAAGUGACAGCCGCAC ..................................(((.((((((((((...)))))))))).)))..((((((.((((.((((..((.((((....))))))..)))))))).)))))). ( -39.60) >DroEre_CAF1 7885 119 - 1 CCCCCAUUCAACCACAUUAAGGUCCCUGGCUGCAGUUGUUAUCAGCAGC-GCUGCUGAUAAGCGCCUUGCGGUCGUCAAUUUGUAGCGAAACAUAUGUUUGCCCCAAGUGACAGCCGCAC ..........(((.......)))....(((.((.....(((((((((..-..)))))))))))))).((((((.((((.((((..((.((((....))))))..)))))))).)))))). ( -40.00) >DroYak_CAF1 8273 118 - 1 CACCCGUUCAACCACUUUUAAGUCUCUUGCUGUUGUUGUUAUCAGCAGC--CGGCUGAUAAGAGCGUUGCGGUCGUCAAUUUGUAGCGAAACAUAUGUUUGCCCCAAGUGACAGCCGCAC ....((((((((.((...((((...))))..)).))).((((((((...--..))))))))))))).((((((.((((.((((..((.((((....))))))..)))))))).)))))). ( -39.00) >consensus CCCCCAUUCAACCCACUUAAAGUCCCUUGCUGUUGUUGUUAUCAGCAGCCGCUGCUGAUAAGAGCGUUGCGGUCGUCAAUUUGUAGCGAAACAUAUGUUUGCCCCAAGUGACAGCCGCAC ..................................(((.((((((((((...)))))))))).)))..((((((.((((.((((..((.((((....))))))..)))))))).)))))). (-36.34 = -36.94 + 0.60)

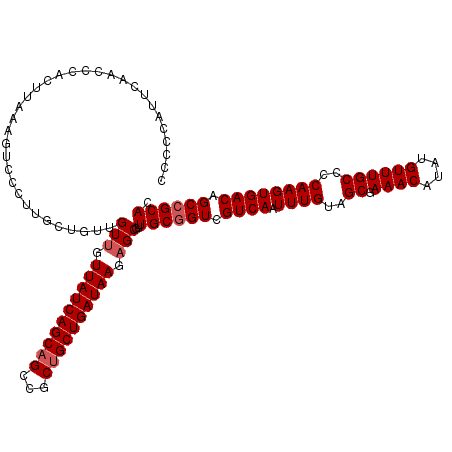
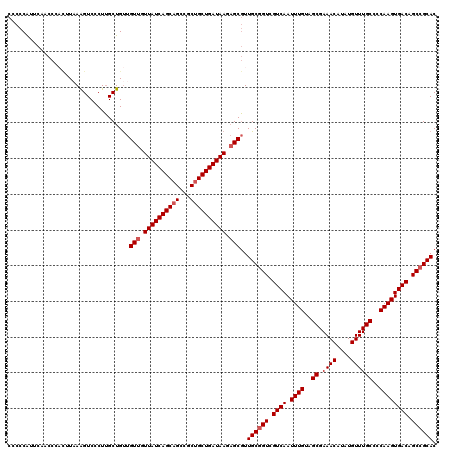
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:39:26 2006