
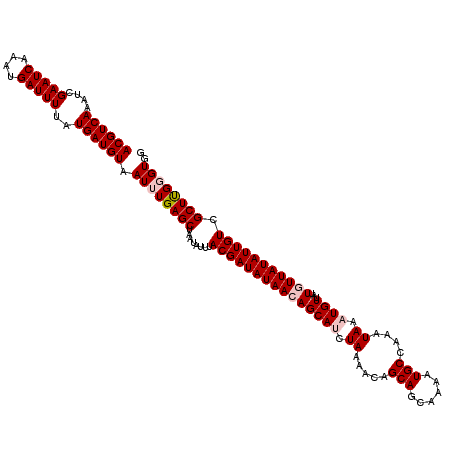
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 20,765,485 – 20,765,604 |
| Length | 119 |
| Max. P | 0.825406 |

| Location | 20,765,485 – 20,765,604 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.22 |
| Mean single sequence MFE | -24.47 |
| Consensus MFE | -22.12 |
| Energy contribution | -22.90 |
| Covariance contribution | 0.78 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.36 |
| Structure conservation index | 0.90 |
| SVM decision value | 0.70 |
| SVM RNA-class probability | 0.825406 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20765485 119 + 22407834 ACGUCAAAUCGAAUCAAAUGAUUUUAUGAUGUAAUUUGAGCUAAUAUUUACGAUAUAAGAGCAGCUAAAACAGCAGCAAAAUGCCAAAUAAAUGUUAUUGUUAUAUUGUCGCUCG-GUGG ((((((....(((((....)))))..))))))...((((((((((((..((((((........(((.....))).((.....))...........)))))).))))))..)))))-)... ( -22.40) >DroVir_CAF1 24323 120 + 1 ACGUCAAAUCGAAUCAAAUGAUUUUAUGAUGUAAUUUGAGCUAAUAUUUACGAUAUAACAGCAUCUAAAACAGCAGCAAAAUGCCAAAUAAAUGUUAUUGUUAUAUUGUCGCUUGGGUGG ((((((....(((((....)))))..)))))).(((..(((........(((((((((((((((.((.....(((......)))....)).))))...))))))))))).)))..))).. ( -25.50) >DroGri_CAF1 19792 120 + 1 ACGUCAAAUCGAAUCAAAUGAUUUUAUGAUGUAAUUUGAGCUAAUAUUUACGAUAUAACAGCAUCUAAAACAGCAGCAAAAUGCCAAAUAAAUGUUAUUGUUAUAUUGUCGCUUGGGUGG ((((((....(((((....)))))..)))))).(((..(((........(((((((((((((((.((.....(((......)))....)).))))...))))))))))).)))..))).. ( -25.50) >DroEre_CAF1 16677 119 + 1 ACGUCAAAUCGAAUCAAAUGAUUUUAUGAUGUAAUUUGAGCUAAUAUUUACGAUAUAAGAGCAGCUAAAACAGCAGCAAAAUGCCAAAUAAAUGUUAUUGUUAUAUUGUCGCUCG-GUGG ((((((....(((((....)))))..))))))...((((((((((((..((((((........(((.....))).((.....))...........)))))).))))))..)))))-)... ( -22.40) >DroWil_CAF1 25920 120 + 1 ACGUCAAAUCGAAUCAAAUGAUUUUAUGAUGUAAUUUGAGCUAAUAUUUACGAUAUAACAGCAUCUAAAACAGCAGCAAAAUGCCAAAUAAAUGUUAUUGUUAUAUUGUCGCUUGGGUGG ((((((....(((((....)))))..)))))).(((..(((........(((((((((((((((.((.....(((......)))....)).))))...))))))))))).)))..))).. ( -25.50) >DroMoj_CAF1 20576 120 + 1 ACGUCAAAUCGAAUCAAAUGAUUUUAUGAUGUAAUUUGAGCUAAUAUUUACGAUAUAACAGCAUCUAAAACAGCAGCAAAAUGCCAAAUAAAUGUUAUUGUUAUAUUGUCGCUUGGGUGG ((((((....(((((....)))))..)))))).(((..(((........(((((((((((((((.((.....(((......)))....)).))))...))))))))))).)))..))).. ( -25.50) >consensus ACGUCAAAUCGAAUCAAAUGAUUUUAUGAUGUAAUUUGAGCUAAUAUUUACGAUAUAACAGCAUCUAAAACAGCAGCAAAAUGCCAAAUAAAUGUUAUUGUUAUAUUGUCGCUUGGGUGG ((((((....(((((....)))))..)))))).((((((((........(((((((((((((((.((.....(((......)))....)).))))...))))))))))).)))))))).. (-22.12 = -22.90 + 0.78)


| Location | 20,765,485 – 20,765,604 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.22 |
| Mean single sequence MFE | -20.23 |
| Consensus MFE | -19.00 |
| Energy contribution | -19.67 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.00 |
| Mean z-score | -0.95 |
| Structure conservation index | 0.94 |
| SVM decision value | 0.29 |
| SVM RNA-class probability | 0.668800 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20765485 119 - 22407834 CCAC-CGAGCGACAAUAUAACAAUAACAUUUAUUUGGCAUUUUGCUGCUGUUUUAGCUGCUCUUAUAUCGUAAAUAUUAGCUCAAAUUACAUCAUAAAAUCAUUUGAUUCGAUUUGACGU ....-.(((((........................(((.....)))((((...))))))))).................(((((((((..((((..........))))..))))))).)) ( -16.90) >DroVir_CAF1 24323 120 - 1 CCACCCAAGCGACAAUAUAACAAUAACAUUUAUUUGGCAUUUUGCUGCUGUUUUAGAUGCUGUUAUAUCGUAAAUAUUAGCUCAAAUUACAUCAUAAAAUCAUUUGAUUCGAUUUGACGU ........(((((.((((((((....((((((..(((((......)))))...)))))).)))))))).))..........(((((((..((((..........))))..)))))))))) ( -21.90) >DroGri_CAF1 19792 120 - 1 CCACCCAAGCGACAAUAUAACAAUAACAUUUAUUUGGCAUUUUGCUGCUGUUUUAGAUGCUGUUAUAUCGUAAAUAUUAGCUCAAAUUACAUCAUAAAAUCAUUUGAUUCGAUUUGACGU ........(((((.((((((((....((((((..(((((......)))))...)))))).)))))))).))..........(((((((..((((..........))))..)))))))))) ( -21.90) >DroEre_CAF1 16677 119 - 1 CCAC-CGAGCGACAAUAUAACAAUAACAUUUAUUUGGCAUUUUGCUGCUGUUUUAGCUGCUCUUAUAUCGUAAAUAUUAGCUCAAAUUACAUCAUAAAAUCAUUUGAUUCGAUUUGACGU ....-.(((((........................(((.....)))((((...))))))))).................(((((((((..((((..........))))..))))))).)) ( -16.90) >DroWil_CAF1 25920 120 - 1 CCACCCAAGCGACAAUAUAACAAUAACAUUUAUUUGGCAUUUUGCUGCUGUUUUAGAUGCUGUUAUAUCGUAAAUAUUAGCUCAAAUUACAUCAUAAAAUCAUUUGAUUCGAUUUGACGU ........(((((.((((((((....((((((..(((((......)))))...)))))).)))))))).))..........(((((((..((((..........))))..)))))))))) ( -21.90) >DroMoj_CAF1 20576 120 - 1 CCACCCAAGCGACAAUAUAACAAUAACAUUUAUUUGGCAUUUUGCUGCUGUUUUAGAUGCUGUUAUAUCGUAAAUAUUAGCUCAAAUUACAUCAUAAAAUCAUUUGAUUCGAUUUGACGU ........(((((.((((((((....((((((..(((((......)))))...)))))).)))))))).))..........(((((((..((((..........))))..)))))))))) ( -21.90) >consensus CCACCCAAGCGACAAUAUAACAAUAACAUUUAUUUGGCAUUUUGCUGCUGUUUUAGAUGCUGUUAUAUCGUAAAUAUUAGCUCAAAUUACAUCAUAAAAUCAUUUGAUUCGAUUUGACGU ........(((((.((((((((....((((((..(((((......)))))...)))))).)))))))).))..........(((((((..((((..........))))..)))))))))) (-19.00 = -19.67 + 0.67)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:37:44 2006