
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 20,752,336 – 20,752,549 |
| Length | 213 |
| Max. P | 0.997488 |

| Location | 20,752,336 – 20,752,441 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 81.84 |
| Mean single sequence MFE | -47.45 |
| Consensus MFE | -33.88 |
| Energy contribution | -33.72 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.41 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.86 |
| SVM RNA-class probability | 0.868632 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20752336 105 + 22407834 GGAGCACCUCCCCCACCGCCGCCACCUAUGCCGGGACGAGCAGGUGGUGGACCUCCGCCUCCACCACCACCUCCCAUGCCGGGAAGGGCAGGUGGACCGCCACCA ((((...)))).(((((.(((.((....)).))).....((.(((((((((........))))))))).((((((.....))).))))).))))).......... ( -44.00) >DroSec_CAF1 4212 105 + 1 GGAGUACCUCCCCCACCGCCGCCACCUAUGCCGGGCCGAGCAGGUGGUGGACCUCCGCCUCCACCACCACCUCCCAUGCCGGGAAGGGCAGGUGGACCGCCACCU ((((...))))....((((((((.......((.(((.(((..(((((((((........)))))))))..)))....))).))...))).))))).......... ( -44.40) >DroSim_CAF1 4204 105 + 1 GGAGCACCUCCCCCACCGCCGCCACCUAUGCCGGGCCGAGCAGGUGGUGGACCUCCGCCUCCACCACCACCUCCCAUGCCGGGCAGGGCAGGUGGACCGCCACCU ((((...))))....((((((((.....((((.(((.(((..(((((((((........)))))))))..)))....))).)))).))).))))).......... ( -49.70) >DroWil_CAF1 5561 105 + 1 GGAGCACCCCCACCUCCACCGCCACCCAUGCCGGGUCGUGGUCCUGGAGGACCACCCCCGCCGCCACCACCGCCAAUGCCUGGAAAAGCUGGAGGACCUCCACCG ((((........((((((.(.(((..((((.((((..(((((((....))))))).)))).)((.......))..)))..)))....).))))))..)))).... ( -37.40) >DroYak_CAF1 4137 105 + 1 GGGGCUCCGCCCCCACCGCCACCACCUAUGCCGGGCAGAGCAGGUGGUGGACCUCCGCCUCCGCCACCACCUCCCAUGCCCGGAAGGGUAGGUGGACCGCCCCCA (((((..((.......))(((((((((...((((((((((..(((((((((........)))))))))..)))...)))))))..)))).)))))...))))).. ( -57.10) >DroAna_CAF1 3845 104 + 1 -GUCCACCACCUCCGCCGCCGCCACCUAUGCCGGGAAGAGCGGGAGGCGGUCCGCCACCGCCUCCUCCACCUCCAAUGCCUGGUAUGGGCGGCGGACCACCUCCU -.................(((((.(((((((((((..(((.((((((((((.....))))))))))....))).....))))))))))).))))).......... ( -52.10) >consensus GGAGCACCUCCCCCACCGCCGCCACCUAUGCCGGGCCGAGCAGGUGGUGGACCUCCGCCUCCACCACCACCUCCCAUGCCGGGAAGGGCAGGUGGACCGCCACCU ............(((((...(((.......((.(((.(((..(((((((((........)))))))))..)))....))).))...))).))))).......... (-33.88 = -33.72 + -0.16)



| Location | 20,752,370 – 20,752,472 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 79.68 |
| Mean single sequence MFE | -55.27 |
| Consensus MFE | -30.17 |
| Energy contribution | -30.53 |
| Covariance contribution | 0.37 |
| Combinations/Pair | 1.29 |
| Mean z-score | -3.68 |
| Structure conservation index | 0.55 |
| SVM decision value | 2.04 |
| SVM RNA-class probability | 0.986303 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20752370 102 + 22407834 GACGAGCAGGUGGUGGACCUCCGCCUCCACCACCACCUCCCAUGCCGGGAAGGGCAGGUGGACCGCCACCACCUCCUCCACCGCCCGGAAUG------GG---AGGCCCAC ........(((((((((........))))))))).((((((((.(((((..(((.((((((.......)))))))))......))))).)))------))---)))..... ( -55.40) >DroSim_CAF1 4238 102 + 1 GCCGAGCAGGUGGUGGACCUCCGCCUCCACCACCACCUCCCAUGCCGGGCAGGGCAGGUGGACCGCCACCUCCUCCUCCACCGCCCGGAAUG------GG---AGGCCCAC ........(((((((((........))))))))).((((((((.((((((((((.((((((....)))))).).))).....)))))).)))------))---)))..... ( -58.50) >DroEre_CAF1 4057 102 + 1 GCAGAGCAGGUGGUGGACCUCCGCCUCCGCCCCCACCUCCCAUGCCGGGAAGGGCAGGUGGACCGCCCCCACCUCCUCCACCGCCCGGAAUG------GG---AGGCCCAC .....((.((.(((((....))))).)))).....((((((((.(((((..(((.((((((.......)))))))))......))))).)))------))---)))..... ( -52.00) >DroWil_CAF1 5595 105 + 1 GUCGUGGUCCUGGAGGACCACCCCCGCCGCCACCACCGCCAAUGCCUGGAAAAGCUGGAGGACCUCCACCGCCACCUCCUCCGCCGGGAAUG------GGUGGUGGGCCAC ...(((((((....))))))).(((((((((....((((((.....)))....((.((((((..............))))))))))).....------))))))))).... ( -46.94) >DroYak_CAF1 4171 102 + 1 GCAGAGCAGGUGGUGGACCUCCGCCUCCGCCACCACCUCCCAUGCCCGGAAGGGUAGGUGGACCGCCCCCACCUCCUCCACCGCCCGGGAUG------GG---AGGCCCAC ........(((((((((........))))))))).((((((((.(((((..((..((((((.......))))))......))..))))))))------))---)))..... ( -53.70) >DroAna_CAF1 3878 108 + 1 GAAGAGCGGGAGGCGGUCCGCCACCGCCUCCUCCACCUCCAAUGCCUGGUAUGGGCGGCGGACCACCUCCUCCGCCUCCCAUGCCCGGUAUGGGUAGGGG---AGGUCCAC ...((.(.(((((((((.....)))))))))(((.(((((.(((((.((((((((.((((((........)))))).)))))))).))))).)).)))))---)).))... ( -65.10) >consensus GCAGAGCAGGUGGUGGACCUCCGCCUCCGCCACCACCUCCCAUGCCGGGAAGGGCAGGUGGACCGCCACCACCUCCUCCACCGCCCGGAAUG______GG___AGGCCCAC ...(((..(((((((((........)))))))))..))).(((.(((((..(((.(((.((.......)).))).))).....))))).)))................... (-30.17 = -30.53 + 0.37)


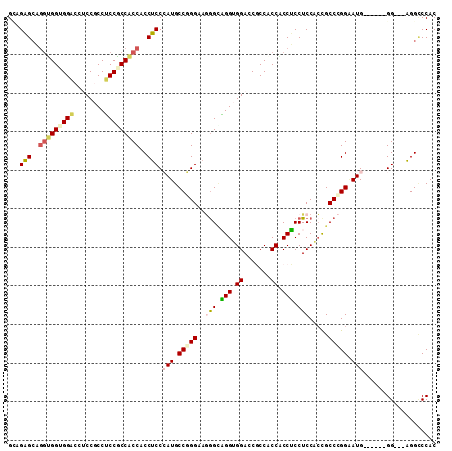
| Location | 20,752,410 – 20,752,509 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 74.24 |
| Mean single sequence MFE | -52.05 |
| Consensus MFE | -27.47 |
| Energy contribution | -29.75 |
| Covariance contribution | 2.28 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.98 |
| Structure conservation index | 0.53 |
| SVM decision value | 2.87 |
| SVM RNA-class probability | 0.997488 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20752410 99 + 22407834 CAUGCCGGGAAGGGCAGG---UGGAC--CGCCACCACCUCCUCCACCGCCCGGAAUG------GG---AGGCCCACCGCCUCCACCCA---UGCCUGGCAUGAUGCGCCCAGGAGG (((((((((..((((.((---((((.--.............))))))))))((....------((---((((.....))))))..)).---..))))))))).............. ( -46.94) >DroPse_CAF1 5343 107 + 1 AAUGCCUGGCAUGGGAGGACCCAGAC--CUCCACCCCCGCCGCCUAUGCCCGGAAUG------GGUGGUGGUCCACCGCCACCGCCCAUGAUGCCUGGCAUGAUGCGACCUGGAG- ....((.(((..(((.(((.......--.))).)))..)))((.((((((.((.(((------(((((((((.....))))))))))))....)).))))))..)).....))..- ( -50.40) >DroGri_CAF1 4207 101 + 1 AAUGCCGGGCAUGGGCGG---UGGUCCA--CCACCGCCACCACCUAUGCCCGGUCGG------GGUGGAGGUCCGCCACCGCCACCAA---UGCCGGGCAUGAUACGGCCAGGCG- ...(((.(((...(((((---(((....--))))))))......((((((((((.((------(((((.((....)).))))).))..---.)))))))))).....))).))).- ( -62.00) >DroWil_CAF1 5635 101 + 1 AAUGCCUGGAAAAGCUGG---AGGAC--CUCCACCGCCACCUCCUCCGCCGGGAAUG------GGUGGUGGGCCACCACCGCCACCGA---UGCCUGGCAUGAUACGACCUGGUG- ...(((.((.......((---((((.--.............))))))((((((.((.------((((((((.......)))))))).)---).)))))).........)).))).- ( -45.74) >DroAna_CAF1 3918 104 + 1 AAUGCCUGGUAUGGGCGG---CGGAC--CACCUCCUCCGCCUCCCAUGCCCGGUAUGGGUAGGGG---AGGUCCACCGCCGCCACCUA---UGCCGGGAAUGAUUCGACCCGGAG- ....(((((((((((.((---(((..--.....(((((.((((((((((...))))))).)))))---))).......))))).))))---))))))).....((((...)))).- ( -56.44) >DroPer_CAF1 5517 107 + 1 AAUGCCUGGCAUGGGAGGACCCAGAC--CUCCACCCCCGCCGCCUAUGCCCGGAAUG------GGUGGUGGUCCACCGCCACCACCCAUGAUGCCUGGCAUGAUGCGACCUGGAG- ....((.(((..(((.(((.......--.))).)))..)))((.((((((.((.(((------(((((((((.....))))))))))))....)).))))))..)).....))..- ( -50.80) >consensus AAUGCCUGGCAUGGGAGG___CGGAC__CUCCACCCCCGCCUCCUAUGCCCGGAAUG______GGUGGAGGUCCACCGCCGCCACCCA___UGCCUGGCAUGAUGCGACCUGGAG_ .(((((.((((((((.((....((.........))....)).)))))))).............(((((((((.....)))))))))..........)))))............... (-27.47 = -29.75 + 2.28)
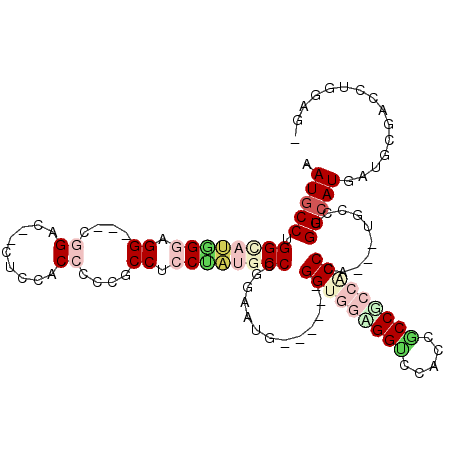
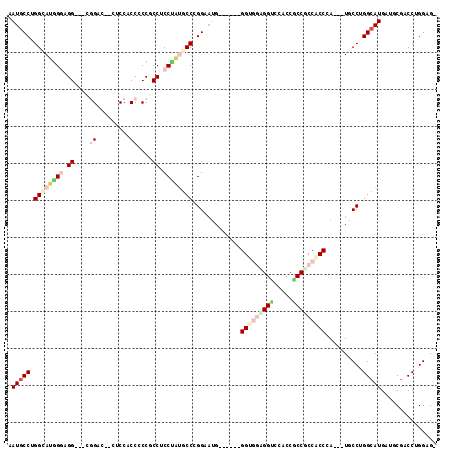
| Location | 20,752,441 – 20,752,549 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 80.06 |
| Mean single sequence MFE | -51.55 |
| Consensus MFE | -25.41 |
| Energy contribution | -27.13 |
| Covariance contribution | 1.73 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.89 |
| Structure conservation index | 0.49 |
| SVM decision value | 1.07 |
| SVM RNA-class probability | 0.909502 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20752441 108 + 22407834 CCUCCUCCACCGCCCGGAAUGGG---AGGCCCACCGCCUCCACCCA---UGCCUGGCAUGAUGCGCCCAGGAGGGGGACCUCCACCACCGCCCAUGAUGAUGGGGCCCAUGGUU ........((((((.((.(((((---((((.....))))...))))---).)).)))...(((.((((.(((((....))))).........(((....))))))).)))))). ( -46.30) >DroPse_CAF1 5377 111 + 1 CCGCCGCCUAUGCCCGGAAUGGGUGGUGGUCCACCGCCACCGCCCAUGAUGCCUGGCAUGAUGCGACCUGGAG---GUCCACCUCCGCCCCCCAUGAUGAUGGGGCCCAUGGUG ....(((.((((((.((.((((((((((((.....))))))))))))....)).))))))..)))(((.((((---(....)))))(..((((((....))))))..)..))). ( -58.80) >DroGri_CAF1 4238 108 + 1 CCACCACCUAUGCCCGGUCGGGGUGGAGGUCCGCCACCGCCACCAA---UGCCGGGCAUGAUACGGCCAGGCG---GUCCACCACCACCGCCCAUGAUGAUGAUGCCGAUGGUA ..((((..((((((((((.(((((((.((....)).))))).))..---.))))))))))...((((..((((---((........))))))(((....)))..)))).)))). ( -52.70) >DroWil_CAF1 5666 108 + 1 CCACCUCCUCCGCCGGGAAUGGGUGGUGGGCCACCACCGCCACCGA---UGCCUGGCAUGAUACGACCUGGUG---GUCCACCGCCACCACCCAUGAUGAUGGGACCAAUGGUU ...........((((((.((.((((((((.......)))))))).)---).)))))).......(((((((((---((.....)))))))(((((....)))))......)))) ( -50.90) >DroMoj_CAF1 5903 105 + 1 CCUCCACCAAUGCCCGGCAUGGGU---GGUCCCCCACCUCCUCCAA---UGCCUGGCAUGAUGCGACCGGGUG---GUCCGCCACCACCGCCCAUGAUGAUGAUGCCCAUGGUG ....((((((((((.(((((((((---((....))))))......)---)))).))))).........(((((---((........)))))))................))))) ( -41.40) >DroPer_CAF1 5551 111 + 1 CCGCCGCCUAUGCCCGGAAUGGGUGGUGGUCCACCGCCACCACCCAUGAUGCCUGGCAUGAUGCGACCUGGAG---GUCCACCUCCGCCCCCCAUGAUGAUGGGGCCCAUGGUG ....(((.((((((.((.((((((((((((.....))))))))))))....)).))))))..)))(((.((((---(....)))))(..((((((....))))))..)..))). ( -59.20) >consensus CCACCACCUAUGCCCGGAAUGGGUGGUGGUCCACCACCACCACCCA___UGCCUGGCAUGAUGCGACCUGGAG___GUCCACCACCACCGCCCAUGAUGAUGGGGCCCAUGGUG ...........(((.((....(((((((((.....))))))))).......)).)))........(((.((((.........))))....(((((....)))))......))). (-25.41 = -27.13 + 1.73)

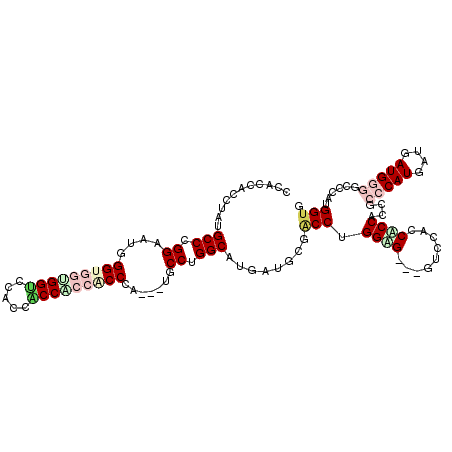
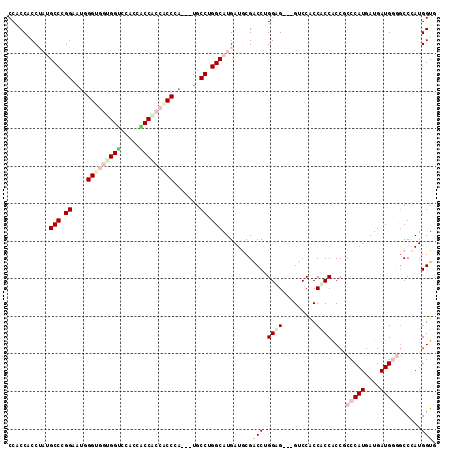
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:37:39 2006