
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 2,123,698 – 2,123,846 |
| Length | 148 |
| Max. P | 0.995152 |

| Location | 2,123,698 – 2,123,806 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.52 |
| Mean single sequence MFE | -33.17 |
| Consensus MFE | -18.65 |
| Energy contribution | -21.07 |
| Covariance contribution | 2.42 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.04 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.70 |
| SVM RNA-class probability | 0.826919 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2123698 108 + 22407834 GGAUUAUGGGAUA-------UAUUGUGUGCAUUUCGAAUGCAGAUAUGCAGU-UAGCCCGCUGGCCAAAUAUUUGCAAUUAGCCCGCGGCU----CGGGGCCAUAAAUUUUUGGCCAGCA .......(((...-------.(((((((((((.....)))))....))))))-...)))((((((((((.(((((......((((.((...----))))))..))))).)))))))))). ( -38.90) >DroSec_CAF1 123994 115 + 1 GGAUUAUGGGAUAUACCAUAUAUUGUGUGCAUUUCGAAUGCAGAUAUGCAGC-UAACCCGCUGGCCAAAUAUUUGCAAUUAGCCCGCGGCU----CGGGGCCAUAAAUUUUUGGCCAGCA ((..(((((......)))))...(((.(((((.....))))).)))......-....))((((((((((.(((((......((((.((...----))))))..))))).)))))))))). ( -40.50) >DroSim_CAF1 123776 110 + 1 GGAUUAUGGGAUAU-----AUAUUGUGUGCAUUUCGAAUGCAGAUACGCAGC-UAGCCCGCUGGCCAAAUAUUUGCAAUUAGCCCGCGGCU----CGGGGCCAUAAAUUUUUGGCCAGCA .......(((....-----....(((.(((((.....))))).)))......-...)))((((((((((.(((((......((((.((...----))))))..))))).)))))))))). ( -38.43) >DroEre_CAF1 126783 107 + 1 GGAUUAUGGGAUAU-------AUAGUGUGCAUAUCGAAUGCAGAUACGCACU-AAACCCACUGGCCAAAUAUUUGCAAUUAGCCCGCGGCU----CG-GGCCAUAAAUUUUUGGCCAGCA ......((((....-------.(((((((..((((.......))))))))))-)..))))(((((((((.(((((......(((((.....----))-)))..))))).))))))))).. ( -37.70) >DroYak_CAF1 129057 111 + 1 GGAUUAUUGGAUAUA----AUAUUGUGUGCAUUUCGAAUGCAGAUACGCAGU-AAGCCCACUGGCCAAAUAUUUGCAAUUAGCCCGCGACU----GGGGGCCAUAAAUUUUUGGCCAGCA ((.((((((((((..----.))))((.(((((.....))))).))...))))-))..)).(((((((((.(((((......((((.(....----).))))..))))).))))))))).. ( -34.10) >DroAna_CAF1 130691 94 + 1 GGAUUAUUGCAC--------------GUGCAUGUUCAAU------AUGAUGUUAAAUGUACUAGGUAAAUAUUUACAAUUAGCUGACAAAUGAGAAGGGGAUAUACAUUUUUGG------ ......((((..--------------((((((...((..------....))....))))))...))))(((((..(..(((.........)))...)..)))))..........------ ( -9.40) >consensus GGAUUAUGGGAUAU______UAUUGUGUGCAUUUCGAAUGCAGAUACGCAGU_AAACCCACUGGCCAAAUAUUUGCAAUUAGCCCGCGGCU____CGGGGCCAUAAAUUUUUGGCCAGCA ......((((.............(((.(((((.....))))).)))..........))))(((((((((.(((((......((((............))))..))))).))))))))).. (-18.65 = -21.07 + 2.42)

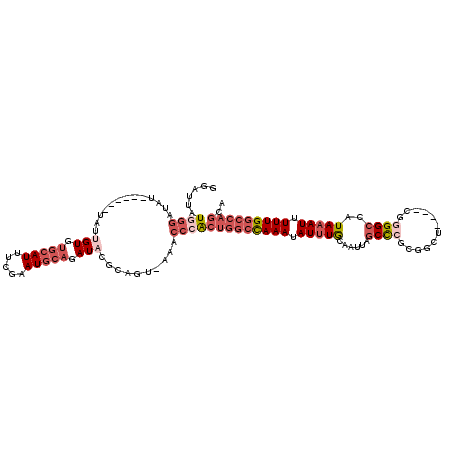
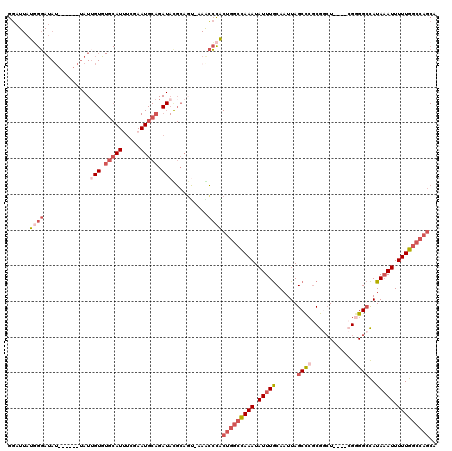
| Location | 2,123,698 – 2,123,806 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.52 |
| Mean single sequence MFE | -32.43 |
| Consensus MFE | -19.85 |
| Energy contribution | -21.97 |
| Covariance contribution | 2.11 |
| Combinations/Pair | 1.11 |
| Mean z-score | -3.05 |
| Structure conservation index | 0.61 |
| SVM decision value | 2.02 |
| SVM RNA-class probability | 0.985749 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2123698 108 - 22407834 UGCUGGCCAAAAAUUUAUGGCCCCG----AGCCGCGGGCUAAUUGCAAAUAUUUGGCCAGCGGGCUA-ACUGCAUAUCUGCAUUCGAAAUGCACACAAUA-------UAUCCCAUAAUCC .((((((((((.((((.((((((((----...)).)))))).....)))).))))))))))(((.((-..(((((.((.......)).))))).......-------)).)))....... ( -35.70) >DroSec_CAF1 123994 115 - 1 UGCUGGCCAAAAAUUUAUGGCCCCG----AGCCGCGGGCUAAUUGCAAAUAUUUGGCCAGCGGGUUA-GCUGCAUAUCUGCAUUCGAAAUGCACACAAUAUAUGGUAUAUCCCAUAAUCC .((((((((((.((((.((((((((----...)).)))))).....)))).))))))))))(((...-(((..((((.(((((.....)))))....))))..)))....)))....... ( -39.80) >DroSim_CAF1 123776 110 - 1 UGCUGGCCAAAAAUUUAUGGCCCCG----AGCCGCGGGCUAAUUGCAAAUAUUUGGCCAGCGGGCUA-GCUGCGUAUCUGCAUUCGAAAUGCACACAAUAU-----AUAUCCCAUAAUCC .((((((((((.((((.((((((((----...)).)))))).....)))).))))))))))(((.((-..........(((((.....)))))........-----.)).)))....... ( -36.51) >DroEre_CAF1 126783 107 - 1 UGCUGGCCAAAAAUUUAUGGCC-CG----AGCCGCGGGCUAAUUGCAAAUAUUUGGCCAGUGGGUUU-AGUGCGUAUCUGCAUUCGAUAUGCACACUAU-------AUAUCCCAUAAUCC .((((((((((.((((.(((((-((----.....))))))).....)))).))))))))))(((..(-((((((((((.......)))))))))....)-------)...)))....... ( -42.70) >DroYak_CAF1 129057 111 - 1 UGCUGGCCAAAAAUUUAUGGCCCCC----AGUCGCGGGCUAAUUGCAAAUAUUUGGCCAGUGGGCUU-ACUGCGUAUCUGCAUUCGAAAUGCACACAAUAU----UAUAUCCAAUAAUCC .((((((((((.((((.((((((.(----....).)))))).....)))).))))))))))((((..-...)).....(((((.....)))))........----.....))........ ( -32.10) >DroAna_CAF1 130691 94 - 1 ------CCAAAAAUGUAUAUCCCCUUCUCAUUUGUCAGCUAAUUGUAAAUAUUUACCUAGUACAUUUAACAUCAU------AUUGAACAUGCAC--------------GUGCAAUAAUCC ------....((((((((...........(((((.(((....)))))))).........)))))))).......(------((((.((......--------------)).))))).... ( -7.75) >consensus UGCUGGCCAAAAAUUUAUGGCCCCG____AGCCGCGGGCUAAUUGCAAAUAUUUGGCCAGCGGGCUA_ACUGCAUAUCUGCAUUCGAAAUGCACACAAUA______AUAUCCCAUAAUCC .((((((((((.((((.((((((............)))))).....)))).)))))))))).........(((((.((.......)).)))))........................... (-19.85 = -21.97 + 2.11)

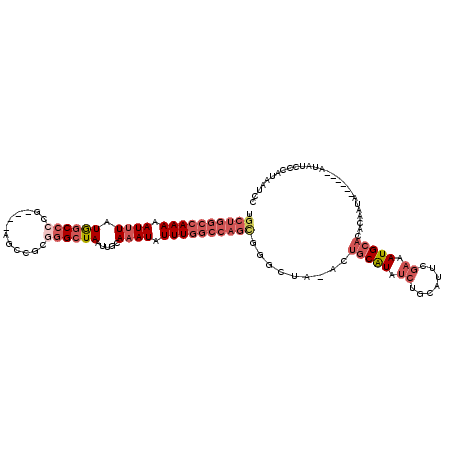
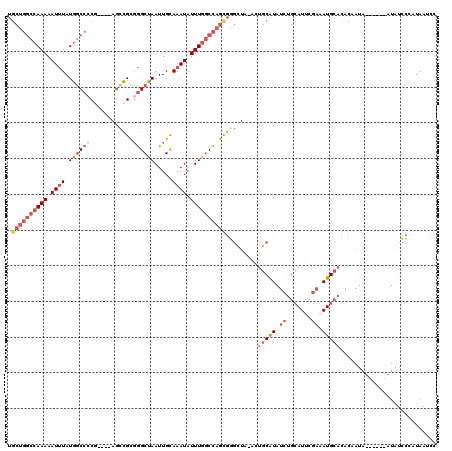
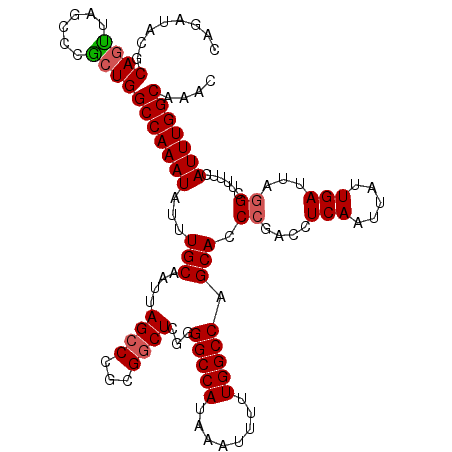
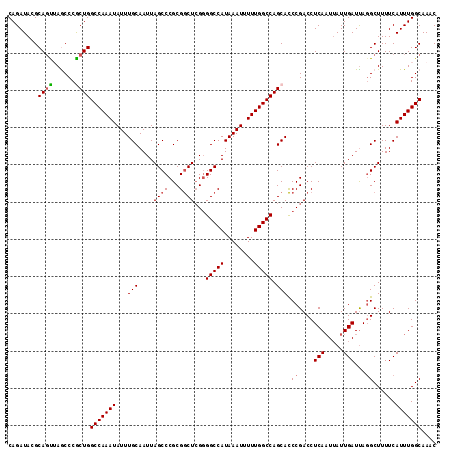
| Location | 2,123,731 – 2,123,846 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 94.26 |
| Mean single sequence MFE | -31.64 |
| Consensus MFE | -26.02 |
| Energy contribution | -26.14 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.69 |
| SVM RNA-class probability | 0.824848 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2123731 115 + 22407834 CAGAUAUGCAGUUAGCCCGCUGGCCAAAUAUUUGCAAUUAGCCCGCGGCUCGGGGCCAUAAAUUUUUGGCCAGCACCCGACCUCAAUUAUUGAUCACGCUUUUCAUUUGGCAAAC ..........(((.(((.((((((((((.(((((......((((.((...))))))..))))).))))))))))....((..(((.....))))).............))).))) ( -32.00) >DroSec_CAF1 124034 114 + 1 CAGAUAUGCAGCUAACCCGCUGGCCAAAUAUUUGCAAUUAGCCCGCGGCUCGGGGCCAUAAAUUUUUGGCCAGCACCCGACCUCA-UUAUUGAUUAGGCUUUUCAUUUGGCAAAC ..........(((((...((((((((((.(((((......((((.((...))))))..))))).))))))))))..((....(((-....)))...))........))))).... ( -33.70) >DroSim_CAF1 123811 115 + 1 CAGAUACGCAGCUAGCCCGCUGGCCAAAUAUUUGCAAUUAGCCCGCGGCUCGGGGCCAUAAAUUUUUGGCCAGCACCCGACCUCAAUUAUUGAUUAGGCUUUUCAUUUGGCAAAC ..........(((((((.((((((((((.(((((......((((.((...))))))..))))).))))))))))........(((.....)))...))))........))).... ( -33.40) >DroEre_CAF1 126816 114 + 1 CAGAUACGCACUAAACCCACUGGCCAAAUAUUUGCAAUUAGCCCGCGGCUCG-GGCCAUAAAUUUUUGGCCAGCACCCGACCUCAAUUAUUGAUUAGGCUUUUCAUUUGGCAAAC ..((...((.((((.....(((((((((.(((((......(((((.....))-)))..))))).))))))))).........(((.....)))))))))...))........... ( -28.60) >DroYak_CAF1 129093 115 + 1 CAGAUACGCAGUAAGCCCACUGGCCAAAUAUUUGCAAUUAGCCCGCGACUGGGGGCCAUAAAUUUUUGGCCAGCAUCCGACCUCAAUAAUUGAUUAGGCUUUUCAUUUGGCAAAC .......((...(((((..(((((((((.(((((......((((.(....).))))..))))).))))))))).........(((.....)))...)))))........)).... ( -30.50) >consensus CAGAUACGCAGUUAGCCCGCUGGCCAAAUAUUUGCAAUUAGCCCGCGGCUCGGGGCCAUAAAUUUUUGGCCAGCACCCGACCUCAAUUAUUGAUUAGGCUUUUCAUUUGGCAAAC ........((((......))))(((((((...(((....((((...))))...(((((........))))).))).((....(((.....)))...))......))))))).... (-26.02 = -26.14 + 0.12)

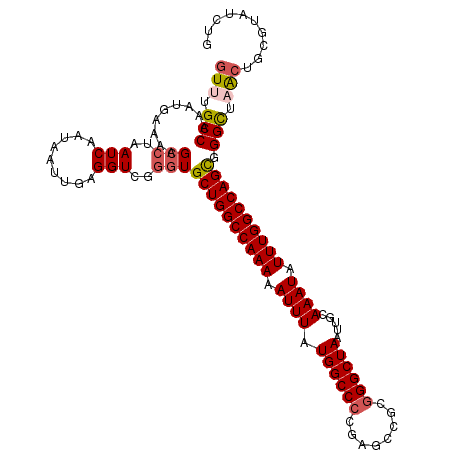
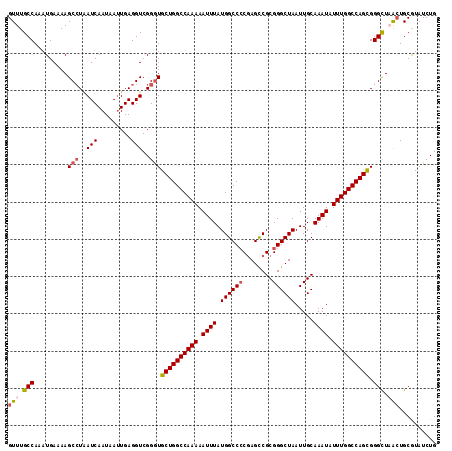
| Location | 2,123,731 – 2,123,846 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 94.26 |
| Mean single sequence MFE | -38.50 |
| Consensus MFE | -33.14 |
| Energy contribution | -33.62 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.20 |
| Structure conservation index | 0.86 |
| SVM decision value | 2.55 |
| SVM RNA-class probability | 0.995152 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2123731 115 - 22407834 GUUUGCCAAAUGAAAAGCGUGAUCAAUAAUUGAGGUCGGGUGCUGGCCAAAAAUUUAUGGCCCCGAGCCGCGGGCUAAUUGCAAAUAUUUGGCCAGCGGGCUAACUGCAUAUCUG (((.(((.........((.(((((.........))))).))((((((((((.((((.((((((((...)).)))))).....)))).)))))))))).))).))).......... ( -41.10) >DroSec_CAF1 124034 114 - 1 GUUUGCCAAAUGAAAAGCCUAAUCAAUAA-UGAGGUCGGGUGCUGGCCAAAAAUUUAUGGCCCCGAGCCGCGGGCUAAUUGCAAAUAUUUGGCCAGCGGGUUAGCUGCAUAUCUG (((.(((.........(((..(((.....-...)))..)))((((((((((.((((.((((((((...)).)))))).....)))).)))))))))).))).))).......... ( -40.00) >DroSim_CAF1 123811 115 - 1 GUUUGCCAAAUGAAAAGCCUAAUCAAUAAUUGAGGUCGGGUGCUGGCCAAAAAUUUAUGGCCCCGAGCCGCGGGCUAAUUGCAAAUAUUUGGCCAGCGGGCUAGCUGCGUAUCUG (((.(((.........(((..(((.........)))..)))((((((((((.((((.((((((((...)).)))))).....)))).)))))))))).))).))).......... ( -40.70) >DroEre_CAF1 126816 114 - 1 GUUUGCCAAAUGAAAAGCCUAAUCAAUAAUUGAGGUCGGGUGCUGGCCAAAAAUUUAUGGCC-CGAGCCGCGGGCUAAUUGCAAAUAUUUGGCCAGUGGGUUUAGUGCGUAUCUG ((..(((.........(((..(((.........)))..)))((((((((((.((((.(((((-((.....))))))).....)))).)))))))))).))).....))....... ( -36.80) >DroYak_CAF1 129093 115 - 1 GUUUGCCAAAUGAAAAGCCUAAUCAAUUAUUGAGGUCGGAUGCUGGCCAAAAAUUUAUGGCCCCCAGUCGCGGGCUAAUUGCAAAUAUUUGGCCAGUGGGCUUACUGCGUAUCUG ((..(((.........((((............)))).....((((((((((.((((.((((((.(....).)))))).....)))).)))))))))).)))..)).......... ( -33.90) >consensus GUUUGCCAAAUGAAAAGCCUAAUCAAUAAUUGAGGUCGGGUGCUGGCCAAAAAUUUAUGGCCCCGAGCCGCGGGCUAAUUGCAAAUAUUUGGCCAGCGGGCUAACUGCGUAUCUG (((.(((.........(((..(((.........)))..)))((((((((((.((((.((((((........)))))).....)))).)))))))))).))).))).......... (-33.14 = -33.62 + 0.48)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:44:21 2006