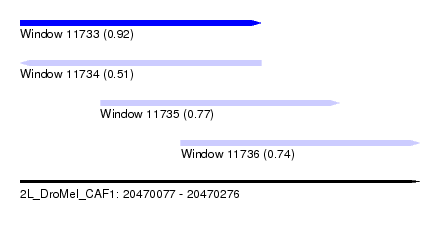
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 20,470,077 – 20,470,276 |
| Length | 199 |
| Max. P | 0.921756 |

| Location | 20,470,077 – 20,470,197 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.08 |
| Mean single sequence MFE | -31.08 |
| Consensus MFE | -24.82 |
| Energy contribution | -25.86 |
| Covariance contribution | 1.04 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.80 |
| SVM decision value | 1.14 |
| SVM RNA-class probability | 0.921756 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20470077 120 + 22407834 UUGCCAUUUUGUGCUGUUUAUUUUACGAGCGAGCAAAUUAAGUGCAUUGAUUUCUAUCGCACAGCAUCAUGAGAUGUUUGCGAUCAUAAAAUUGUUGCUCGUAUGCGUUUUUAUGGUCCU ..(((((....(((.........((((((((((((.......)))...(((((..((((((.((((((....))))))))))))....))))).))))))))).))).....)))))... ( -32.70) >DroSec_CAF1 23075 120 + 1 UUGCCAUUUUGUGCUGUUUAUUUUACGAGCGAGCAAAUUAAGUGCAUUGAUUUCUAUCGCACAGCAUCAUGAGAUGUUUGCGAUCAUAAAAUUGUGGCUCGUAUGCGUUUUUAUGGUCCU ..(((((....(((.........(((((((.((.(((((((.....)))))))))((((((.((((((....))))))))))))((((....))))))))))).))).....)))))... ( -32.10) >DroSim_CAF1 25023 120 + 1 UUGCCAUUUUGUGCUGUUUAUUUUACGAGCGAGCAAAUUAAGUGCAUUGAUUUCUAUCGCACAGCAUCAAUAGAUGUUUGCGAUCAUAAAAUUGUGGCUCGUAUGCGUUUUUAUGGUUCU ..(((((....(((.........(((((((.((.(((((((.....)))))))))((((((.((((((....))))))))))))((((....))))))))))).))).....)))))... ( -32.10) >DroEre_CAF1 25344 118 + 1 UGGCCAUU-UUUGCUGUUUAUUUUACGACCAAGCAAAUUAAUGGCAUUGAUUUCAAUCGC-CAGCAUCAAGUGAUGUUUGCGUUCAUAAAAUUCCGGCUCGUGUGCGUUUUUAUGGUCCA .((((((.-...((((...((((((.((....((((.....((((((((....)))).))-))(((((....)))))))))..)).))))))..)))).((....)).....)))))).. ( -30.60) >DroYak_CAF1 22539 119 + 1 UGGCAAUUUUUUGCUGUUUGU-UUACGACCAAGCAAAUUAAGUGCAUUGAUUUCAAUCGCCCAGCAUCAAGAGAUGUUUGCGAUCAUAAAAUUCUGGCUCGUAUGCGUUUUUAUGGUCCA ..(((....((((((..(((.-...)))...)))))).....)))((((....)))).((..((((((....)))))).))((((((((((.....((......))..)))))))))).. ( -27.90) >consensus UUGCCAUUUUGUGCUGUUUAUUUUACGAGCGAGCAAAUUAAGUGCAUUGAUUUCUAUCGCACAGCAUCAAGAGAUGUUUGCGAUCAUAAAAUUGUGGCUCGUAUGCGUUUUUAUGGUCCU ..(((((....(((.........(((((((..(.(((((((.....)))))))).((((((.((((((....))))))))))))............))))))).))).....)))))... (-24.82 = -25.86 + 1.04)

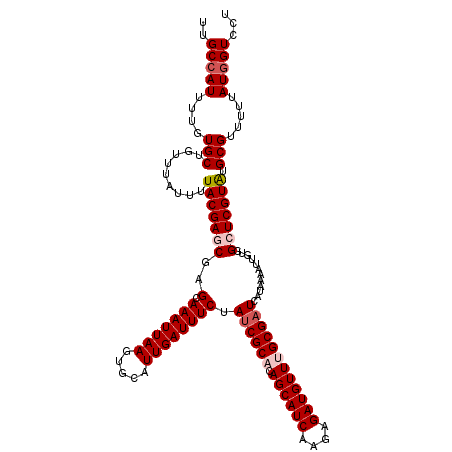

| Location | 20,470,077 – 20,470,197 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.08 |
| Mean single sequence MFE | -26.90 |
| Consensus MFE | -18.30 |
| Energy contribution | -19.70 |
| Covariance contribution | 1.40 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.44 |
| Structure conservation index | 0.68 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.510590 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20470077 120 - 22407834 AGGACCAUAAAAACGCAUACGAGCAACAAUUUUAUGAUCGCAAACAUCUCAUGAUGCUGUGCGAUAGAAAUCAAUGCACUUAAUUUGCUCGCUCGUAAAAUAAACAGCACAAAAUGGCAA ..............((.(((((((....(((((...((((((..((((....))))...)))))).)))))....(((.......)))..))))))).......((........)))).. ( -27.30) >DroSec_CAF1 23075 120 - 1 AGGACCAUAAAAACGCAUACGAGCCACAAUUUUAUGAUCGCAAACAUCUCAUGAUGCUGUGCGAUAGAAAUCAAUGCACUUAAUUUGCUCGCUCGUAAAAUAAACAGCACAAAAUGGCAA ..............((.(((((((....(((((...((((((..((((....))))...)))))).)))))....(((.......)))..))))))).......((........)))).. ( -26.40) >DroSim_CAF1 25023 120 - 1 AGAACCAUAAAAACGCAUACGAGCCACAAUUUUAUGAUCGCAAACAUCUAUUGAUGCUGUGCGAUAGAAAUCAAUGCACUUAAUUUGCUCGCUCGUAAAAUAAACAGCACAAAAUGGCAA ..............((.(((((((....(((((...((((((..((((....))))...)))))).)))))....(((.......)))..))))))).......((........)))).. ( -26.40) >DroEre_CAF1 25344 118 - 1 UGGACCAUAAAAACGCACACGAGCCGGAAUUUUAUGAACGCAAACAUCACUUGAUGCUG-GCGAUUGAAAUCAAUGCCAUUAAUUUGCUUGGUCGUAAAAUAAACAGCAAA-AAUGGCCA .((.((((.....((....)).((.(..(((((((((..(((((((((....)))).((-((.((((....))))))))....)))))....)))))))))...).))...-.)))))). ( -30.80) >DroYak_CAF1 22539 119 - 1 UGGACCAUAAAAACGCAUACGAGCCAGAAUUUUAUGAUCGCAAACAUCUCUUGAUGCUGGGCGAUUGAAAUCAAUGCACUUAAUUUGCUUGGUCGUAA-ACAAACAGCAAAAAAUUGCCA .................(((((.((((........((((((..(((((....)))).)..)))))).........(((.......)))))))))))).-.......((((....)))).. ( -23.60) >consensus AGGACCAUAAAAACGCAUACGAGCCACAAUUUUAUGAUCGCAAACAUCUCUUGAUGCUGUGCGAUAGAAAUCAAUGCACUUAAUUUGCUCGCUCGUAAAAUAAACAGCACAAAAUGGCAA .................(((((((....(((((...((((((..((((....))))...)))))).)))))....(((.......)))..))))))).........((........)).. (-18.30 = -19.70 + 1.40)

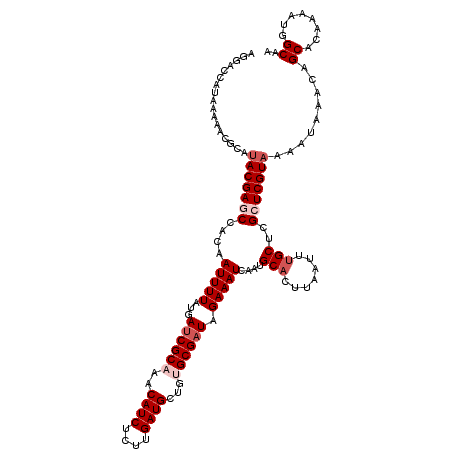
| Location | 20,470,117 – 20,470,236 |
|---|---|
| Length | 119 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.55 |
| Mean single sequence MFE | -28.86 |
| Consensus MFE | -24.48 |
| Energy contribution | -24.96 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.42 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.51 |
| SVM RNA-class probability | 0.765456 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20470117 119 + 22407834 AGUGCAUUGAUUUCUAUCGCACAGCAUCAUGAGAUGUUUGCGAUCAUAAAAUUGUUGCUCGUAUGCGUUUUUAUGGUCCUUUCUUAGGGGACAAAACGUAUC-UAAAUAUGUAUUUGUAU .(.(((..(((((..((((((.((((((....))))))))))))....)))))..))).)(.(((((((((....((((((.....))))))))))))))))-................. ( -32.10) >DroSec_CAF1 23115 119 + 1 AGUGCAUUGAUUUCUAUCGCACAGCAUCAUGAGAUGUUUGCGAUCAUAAAAUUGUGGCUCGUAUGCGUUUUUAUGGUCCUUUCUUAGGGGACAAAACGUAUC-UAAAUAUGUAUUUGUAU .((((...(((....))))))).(((.((((((((((.((((((((((....))))).))))).))).)))))))((((((.....))))))...((((((.-...))))))...))).. ( -30.40) >DroSim_CAF1 25063 119 + 1 AGUGCAUUGAUUUCUAUCGCACAGCAUCAAUAGAUGUUUGCGAUCAUAAAAUUGUGGCUCGUAUGCGUUUUUAUGGUUCUUUCUUUGGGGACAAAACGUAUC-UAAAUAUGUAUUUGUAU .((((((((.(..((((((((.((((((....)))))))))))((((((((.((((.......)))).)))))))).........)))..)))).((((((.-...))))))...))))) ( -27.70) >DroEre_CAF1 25383 118 + 1 AUGGCAUUGAUUUCAAUCGC-CAGCAUCAAGUGAUGUUUGCGUUCAUAAAAUUCCGGCUCGUGUGCGUUUUUAUGGUCCAUUCUCAGGGGACAAAAUGUAUC-UAAAUAUGUAUUUGUAU .((((((((....)))).))-))(((((....))))).(((((((((((((...((((....)).)).)))))))((((.........))))..))))))..-................. ( -24.80) >DroYak_CAF1 22578 120 + 1 AGUGCAUUGAUUUCAAUCGCCCAGCAUCAAGAGAUGUUUGCGAUCAUAAAAUUCUGGCUCGUAUGCGUUUUUAUGGUCCAUUCUUAGGGGACAGAAUGUAUCCUAAAUAUGUAUUUGUAU .(.((...(((((..(((((..((((((....)))))).)))))....)))))...)).)..((((((.((((.(((.((((((..(....))))))).))).)))).))))))...... ( -29.30) >consensus AGUGCAUUGAUUUCUAUCGCACAGCAUCAAGAGAUGUUUGCGAUCAUAAAAUUGUGGCUCGUAUGCGUUUUUAUGGUCCUUUCUUAGGGGACAAAACGUAUC_UAAAUAUGUAUUUGUAU .(.((...(((((..((((((.((((((....))))))))))))....)))))...)).)..(((((((((....((((((.....)))))))))))))))................... (-24.48 = -24.96 + 0.48)


| Location | 20,470,157 – 20,470,276 |
|---|---|
| Length | 119 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.47 |
| Mean single sequence MFE | -30.16 |
| Consensus MFE | -25.80 |
| Energy contribution | -26.08 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.39 |
| Structure conservation index | 0.86 |
| SVM decision value | 0.44 |
| SVM RNA-class probability | 0.737805 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20470157 119 + 22407834 CGAUCAUAAAAUUGUUGCUCGUAUGCGUUUUUAUGGUCCUUUCUUAGGGGACAAAACGUAUC-UAAAUAUGUAUUUGUAUCUGCAUCUGCAUAAGUGGCAUGUUCUCUGCAUGUCCCGCA .((((((((((....(((......))).)))))))))).........((((((..((((((.-...))))))...((((...(((((..(....)..).))))....))))))))))... ( -32.30) >DroSec_CAF1 23155 119 + 1 CGAUCAUAAAAUUGUGGCUCGUAUGCGUUUUUAUGGUCCUUUCUUAGGGGACAAAACGUAUC-UAAAUAUGUAUUUGUAUCUGCAUCUGCAUAAGUGGCAUGUUCUCUGCAUGUCCCGCA ((((((((....))))).))).(((((((((....((((((.....))))))))))))))).-....((((((..(((....)))..)))))).(((((((((.....)))))..)))). ( -32.80) >DroSim_CAF1 25103 119 + 1 CGAUCAUAAAAUUGUGGCUCGUAUGCGUUUUUAUGGUUCUUUCUUUGGGGACAAAACGUAUC-UAAAUAUGUAUUUGUAUCUGCAUCUGCAUAAGUGGCAUGUUCUCUGCAUGUCCCGCA ((((((((....))))).))).(((((((((....((((((.....))))))))))))))).-....((((((..(((....)))..)))))).(((((((((.....)))))..)))). ( -30.10) >DroEre_CAF1 25422 119 + 1 CGUUCAUAAAAUUCCGGCUCGUGUGCGUUUUUAUGGUCCAUUCUCAGGGGACAAAAUGUAUC-UAAAUAUGUAUUUGUAUCUGCAUCUGCAUAAGUGGCAUGUUCUCUGCAUGUACCGCA ....(((((((...((((....)).)).)))))))((((.........))))..........-....((((((..(((....)))..)))))).(((((((((.....)))))..)))). ( -25.50) >DroYak_CAF1 22618 120 + 1 CGAUCAUAAAAUUCUGGCUCGUAUGCGUUUUUAUGGUCCAUUCUUAGGGGACAGAAUGUAUCCUAAAUAUGUAUUUGUAUCUGCAUCUGCAUAAGUGGCAUGUUCUCUUGAUGUCCCGCA .((((((((((.....((......))..)))))))))).........((((((((((((...(((..((((((..(((....)))..))))))..))).))))))......))))))... ( -30.10) >consensus CGAUCAUAAAAUUGUGGCUCGUAUGCGUUUUUAUGGUCCUUUCUUAGGGGACAAAACGUAUC_UAAAUAUGUAUUUGUAUCUGCAUCUGCAUAAGUGGCAUGUUCUCUGCAUGUCCCGCA ................((....(((((((((....((((((.....)))))))))))))))......((((((..(((....)))..)))))).(.(((((((.....))))))).))). (-25.80 = -26.08 + 0.28)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:33:30 2006