
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 20,449,842 – 20,450,042 |
| Length | 200 |
| Max. P | 0.992396 |

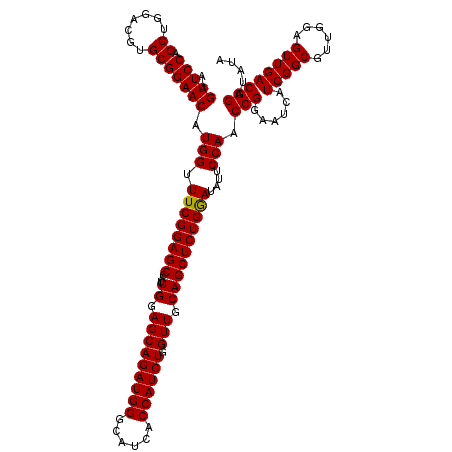
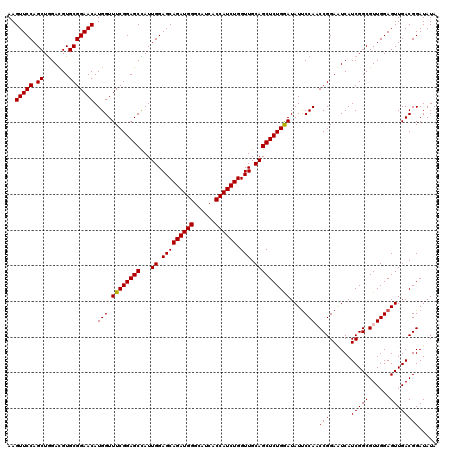
| Location | 20,449,842 – 20,449,962 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.67 |
| Mean single sequence MFE | -43.24 |
| Consensus MFE | -41.30 |
| Energy contribution | -41.06 |
| Covariance contribution | -0.24 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.73 |
| Structure conservation index | 0.96 |
| SVM decision value | 2.33 |
| SVM RNA-class probability | 0.992396 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
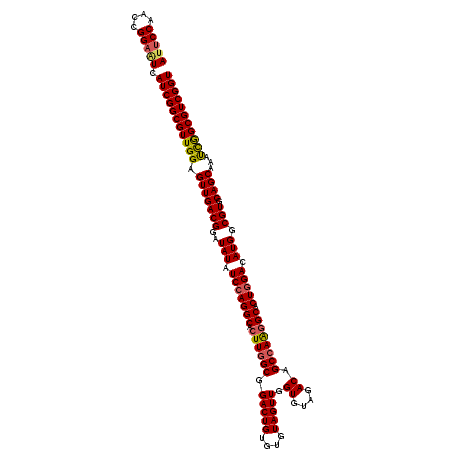
>2L_DroMel_CAF1 20449842 120 + 22407834 AAGUUCCAGCUGGACGUGCGGAACAUGGUUUCGGAGCCAUUGGAGCAGAUGGGCAUCACCAUCUGGUUGCAGCUCUGGAUAUUCCAACCGGAGUCAUCGGCGUUGGAGUUGACGGAUAUA ..(((((.((.......))))))).....((((((((...((.(((((((((......)))))).))).)))))))))).(((((((((.((....)).).))))))))........... ( -43.60) >DroSec_CAF1 2828 120 + 1 AAGUUCCAGCUGGACGUGCGGAACAUGGUUUCGGAGCCAUUGGAGCAGAUGGGCAUCACCAUCUGGUUGCAGCUCUGGAUAUUCCAACCGGAAUCAUCGGCGUUGGAGUUGACGGAUAUA ..(((((.((.......))))))).....((((((((...((.(((((((((......)))))).))).)))))))))).(((((((((.((....)).).))))))))........... ( -42.80) >DroSim_CAF1 2882 120 + 1 AAGUUCCAGCUGGACGUGCGGAACAUGGUUUCGGAGCCAUUGGAGCAGAUGGGCAUCACCAUCUGGUUGCAGCUCUGGAUAUUCCAACCGGAGUCAUCGGCGUUGGAGUUGACGGAUAUA ..(((((.((.......))))))).....((((((((...((.(((((((((......)))))).))).)))))))))).(((((((((.((....)).).))))))))........... ( -43.60) >DroEre_CAF1 5085 120 + 1 AAGUUCCAGCUGGAUGUGCGGAACAUGGUUUCGGAGCUAUUGGAGCAGAUGGGCAUCACCAUCUGGUUGCAGCUCUGAAUAUCCCACCCGGAUUCAUCGGCGUUGGAGUUGACGGAUAUA ...((((((((.(((((.(((....(((.(((((((((....((.(((((((......))))))).))..)))))))))....))).))).)..)))).).)))))))............ ( -43.00) >DroYak_CAF1 2876 120 + 1 AAGUUCCAGCUAGAUGUGCGGAACAUGGUUUCGGAGCUAUUGGAGCAGAUGGGCAUCACCAUCUGGUUGCAGCUCUGAAUAUUCCACCCGGAAUCAUCGGCGUUGGAGUUGACGGAUAUA ...(((((((..(((((.(((....(((.(((((((((....((.(((((((......))))))).))..)))))))))....))).))).)..))))...)))))))............ ( -43.20) >consensus AAGUUCCAGCUGGACGUGCGGAACAUGGUUUCGGAGCCAUUGGAGCAGAUGGGCAUCACCAUCUGGUUGCAGCUCUGGAUAUUCCAACCGGAAUCAUCGGCGUUGGAGUUGACGGAUAUA ..(((((.((.......))))))).(((.((((((((...((.(((((((((......)))))).))).))))))))))....))).(((......(((((......))))))))..... (-41.30 = -41.06 + -0.24)

| Location | 20,449,842 – 20,449,962 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.67 |
| Mean single sequence MFE | -34.49 |
| Consensus MFE | -31.42 |
| Energy contribution | -31.26 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.45 |
| Structure conservation index | 0.91 |
| SVM decision value | 0.96 |
| SVM RNA-class probability | 0.889493 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20449842 120 - 22407834 UAUAUCCGUCAACUCCAACGCCGAUGACUCCGGUUGGAAUAUCCAGAGCUGCAACCAGAUGGUGAUGCCCAUCUGCUCCAAUGGCUCCGAAACCAUGUUCCGCACGUCCAGCUGGAACUU .......((((.(.........).))))((((((((((.......((((......(((((((......))))))).....((((........))))))))......)))))))))).... ( -33.72) >DroSec_CAF1 2828 120 - 1 UAUAUCCGUCAACUCCAACGCCGAUGAUUCCGGUUGGAAUAUCCAGAGCUGCAACCAGAUGGUGAUGCCCAUCUGCUCCAAUGGCUCCGAAACCAUGUUCCGCACGUCCAGCUGGAACUU ..(((((((........)))..)))).(((((((((((.......((((......(((((((......))))))).....((((........))))))))......)))))))))))... ( -33.92) >DroSim_CAF1 2882 120 - 1 UAUAUCCGUCAACUCCAACGCCGAUGACUCCGGUUGGAAUAUCCAGAGCUGCAACCAGAUGGUGAUGCCCAUCUGCUCCAAUGGCUCCGAAACCAUGUUCCGCACGUCCAGCUGGAACUU .......((((.(.........).))))((((((((((.......((((......(((((((......))))))).....((((........))))))))......)))))))))).... ( -33.72) >DroEre_CAF1 5085 120 - 1 UAUAUCCGUCAACUCCAACGCCGAUGAAUCCGGGUGGGAUAUUCAGAGCUGCAACCAGAUGGUGAUGCCCAUCUGCUCCAAUAGCUCCGAAACCAUGUUCCGCACAUCCAGCUGGAACUU .............((((.(.(((.......)))(((((((((((.((((((....(((((((......)))))))......)))))).))....))))))))).......).)))).... ( -35.30) >DroYak_CAF1 2876 120 - 1 UAUAUCCGUCAACUCCAACGCCGAUGAUUCCGGGUGGAAUAUUCAGAGCUGCAACCAGAUGGUGAUGCCCAUCUGCUCCAAUAGCUCCGAAACCAUGUUCCGCACAUCUAGCUGGAACUU .............((((.(.(((.......)))(((((((((((.((((((....(((((((......)))))))......)))))).))....))))))))).......).)))).... ( -35.80) >consensus UAUAUCCGUCAACUCCAACGCCGAUGACUCCGGUUGGAAUAUCCAGAGCUGCAACCAGAUGGUGAUGCCCAUCUGCUCCAAUGGCUCCGAAACCAUGUUCCGCACGUCCAGCUGGAACUU ..(((((((........)))..))))..((((((((((.......((((((....(((((((......)))))))......))))))((.(((...))).))....)))))))))).... (-31.42 = -31.26 + -0.16)

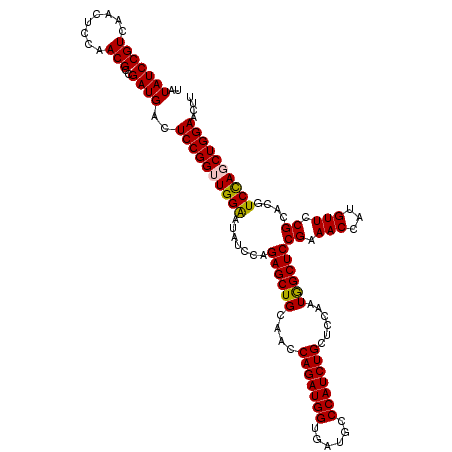
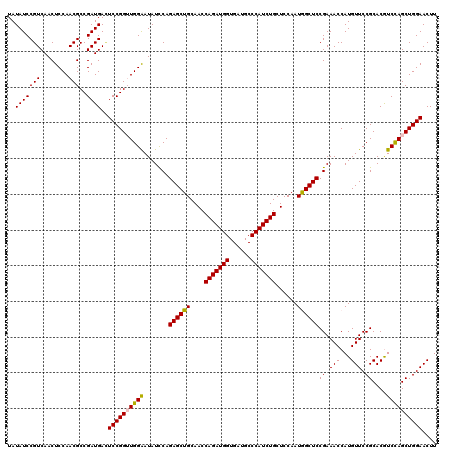
| Location | 20,449,922 – 20,450,042 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.08 |
| Mean single sequence MFE | -45.64 |
| Consensus MFE | -42.82 |
| Energy contribution | -42.94 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.54 |
| Structure conservation index | 0.94 |
| SVM decision value | 1.80 |
| SVM RNA-class probability | 0.977635 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20449922 120 + 22407834 AUUCCAACCGGAGUCAUCGGCGUUGGAGUUGACGGAUAUAUCGAGGCACUUGGCGGACUGUGUGUAGUUGGUGUAGACAGCCAAAGCGCUGGACAUGGCGUGCAGCAAAUCGGCGUCGGU (((((((((.((....)).).))))))))(((((..((((((((.((((.(((....))).))))..))))))))..).(((.....((((.((.....)).)))).....))))))).. ( -41.10) >DroSec_CAF1 2908 120 + 1 AUUCCAACCGGAAUCAUCGGCGUUGGAGUUGACGGAUAUAUCCAGGCACUUGGCGGACUGUGUGUAGUUGGUGUAGACAGCGAGGGCACUGGACAUGGCGUGCAGCAAAUCGGCGUCGGU (((((....))))).(((((((((((.(((((((..(((.(((((((.(((.((...(((..(.......)..)))...)).))))).))))).))).))).))))...))))))))))) ( -42.50) >DroSim_CAF1 2962 120 + 1 AUUCCAACCGGAGUCAUCGGCGUUGGAGUUGACGGAUAUAUCCAGGCACUUGGCGGACUGUGUGUAGUUGGUGUAGACAGCCAGGGCACUGGACAUGGCGUGCAGCAAAUCGGCGUCGGU (((((....))))).(((((((((((.(((((((..(((.(((((((.((((((...(((..(.......)..)))...)))))))).))))).))).))).))))...))))))))))) ( -46.90) >DroEre_CAF1 5165 120 + 1 AUCCCACCCGGAUUCAUCGGCGUUGGAGUUGACGGAUAUAUCCAGGCACUUGGCGGACUGUGUGUAGUUGGUGUACACAGCCAAGGCACUGGACAUGGCGUGCAGCAAGCUGGCGUCGGU ((((.....))))..((((((((..(.(((((((..(((.(((((((.((((((....((((((((.....)))))))))))))))).))))).))).))).))))...)..)))))))) ( -48.70) >DroYak_CAF1 2956 120 + 1 AUUCCACCCGGAAUCAUCGGCGUUGGAGUUGACGGAUAUAUCCAGGCACUUGGCAGACUGUGUGUAGUUGGUGUAAACUGCCAAGGCACUGGACAUGGCGUGCAGCAAAUCAGCGUCGGU (((((....))))).(((((((((((.(((((((..(((.(((((((.((((((((..((..(.......)..))..)))))))))).))))).))).))).))))...))))))))))) ( -49.00) >consensus AUUCCAACCGGAAUCAUCGGCGUUGGAGUUGACGGAUAUAUCCAGGCACUUGGCGGACUGUGUGUAGUUGGUGUAGACAGCCAAGGCACUGGACAUGGCGUGCAGCAAAUCGGCGUCGGU (((((....))))).(((((((((((.(((((((..(((.(((((((.((((((.(((((....))))).((....)).)))))))).))))).))).))).))))...))))))))))) (-42.82 = -42.94 + 0.12)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:32:38 2006