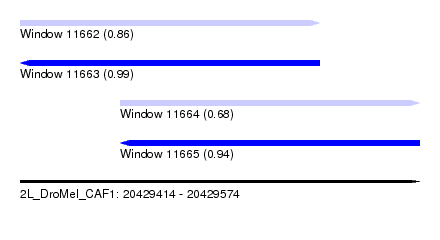
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 20,429,414 – 20,429,574 |
| Length | 160 |
| Max. P | 0.994410 |

| Location | 20,429,414 – 20,429,534 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.89 |
| Mean single sequence MFE | -49.41 |
| Consensus MFE | -41.63 |
| Energy contribution | -41.38 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.81 |
| SVM RNA-class probability | 0.857191 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
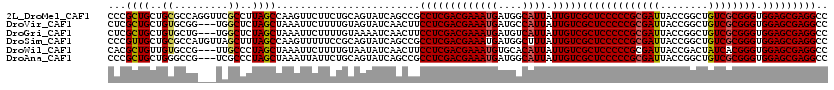
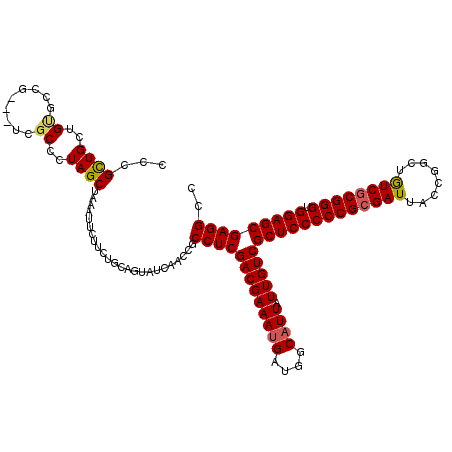
>2L_DroMel_CAF1 20429414 120 + 22407834 UCACCCGUCUCGCCGAUCGCCAGCACGUCCAGUUAGGGGCGGCCUCGCUCCACCCGCGACAGCCGGUAAUCGCGGGGGAGCGACAAUAAUGCCAUCAUUUCGUCGAGGCGGCUGAUACUG (((.((((((((.(((......((((((((......)))))...(((((((.(((((((..........))))))))))))))......))).......))).)))))))).)))..... ( -56.32) >DroVir_CAF1 11370 120 + 1 UCACCCGUCUCGCCCAUUGCGAGCACCUCCAGUUAGGGGCGGCCUCGCUCCACCCGCGACAGCCGGUAAUCGCGGGGGAGCGACAAUAAUGGCAUCAUUUCGUCGAGGAAGUUGAUACUA (((.(((((((....((((.(((...)))))))..)))))))(((((((((.(((((((..........))))))))))))(((...((((....))))..)))))))....)))..... ( -48.00) >DroGri_CAF1 11854 120 + 1 UCACCCGUCUCGCCGAUCGCCAGUACGUCCAGUUAGGGGCGGCCUCGCUCCACCCGCGACAGCCGGUAAUCGCGGGGGAGCGACAAUAAUGACAUCAUUUCGUCGAGGAAGUUGAUUUUA (((.(..(((((.((((((((...((.....))....)))))..(((((((.(((((((..........))))))))))))))................))).)))))..).)))..... ( -45.80) >DroWil_CAF1 22287 120 + 1 UCACCAGUAUCACCGAUUGCCAGUACGUCCAGUUAGGGGCGGCCUCGCUCCACCCGUGAUAGUCGGUAAUCGCGGGGGAGCGACAAUAAUGUGCACAUUUCGUCGAGGAAGUUGAUAUUA .....(((((((.(...........(((((......))))).(((((((((.((((((((........)))))))))))))(((...((((....))))..)))))))..).))))))). ( -48.30) >DroMoj_CAF1 13928 120 + 1 UCACCCGUCUCGCCCAUCGCCAGUACGUCCAGUUAGGGGCGGCCUCGCUCCACCCGCGACAGCCGGUAAUCGCGGGGGAGCGACAAUAAUGACAUCAUUUCGUCGAGGAAGUUGAUUUUA (((.(((((((......((......))........)))))))(((((((((.(((((((..........))))))))))))(((...((((....))))..)))))))....)))..... ( -45.14) >DroAna_CAF1 7644 120 + 1 UCCCCCGUCUCGCCGAUCGCCAGUACGUCCAGUUAGGGGCGGCCUCGCUCCACCCGCGACAGCCGGUAAUCGCGGGGGAGCGACAAUAAUGCCAUCAUUUCGUCGAGGCGGCUGAUACUG ((..((((((((.(((..(((...((.....))....)))(((.(((((((.(((((((..........)))))))))))))).......)))......))).))))))))..))..... ( -52.90) >consensus UCACCCGUCUCGCCGAUCGCCAGUACGUCCAGUUAGGGGCGGCCUCGCUCCACCCGCGACAGCCGGUAAUCGCGGGGGAGCGACAAUAAUGACAUCAUUUCGUCGAGGAAGUUGAUACUA ....................((((.(((((......))))).(((((((((.(((((((..........))))))))))))(((...((((....))))..)))))))..))))...... (-41.63 = -41.38 + -0.25)

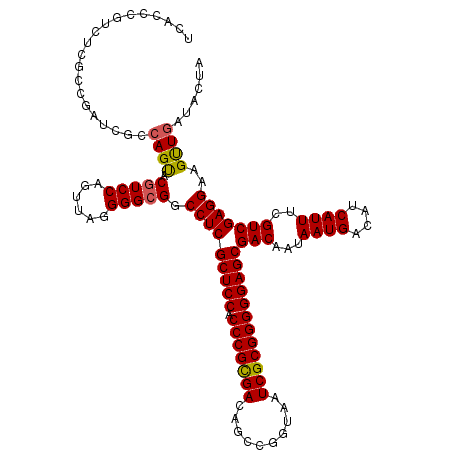
| Location | 20,429,414 – 20,429,534 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.89 |
| Mean single sequence MFE | -52.70 |
| Consensus MFE | -47.03 |
| Energy contribution | -47.58 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.48 |
| Structure conservation index | 0.89 |
| SVM decision value | 2.48 |
| SVM RNA-class probability | 0.994410 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20429414 120 - 22407834 CAGUAUCAGCCGCCUCGACGAAAUGAUGGCAUUAUUGUCGCUCCCCCGCGAUUACCGGCUGUCGCGGGUGGAGCGAGGCCGCCCCUAACUGGACGUGCUGGCGAUCGGCGAGACGGGUGA .....(((.((((((((((((((((....)))).)))))(((((((((((((........)))))))).))))))))))((((((.....)).(((....)))...))))....)).))) ( -57.70) >DroVir_CAF1 11370 120 - 1 UAGUAUCAACUUCCUCGACGAAAUGAUGCCAUUAUUGUCGCUCCCCCGCGAUUACCGGCUGUCGCGGGUGGAGCGAGGCCGCCCCUAACUGGAGGUGCUCGCAAUGGGCGAGACGGGUGA ............(((((((((((((....)))).)))))(((((((((((((........)))))))).))))))))).(((((((......))((.(((((.....)))))))))))). ( -54.50) >DroGri_CAF1 11854 120 - 1 UAAAAUCAACUUCCUCGACGAAAUGAUGUCAUUAUUGUCGCUCCCCCGCGAUUACCGGCUGUCGCGGGUGGAGCGAGGCCGCCCCUAACUGGACGUACUGGCGAUCGGCGAGACGGGUGA ............(((((((((((((....)))).)))))(((((((((((((........)))))))).))))))))).(((((((..((((.(((....))).))))..))..))))). ( -53.40) >DroWil_CAF1 22287 120 - 1 UAAUAUCAACUUCCUCGACGAAAUGUGCACAUUAUUGUCGCUCCCCCGCGAUUACCGACUAUCACGGGUGGAGCGAGGCCGCCCCUAACUGGACGUACUGGCAAUCGGUGAUACUGGUGA ............(((((((((((((....)))).)))))(((((((((.(((........))).)))).))))))))).((((((.....))...(((((.....))))).....)))). ( -42.00) >DroMoj_CAF1 13928 120 - 1 UAAAAUCAACUUCCUCGACGAAAUGAUGUCAUUAUUGUCGCUCCCCCGCGAUUACCGGCUGUCGCGGGUGGAGCGAGGCCGCCCCUAACUGGACGUACUGGCGAUGGGCGAGACGGGUGA ............(((((((((((((....)))).)))))(((((((((((((........)))))))).))))))))).((((((.....)).(((.((.((.....)).))))))))). ( -52.60) >DroAna_CAF1 7644 120 - 1 CAGUAUCAGCCGCCUCGACGAAAUGAUGGCAUUAUUGUCGCUCCCCCGCGAUUACCGGCUGUCGCGGGUGGAGCGAGGCCGCCCCUAACUGGACGUACUGGCGAUCGGCGAGACGGGGGA ........((.((((((((((((((....)))).)))))(((((((((((((........)))))))).)))))))))).))((((.......(((.((.((.....)).))))))))). ( -56.01) >consensus UAAUAUCAACUUCCUCGACGAAAUGAUGGCAUUAUUGUCGCUCCCCCGCGAUUACCGGCUGUCGCGGGUGGAGCGAGGCCGCCCCUAACUGGACGUACUGGCGAUCGGCGAGACGGGUGA ............(((((((((((((....)))).)))))(((((((((((((........)))))))).))))))))).((((((.....)).(((.((.((.....)).))))))))). (-47.03 = -47.58 + 0.56)
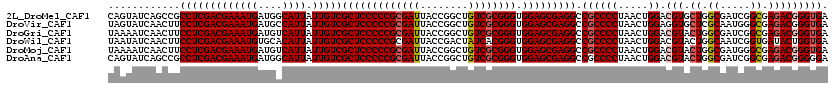
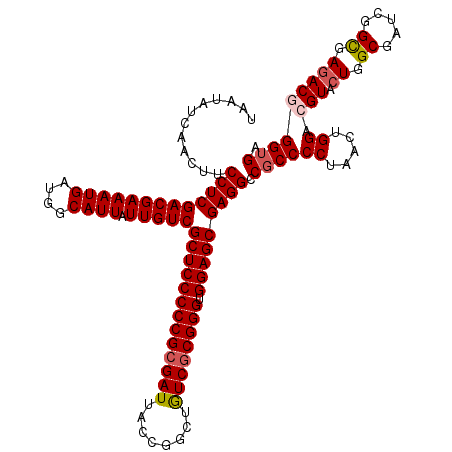
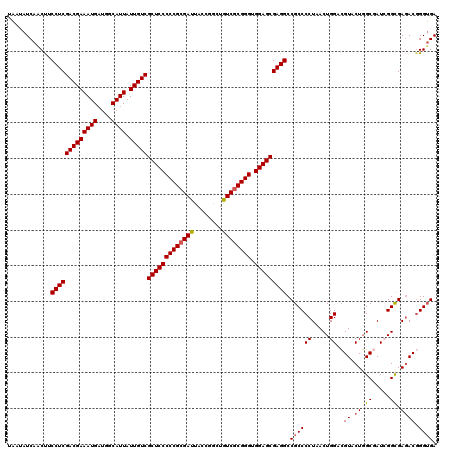
| Location | 20,429,454 – 20,429,574 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 86.59 |
| Mean single sequence MFE | -45.80 |
| Consensus MFE | -36.72 |
| Energy contribution | -36.02 |
| Covariance contribution | -0.69 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.30 |
| SVM RNA-class probability | 0.676875 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20429454 120 + 22407834 GGCCUCGCUCCACCCGCGACAGCCGGUAAUCGCGGGGGAGCGACAAUAAUGCCAUCAUUUCGUCGAGGCGGCUGAUACUGCAGAAGAACUUGGCUAAGGCGAACCUGGCGCAGCAGCGGG .((((((((((.(((((((..........))))))))))))(((...((((....))))..)))))))).((((...((((((.....))..((((.((....))))))))))))))... ( -51.00) >DroVir_CAF1 11410 117 + 1 GGCCUCGCUCCACCCGCGACAGCCGGUAAUCGCGGGGGAGCGACAAUAAUGGCAUCAUUUCGUCGAGGAAGUUGAUACUACAAAAGAAUUUAGCUAGAGCCA---CCGCACAGCAGCGAG (((((((((((.(((((((..........))))))))))))(((...((((....))))..))))))..((((((..((.....))...))))))...))).---.(((......))).. ( -41.80) >DroGri_CAF1 11894 117 + 1 GGCCUCGCUCCACCCGCGACAGCCGGUAAUCGCGGGGGAGCGACAAUAAUGACAUCAUUUCGUCGAGGAAGUUGAUUUUACAAAAGAAUUUAGCUAGAGCCA---CAGCACAGCAGCGAG (((((((((((.(((((((..........))))))))))))(((...((((....))))..))))))..((((((.(((......))).))))))...))).---..((......))... ( -42.20) >DroSim_CAF1 1185 120 + 1 GGCCUCGCUCCACCCGCGACAGCCGGUAAUCGCGGGGGAGCGACAAUAAAGCCAUCAUUUCGUCGAGGCGGCUGAUACUGCGGAAAAACUUGGCUAAAGCUAACAUGGCGCAGCAACGGG (((((((((((.(((((((..........)))))))))))))).......(((.((........)))))))))....(((((.......(((((....))))).....)))))....... ( -47.50) >DroWil_CAF1 22327 117 + 1 GGCCUCGCUCCACCCGUGAUAGUCGGUAAUCGCGGGGGAGCGACAAUAAUGUGCACAUUUCGUCGAGGAAGUUGAUAUUACAAAAGAAUUUAGCUAGGGCAA---CGGCACAACAGCGUG ..(((((((((.((((((((........)))))))))))))(((...((((....))))..)))))))..((((......(....)......(((..(....---)))).))))...... ( -40.20) >DroAna_CAF1 7684 117 + 1 GGCCUCGCUCCACCCGCGACAGCCGGUAAUCGCGGGGGAGCGACAAUAAUGCCAUCAUUUCGUCGAGGCGGCUGAUACUGCAGAAUAAUUUAGCUAGGGCGA---CGGCCCAGCAGCGGG (((((((((((.(((((((..........)))))))))))))).......(((.((........)))))))))....(((((((....))).(((.((((..---..))))))).)))). ( -52.10) >consensus GGCCUCGCUCCACCCGCGACAGCCGGUAAUCGCGGGGGAGCGACAAUAAUGCCAUCAUUUCGUCGAGGAAGCUGAUACUACAAAAGAAUUUAGCUAGAGCCA___CGGCACAGCAGCGGG ..(((((((((.(((((((..........))))))))))))(((...((((....))))..))))))).((((((..............))))))...((.......))........... (-36.72 = -36.02 + -0.69)

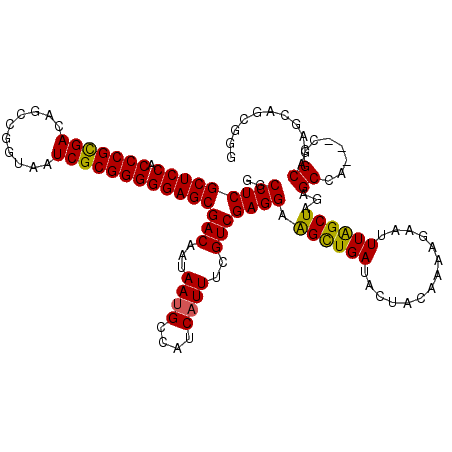
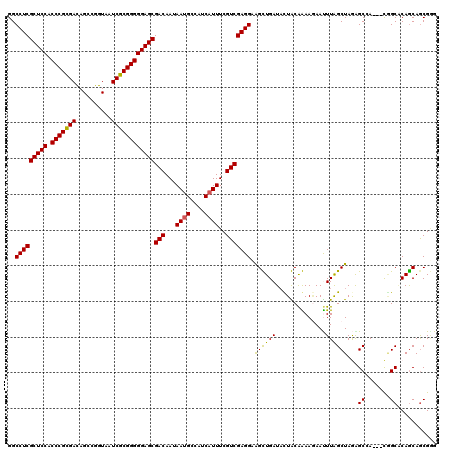
| Location | 20,429,454 – 20,429,574 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 86.59 |
| Mean single sequence MFE | -48.05 |
| Consensus MFE | -40.13 |
| Energy contribution | -40.18 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.74 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.26 |
| SVM RNA-class probability | 0.937331 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 20429454 120 - 22407834 CCCGCUGCUGCGCCAGGUUCGCCUUAGCCAAGUUCUUCUGCAGUAUCAGCCGCCUCGACGAAAUGAUGGCAUUAUUGUCGCUCCCCCGCGAUUACCGGCUGUCGCGGGUGGAGCGAGGCC ...((((((((((.(((....)))..))..((.....))))))...)))).((((((((((((((....)))).)))))(((((((((((((........)))))))).)))))))))). ( -54.30) >DroVir_CAF1 11410 117 - 1 CUCGCUGCUGUGCGG---UGGCUCUAGCUAAAUUCUUUUGUAGUAUCAACUUCCUCGACGAAAUGAUGCCAUUAUUGUCGCUCCCCCGCGAUUACCGGCUGUCGCGGGUGGAGCGAGGCC ...(((((.......---((((....)))).........)))))........(((((((((((((....)))).)))))(((((((((((((........)))))))).))))))))).. ( -46.89) >DroGri_CAF1 11894 117 - 1 CUCGCUGCUGUGCUG---UGGCUCUAGCUAAAUUCUUUUGUAAAAUCAACUUCCUCGACGAAAUGAUGUCAUUAUUGUCGCUCCCCCGCGAUUACCGGCUGUCGCGGGUGGAGCGAGGCC ...((..(......)---..))..............................(((((((((((((....)))).)))))(((((((((((((........)))))))).))))))))).. ( -45.10) >DroSim_CAF1 1185 120 - 1 CCCGUUGCUGCGCCAUGUUAGCUUUAGCCAAGUUUUUCCGCAGUAUCAGCCGCCUCGACGAAAUGAUGGCUUUAUUGUCGCUCCCCCGCGAUUACCGGCUGUCGCGGGUGGAGCGAGGCC ...(.(((((((.......(((((.....)))))....))))))).)....((((((((((..(((.....))))))))(((((((((((((........)))))))).)))))))))). ( -50.50) >DroWil_CAF1 22327 117 - 1 CACGCUGUUGUGCCG---UUGCCCUAGCUAAAUUCUUUUGUAAUAUCAACUUCCUCGACGAAAUGUGCACAUUAUUGUCGCUCCCCCGCGAUUACCGACUAUCACGGGUGGAGCGAGGCC ...((((..((....---..))..))))........................(((((((((((((....)))).)))))(((((((((.(((........))).)))).))))))))).. ( -37.30) >DroAna_CAF1 7684 117 - 1 CCCGCUGCUGGGCCG---UCGCCCUAGCUAAAUUAUUCUGCAGUAUCAGCCGCCUCGACGAAAUGAUGGCAUUAUUGUCGCUCCCCCGCGAUUACCGGCUGUCGCGGGUGGAGCGAGGCC ...((((..((((..---..))))..((...........)).....)))).((((((((((((((....)))).)))))(((((((((((((........)))))))).)))))))))). ( -54.20) >consensus CCCGCUGCUGUGCCG___UCGCCCUAGCUAAAUUCUUCUGCAGUAUCAACCGCCUCGACGAAAUGAUGGCAUUAUUGUCGCUCCCCCGCGAUUACCGGCUGUCGCGGGUGGAGCGAGGCC ...((((..((.........))..))))........................(((((((((((((....)))).)))))(((((((((((((........)))))))).))))))))).. (-40.13 = -40.18 + 0.06)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:32:26 2006