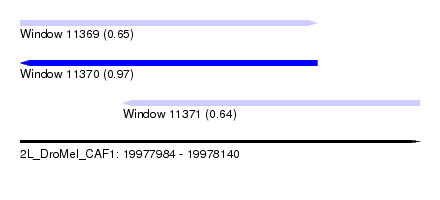
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 19,977,984 – 19,978,140 |
| Length | 156 |
| Max. P | 0.969728 |

| Location | 19,977,984 – 19,978,100 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 84.22 |
| Mean single sequence MFE | -28.40 |
| Consensus MFE | -20.80 |
| Energy contribution | -21.16 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.72 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.25 |
| SVM RNA-class probability | 0.652880 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 19977984 116 + 22407834 AACAAUGCCCUUGUCGAAAAUUGCAUUGUUUAGUUUCCAUCUGUCGUAUUAUGACAAAGACAUAGCUUAAUCUUUGCGGGAAGCCACAAAGGACAGUAGAGUGUAGAACAAUACAU ((((((((..((......))..)))))))).....((...(((((...(((((.......)))))......(((((.((....)).))))))))))..))(((((......))))) ( -26.10) >DroSec_CAF1 160887 116 + 1 AACAAUGCCCUUGUCGAAAAUUGCAUUGCUUAGUUUCCAUCUGUCGUAUUAUGACAGAGACAUUGCUCAGUCUUUGCGGGAAGCCACAAAGGACAGUAGAGUGUAGAACAAUGCAU .....................(((((((...........((((((((...)))))))).((((((((...((((((.((....)).))))))..))))..))))....))))))). ( -30.70) >DroSim_CAF1 163423 116 + 1 AACAAUGCCCUUGUCGAAAAUUGCAUUGUUUAGUUUCCAUCUGUCGUAUUAUGACAGAGACAUUGCUCAAUCUUUGUGGGAAGCCACAAAGCACAGUAGAGUGUAGAACAACGCAU ((((((((..((......))..))))))))..(((....((((((((...))))))))))).(((((....((((((((....))))))))...))))).((((......)))).. ( -33.40) >DroEre_CAF1 158977 109 + 1 AGCAAUGA-------GGCAAUUGCAUUGCUUAGUUUCCAUUUGUCGUAUUAUGACAGGGACAUUGCUCAAUCUUUGGGACAAGCCACAAGGGACAGUAGAGUAUAAAACAACACAU ((((((((-------((((((...)))))))....(((...((((((...)))))).))))))))))...((((((((.....)).))))))........................ ( -26.10) >DroYak_CAF1 159922 107 + 1 AACAAUGA---------CAAUGUCAUUGCUUAGUUUCUAUAUGUCGUAUUAUGACAUGGACAUUACUCAAUCUUUGCGACAAGCCACAAAGGACAGUUGAGUGUAGAACAACAAAG ..((((((---------(...)))))))(((.(((((((((((((((...)))))))).....(((((((((((((.(......).)))))....))))))))))))..))).))) ( -25.70) >consensus AACAAUGCCCUUGUCGAAAAUUGCAUUGCUUAGUUUCCAUCUGUCGUAUUAUGACAGAGACAUUGCUCAAUCUUUGCGGGAAGCCACAAAGGACAGUAGAGUGUAGAACAACACAU ((((((((..............))))))))..((((...((((((((...)))))))).((.(((((...((((((.((....)).))))))..))))).))...))))....... (-20.80 = -21.16 + 0.36)

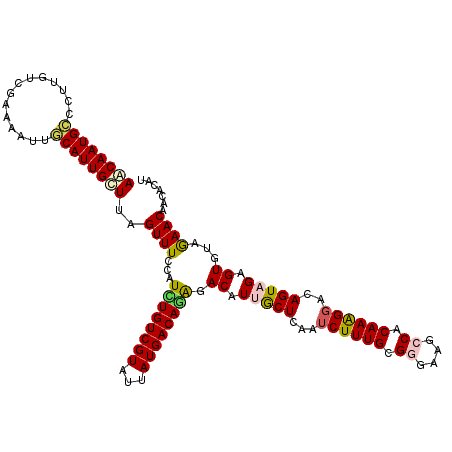
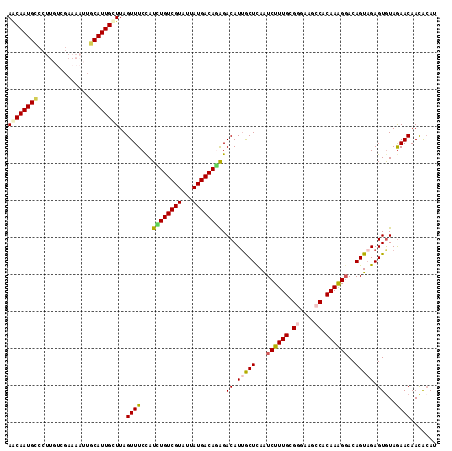
| Location | 19,977,984 – 19,978,100 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 84.22 |
| Mean single sequence MFE | -30.22 |
| Consensus MFE | -23.22 |
| Energy contribution | -22.42 |
| Covariance contribution | -0.80 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.45 |
| Structure conservation index | 0.77 |
| SVM decision value | 1.65 |
| SVM RNA-class probability | 0.969728 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 19977984 116 - 22407834 AUGUAUUGUUCUACACUCUACUGUCCUUUGUGGCUUCCCGCAAAGAUUAAGCUAUGUCUUUGUCAUAAUACGACAGAUGGAAACUAAACAAUGCAAUUUUCGACAAGGGCAUUGUU .(((((((((......(((((((((((((((((....)))))))).......((((.......))))....))))).)))).....)))))))))......(((((.....))))) ( -30.10) >DroSec_CAF1 160887 116 - 1 AUGCAUUGUUCUACACUCUACUGUCCUUUGUGGCUUCCCGCAAAGACUGAGCAAUGUCUCUGUCAUAAUACGACAGAUGGAAACUAAGCAAUGCAAUUUUCGACAAGGGCAUUGUU ..(((((((((.(((......))).((((((((....))))))))...))))))))).((((((.......)))))).(....)..((((((((..((......))..)))))))) ( -35.50) >DroSim_CAF1 163423 116 - 1 AUGCGUUGUUCUACACUCUACUGUGCUUUGUGGCUUCCCACAAAGAUUGAGCAAUGUCUCUGUCAUAAUACGACAGAUGGAAACUAAACAAUGCAAUUUUCGACAAGGGCAUUGUU ..(((((((((..(((......)))((((((((....))))))))...))))))))).((((((.......)))))).(....)..((((((((..((......))..)))))))) ( -38.30) >DroEre_CAF1 158977 109 - 1 AUGUGUUGUUUUAUACUCUACUGUCCCUUGUGGCUUGUCCCAAAGAUUGAGCAAUGUCCCUGUCAUAAUACGACAAAUGGAAACUAAGCAAUGCAAUUGCC-------UCAUUGCU .((((((((((.....((((.((((..((((((((((..(........)..))).......)))))))...))))..))))....))))))))))......-------........ ( -19.61) >DroYak_CAF1 159922 107 - 1 CUUUGUUGUUCUACACUCAACUGUCCUUUGUGGCUUGUCGCAAAGAUUGAGUAAUGUCCAUGUCAUAAUACGACAUAUAGAAACUAAGCAAUGACAUUG---------UCAUUGUU ........(((((.((((((.....((((((((....)))))))).)))))).......(((((.......))))).)))))....(((((((((...)---------)))))))) ( -27.60) >consensus AUGUGUUGUUCUACACUCUACUGUCCUUUGUGGCUUCCCGCAAAGAUUGAGCAAUGUCUCUGUCAUAAUACGACAGAUGGAAACUAAGCAAUGCAAUUUUCGACAAGGGCAUUGUU ...((((((((...((......)).((((((((....))))))))...))))))))((((((((.......)))))..))).....((((((((..............)))))))) (-23.22 = -22.42 + -0.80)

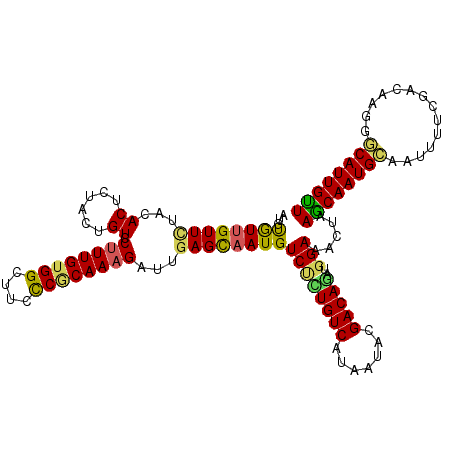
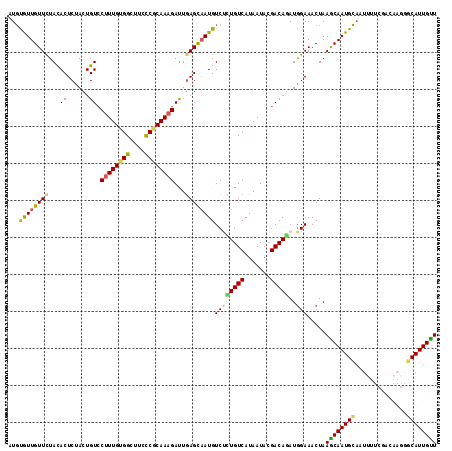
| Location | 19,978,024 – 19,978,140 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 85.95 |
| Mean single sequence MFE | -29.32 |
| Consensus MFE | -21.84 |
| Energy contribution | -22.64 |
| Covariance contribution | 0.80 |
| Combinations/Pair | 1.30 |
| Mean z-score | -1.69 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.21 |
| SVM RNA-class probability | 0.635222 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 19978024 116 - 22407834 CCUGUUCCUGUGGACUUUGUGCAAAAUUGUCAUUAGAUUAAUGUAUUGUUCUACACUCUACUGUCCUUUGUGGCUUCCCGCAAAGAUUAAGCUAUGUCUUUGUCAUAAUACGACAG ........((((((....(((((.....(((....)))...)))))...)))))).....(((((((((((((....)))))))).......((((.......))))....))))) ( -25.20) >DroSec_CAF1 160927 115 - 1 CCUGUUCCUGUGGACAGUGUGGAAAAA-GUCAUUAGAUUAAUGCAUUGUUCUACACUCUACUGUCCUUUGUGGCUUCCCGCAAAGACUGAGCAAUGUCUCUGUCAUAAUACGACAG ..(((((..(((((..((((((((...-..(((((...))))).....))))))))))))).(((.(((((((....)))))))))).)))))......(((((.......))))) ( -34.60) >DroSim_CAF1 163463 116 - 1 CCUGUUUCCGUGGACAUUGUGGAAAAAUGUCAUUAGAUUAAUGCGUUGUUCUACACUCUACUGUGCUUUGUGGCUUCCCACAAAGAUUGAGCAAUGUCUCUGUCAUAAUACGACAG .((((...((((((((((.......))))))....(((....(((((((((..(((......)))((((((((....))))))))...)))))))))....)))....)))))))) ( -35.40) >DroEre_CAF1 159010 116 - 1 CCUGAUUCUGUGGACAUUGUGGCAAAAUGUCAUUAGAUUAAUGUGUUGUUUUAUACUCUACUGUCCCUUGUGGCUUGUCCCAAAGAUUGAGCAAUGUCCCUGUCAUAAUACGACAA ..((((...(.((((((((((((((...(((((..(((....((((......))))......)))....)))))))))).((.....)).)))))))))).))))........... ( -23.90) >DroYak_CAF1 159953 112 - 1 ----GUCCUGUGGACAUUGUAAAAAAAUGUCAUUGGAUUACUUUGUUGUUCUACACUCAACUGUCCUUUGUGGCUUGUCGCAAAGAUUGAGUAAUGUCCAUGUCAUAAUACGACAU ----(((.((((((((((.......))))))..((((((((((.((((.........))))....((((((((....))))))))...)))))..)))))...))))....))).. ( -27.50) >consensus CCUGUUCCUGUGGACAUUGUGGAAAAAUGUCAUUAGAUUAAUGUGUUGUUCUACACUCUACUGUCCUUUGUGGCUUCCCGCAAAGAUUGAGCAAUGUCUCUGUCAUAAUACGACAG ...........(((((((((((((.((((.(((((...))))))))).)))))).(((....((.((((((((....)))))))))).))).)))))))(((((.......))))) (-21.84 = -22.64 + 0.80)

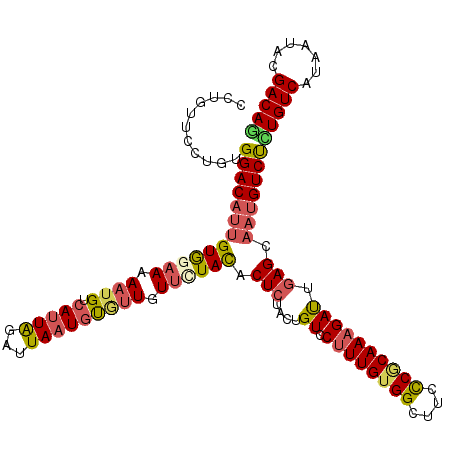
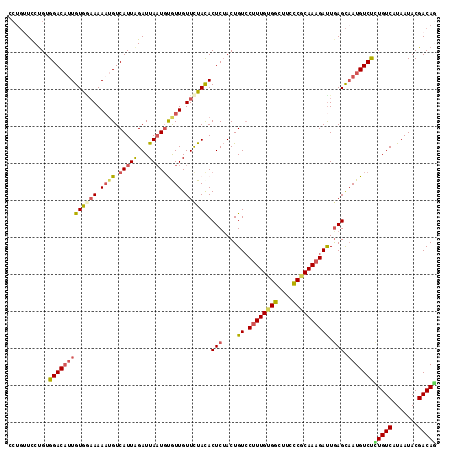
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:28:02 2006