
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 19,591,650 – 19,591,777 |
| Length | 127 |
| Max. P | 0.746469 |

| Location | 19,591,650 – 19,591,741 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 92 |
| Reading direction | forward |
| Mean pairwise identity | 77.69 |
| Mean single sequence MFE | -27.05 |
| Consensus MFE | -17.18 |
| Energy contribution | -17.43 |
| Covariance contribution | 0.25 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.45 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.34 |
| SVM RNA-class probability | 0.694753 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
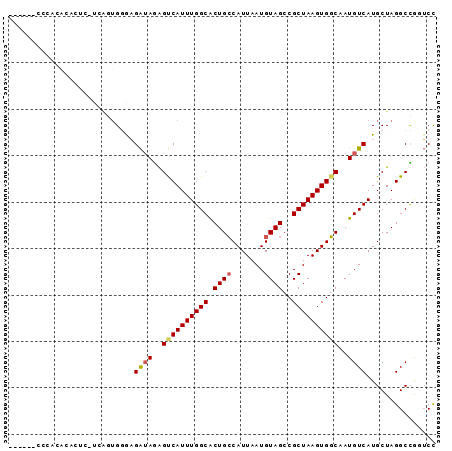
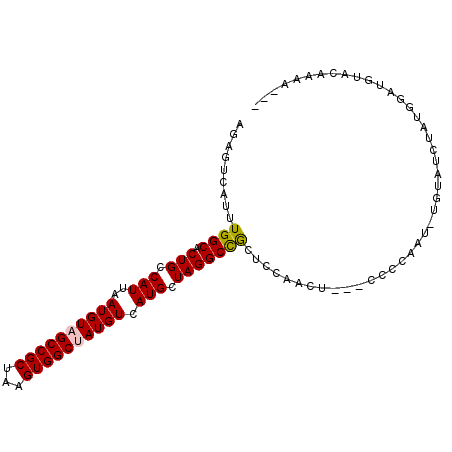
>2L_DroMel_CAF1 19591650 91 + 22407834 GACACACCCACACACUC-UCAGUGGAAGAUAGAGUCAUUUGGCACUGCCAUUAAUGUAGCCGCUAAGUGGCUAUGUCAUGCUAGGCCGCUCC .......((((......-...))))..((((.(((((((((((.((((.......))))..))))))))))).))))..((......))... ( -26.10) >DroEre_CAF1 12818 85 + 1 ------CCCACACACUC-UCGGUGGAAGAUAGAGUCAUUUGGCACUGCCAUUAAUGUAGCCGCUAAGUGGCUAUGUCAUGCUAGGCCGGUCC ------...........-(((((((..((((.(((((((((((.((((.......))))..))))))))))).))))...))..)))))... ( -26.50) >DroWil_CAF1 25261 91 + 1 GAGACAGUGAGAGUGAG-UGAAUGAGAGAGAAAGCCAUUUGGCACUGCCAUUAAUGUAGCCGCUAAGUGGCAAUGUCAUGCUAGGCUAGUCC ..((((((.((.((((.-....(....).....((((((((((.((((.......))))..))))))))))....)))).))..))).))). ( -26.20) >DroYak_CAF1 12843 85 + 1 ------CCCACACACUC-UCAGUGGGAGAUAGAGUCAUUUGGCACUGCCAUUAAUGUAGCCGCUAAGUGGCUAUGUCAUGCUAGGCCGGUCC ------(((((......-...))))).((((.(((((((((((.((((.......))))..))))))))))).))))............... ( -28.50) >DroMoj_CAF1 21799 77 + 1 ----------GGGAGUG-----UGGGCGGUAGGGCCAUUUGGCGCUGGCAUUAAUGUAGCCGCUAAGUGCCAAUGUCAUGCUAGGCUAGGCC ----------....((.-----(((.(((((.(((...(((((((((((.........)))....)))))))).))).)))).).))).)). ( -25.90) >DroAna_CAF1 12667 83 + 1 ---------AUGGAGUCGUGGGAGUGAGAUAGAGUCAUUUGGCACUGCCAUUAAUGUAGCCGCUAAGUGGCAAUGUCAUGCUAGGCUGCUCU ---------..(((((.((...((((.((((..((((((((((.((((.......))))..))))))))))..)))).))))..)).))))) ( -29.10) >consensus ______CCCACACACUC_UCAGUGGGAGAUAGAGUCAUUUGGCACUGCCAUUAAUGUAGCCGCUAAGUGGCAAUGUCAUGCUAGGCCGGUCC ...........................((((..((((((((((.((((.......))))..))))))))))..))))............... (-17.18 = -17.43 + 0.25)


| Location | 19,591,679 – 19,591,777 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 101 |
| Reading direction | forward |
| Mean pairwise identity | 82.01 |
| Mean single sequence MFE | -25.48 |
| Consensus MFE | -18.09 |
| Energy contribution | -18.07 |
| Covariance contribution | -0.03 |
| Combinations/Pair | 1.10 |
| Mean z-score | -1.89 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.46 |
| SVM RNA-class probability | 0.746469 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
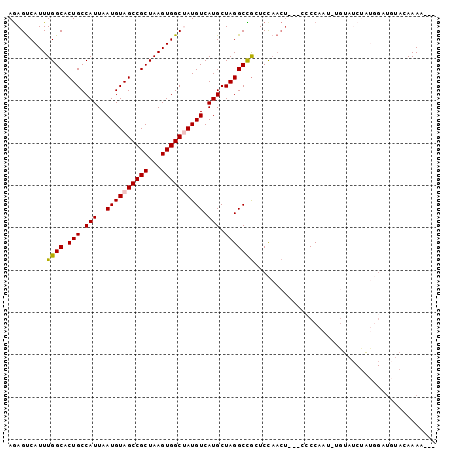
>2L_DroMel_CAF1 19591679 98 + 22407834 AGAGUCAUUUGGCACUGCCAUUAAUGUAGCCGCUAAGUGGCUAUGUCAUGCUAGGCCGCUCCAACU---CCCCAAUUUGUAUCUAUGGAUGUACAAAAAAA .((((....((((.(((.(((..((((((((((...)))))))))).))).))))))).....)))---).....(((((((........))))))).... ( -26.80) >DroSim_CAF1 12720 95 + 1 AGAGUCAUUUGGCACUGCCAUUAAUGUAGCCGCUAAGUGGCUAUGUCAUGCUAGGCCGCUCCAACU---CCCCAAUUUGUAUCUAUGGCUGUAUAAAA--- .((((....((((.(((.(((..((((((((((...)))))))))).))).))))))).....)))---).....(((((((........))))))).--- ( -24.90) >DroEre_CAF1 12841 95 + 1 AGAGUCAUUUGGCACUGCCAUUAAUGUAGCCGCUAAGUGGCUAUGUCAUGCUAGGCCGGUCCAACU---CCCCAAUAUGUAUCUAUGGAUGUAUAAAA--- .((((...(((((.(((.(((..((((((((((...)))))))))).))).))))))))....)))---).....((((((((....))))))))...--- ( -27.80) >DroWil_CAF1 25290 82 + 1 AAAGCCAUUUGGCACUGCCAUUAAUGUAGCCGCUAAGUGGCAAUGUCAUGCUAGGCUAGUCC---------CCACU--GUAACCGCAGC-----AAAA--- ...((((((((((.((((.......))))..)))))))))).......((((.((.((((..---------..)))--)...))..)))-----)...--- ( -25.40) >DroYak_CAF1 12866 95 + 1 AGAGUCAUUUGGCACUGCCAUUAAUGUAGCCGCUAAGUGGCUAUGUCAUGCUAGGCCGGUCCAACU---CCCCAAUAUGUAUCUAUGGAUGUAAAAAA--- .((((...(((((.(((.(((..((((((((((...)))))))))).))).))))))))....)))---).........((((....)))).......--- ( -25.40) >DroAna_CAF1 12688 86 + 1 AGAGUCAUUUGGCACUGCCAUUAAUGUAGCCGCUAAGUGGCAAUGUCAUGCUAGGCUGCUCUAACUGUGCCCCAUU-CC----------UU--CAAAAA-- .(((..((..(((((.....(((..((((((.....(((((...)))))....))))))..)))..)))))..)).-.)----------))--......-- ( -22.60) >consensus AGAGUCAUUUGGCACUGCCAUUAAUGUAGCCGCUAAGUGGCUAUGUCAUGCUAGGCCGCUCCAACU___CCCCAAU_UGUAUCUAUGGAUGUACAAAA___ .........((((.(((.(((..((((((((((...)))))))))).))).)))))))........................................... (-18.09 = -18.07 + -0.03)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:25:33 2006