
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 19,461,106 – 19,461,213 |
| Length | 107 |
| Max. P | 0.985969 |

| Location | 19,461,106 – 19,461,213 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 76.76 |
| Mean single sequence MFE | -52.37 |
| Consensus MFE | -38.72 |
| Energy contribution | -38.53 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.57 |
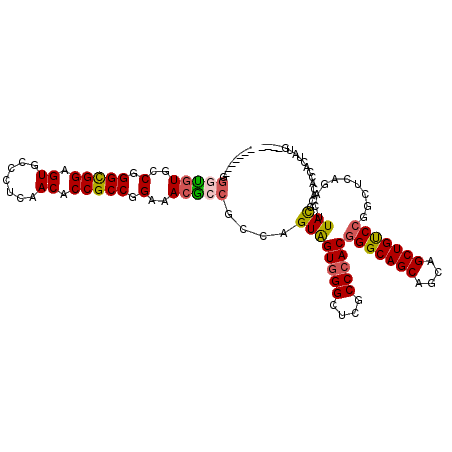
| Structure conservation index | 0.74 |
| SVM decision value | 2.03 |
| SVM RNA-class probability | 0.985969 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
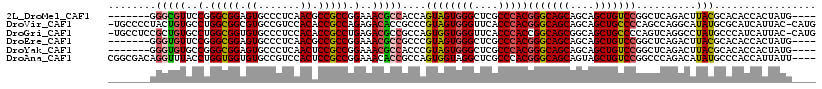
>2L_DroMel_CAF1 19461106 107 + 22407834 -------GGGCGUUCCGGGCGGAGUGCCCUCAACGCCGCCGGAAACGCCACCAGUAGUGGGCUCGCCCACGGGCAGCAGCAGCUGUCCGGCUCAGACUUACGCACACCACUAUG---- -------.((((((((((.(((.((.......)).))))))).))))))....(((((((.........((((((((....))))))))((..........))...))))))).---- ( -49.70) >DroVir_CAF1 19882 116 + 1 -UGCCCCUACUGUGCCUGGCGGCGUGCCGUCCACACCGCCAGAGACACCGCCCGUAGUGGGUUCACCCACGGGCAGCAGCAGCUGCCCCAGCCAGGCAUAUGCGCAUCAUUAC-CAUG -.........((((((((((((.(((.....))).))))))).......(((....(((((....)))))(((((((....)))))))......)))....))))).......-.... ( -52.40) >DroGri_CAF1 17839 116 + 1 -UGCCUCCGCUGUGCCUGGCGGUGUGCCCUCCACACCGCCUGAGACGCCGCCAGUGGUGGGUUCACCCACCGGCAGCGGCAGCUGCCCCAGUCAGGCCUAUGCCCAUCAUUAC-CAUG -.......((((.(((((((((((((.....)))))))))...(((.........((((((....))))))((((((....))))))...))))))).)).))..........-.... ( -52.70) >DroEre_CAF1 16903 107 + 1 -------GGGUGUUCCGGGCGGAGUGCCCUCAACGCCGCCGGAAACGCCGCCCGUAGUGGGCUCGCCCACGGGCAGCAGCAGCUGUCCGGCUCAGACUUACGCACACCACUAUG---- -------.((((((((((.(((.((.......)).))))))).))))))(((....(((((....)))))(((((((....)))))))))).......................---- ( -49.80) >DroYak_CAF1 17831 107 + 1 -------GGGUGUGCCGGGCGGAGUGCCCUCAACUCCGCCGGAAACGCCACCCGUAGUGGGCUCGCCCACGGGCAGCAGCAGCUGUCCGGCUCAGACUUACGCACACCACUAUG---- -------.(((((((..((((((((.......))))))))(....)(((.......(((((....)))))(((((((....))))))))))..........)))))))......---- ( -59.20) >DroAna_CAF1 16177 114 + 1 CGGCGACAGGUUUACCUGGUGGUGUGCCGUCCACUCCGCCGGAAACACCGCCAGUGGUAGGCUCGCCCACGGGCAGCAGUAGCUGUCCGGCCCAGACAUAUGCCCACCAUUAUU---- .((((....((((.((.(((((.(((.....))).)))))))))))..))))((((((.(((((.....((((((((....)))))))).....)).....))).))))))...---- ( -50.40) >consensus _______GGGUGUGCCGGGCGGAGUGCCCUCAACACCGCCGGAAACGCCGCCAGUAGUGGGCUCGCCCACGGGCAGCAGCAGCUGUCCGGCUCAGACUUACGCACACCACUAUG____ ........(((((..(.(((((.((.......)).))))).)..)))))....((((((((....)))))(((((((....)))))))..........)))................. (-38.72 = -38.53 + -0.19)
| Location | 19,461,106 – 19,461,213 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 76.76 |
| Mean single sequence MFE | -55.80 |
| Consensus MFE | -39.19 |
| Energy contribution | -38.75 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.27 |
| Mean z-score | -2.04 |
| Structure conservation index | 0.70 |
| SVM decision value | 1.65 |
| SVM RNA-class probability | 0.969936 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
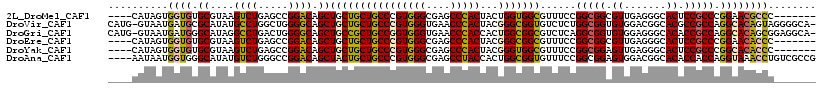
>2L_DroMel_CAF1 19461106 107 - 22407834 ----CAUAGUGGUGUGCGUAAGUCUGAGCCGGACAGCUGCUGCUGCCCGUGGGCGAGCCCACUACUGGUGGCGUUUCCGGCGGCGUUGAGGGCACUCCGCCCGGAACGCCC------- ----..((((((((.((....(((((...(((.((((....)))).))))))))..)).))))))))..((((((((.(((((.((.......)).))))).)))))))).------- ( -51.00) >DroVir_CAF1 19882 116 - 1 CAUG-GUAAUGAUGCGCAUAUGCCUGGCUGGGGCAGCUGCUGCUGCCCGUGGGUGAACCCACUACGGGCGGUGUCUCUGGCGGUGUGGACGGCACGCCGCCAGGCACAGUAGGGGCA- (((.-...))).(((.(.((((((((....(((((((....)))))))(((((....)))))..))))).((((..(((((((((((.....))))))))))))))).))).).)))- ( -65.40) >DroGri_CAF1 17839 116 - 1 CAUG-GUAAUGAUGGGCAUAGGCCUGACUGGGGCAGCUGCCGCUGCCGGUGGGUGAACCCACCACUGGCGGCGUCUCAGGCGGUGUGGAGGGCACACCGCCAGGCACAGCGGAGGCA- (((.-...)))...((((...((((.....))))...))))((((((((((((....))))))...))))))(((((.(((((((((.....)))))))))..((...)).))))).- ( -62.60) >DroEre_CAF1 16903 107 - 1 ----CAUAGUGGUGUGCGUAAGUCUGAGCCGGACAGCUGCUGCUGCCCGUGGGCGAGCCCACUACGGGCGGCGUUUCCGGCGGCGUUGAGGGCACUCCGCCCGGAACACCC------- ----......(((((.(....)((((.((((((.(((....((((((((((((....)))))...))))))))))))))))((((..(((....)))))))))))))))).------- ( -52.50) >DroYak_CAF1 17831 107 - 1 ----CAUAGUGGUGUGCGUAAGUCUGAGCCGGACAGCUGCUGCUGCCCGUGGGCGAGCCCACUACGGGUGGCGUUUCCGGCGGAGUUGAGGGCACUCCGCCCGGCACACCC------- ----......(((((((.(((.(((..((((((.(((....((..((((((((....)))))...)))..)))))))))))))).))).((((.....)))).))))))).------- ( -55.30) >DroAna_CAF1 16177 114 - 1 ----AAUAAUGGUGGGCAUAUGUCUGGGCCGGACAGCUACUGCUGCCCGUGGGCGAGCCUACCACUGGCGGUGUUUCCGGCGGAGUGGACGGCACACCACCAGGUAAACCUGUCGCCG ----.....((((((((...((((((...(((.((((....)))).))))))))).))))))))..((((((((..(((..........))).)))))..((((....))))..))). ( -48.00) >consensus ____CAUAAUGGUGUGCAUAAGUCUGAGCCGGACAGCUGCUGCUGCCCGUGGGCGAGCCCACUACGGGCGGCGUUUCCGGCGGAGUGGAGGGCACUCCGCCAGGCACACCC_______ ..........((((.((....((((.....)))).))((((((((((((((((....)))))...)))))))......(((((.((.......)).))))).))))))))........ (-39.19 = -38.75 + -0.44)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:24:49 2006