
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 19,319,585 – 19,319,705 |
| Length | 120 |
| Max. P | 0.999989 |

| Location | 19,319,585 – 19,319,705 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.16 |
| Mean single sequence MFE | -49.14 |
| Consensus MFE | -46.70 |
| Energy contribution | -46.66 |
| Covariance contribution | -0.04 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.37 |
| Structure conservation index | 0.95 |
| SVM decision value | 5.55 |
| SVM RNA-class probability | 0.999989 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
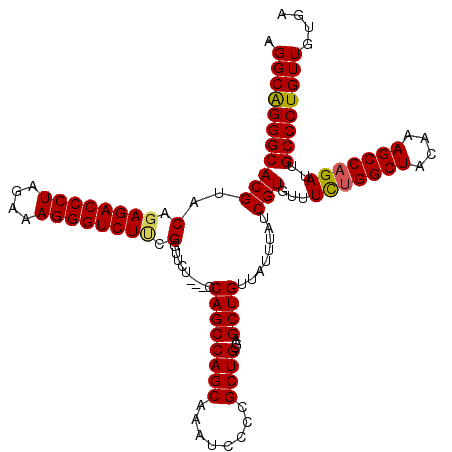
>2L_DroMel_CAF1 19319585 120 + 22407834 AGGCAGGGCACGUACAGAGACCCUAGAAAGGGUCUUCGGUUCUCCUCCAGCCAGCAAAUCCCCGCUGCGAGCUGUUAUUUAUCGUGUUUCUGGCUACAAAGCCAGAUUGCCCUGUUGUGA .(((((((((...(((((((((((....))))))))((((.......((((((((........))))...))))......))))))).(((((((....))))))).))))))))).... ( -48.12) >DroSec_CAF1 12168 116 + 1 AGGCAGGGCACGUACGGAGACCCUAGAAAGGGUCUUCGGUUCU---CCAGCCAGCAAAUCCCCGCUGCGAGCUGUUAUUUAUCGUGU-UCUGGCUACAAAGCCAGAUUGCCCUGUUGUGA .(((((((((((..((((((((((....)))))))))).....---.((((((((........))))...))))........)))..-(((((((....)))))))..)))))))).... ( -52.70) >DroSim_CAF1 13997 117 + 1 AGGCGGGGCACGUACGGAGACCCUAGAAAGGGUCUUCGGUUCU---CCAGCCAGCAAAUCCCCGCUGCGAGCUGUUAUUUAUCGUGUUUCUGGCUACAAAGCCAGAUUGCCCUGUUGUGA .(((((((((((..((((((((((....)))))))))).....---.((((((((........))))...))))........)))...(((((((....)))))))..)))))))).... ( -52.00) >DroEre_CAF1 11303 116 + 1 AGGCAGGGCACGUACAGAGACCCUAGA-AGGGUCUGCGGAUCC---CCAGCCAGCCAAUCCCCGCUGCGAGCUGUUAUUUAUCGUGUUUUGGGCUGCAAAGCCAGAUUGCCCUGUUGUGA .((((((((.(((....((((((....-.)))))))))((((.---.((((((((........))))...)))).................((((....)))).)))))))))))).... ( -42.60) >DroYak_CAF1 12449 117 + 1 AGGCAGGGCACGUACAGAGACCCUAGAAAGGGUCUCCGAAUCU---CCAGCCAGCAAAUCCCCGCUGCGAGCUGUUAUUUAUCGUGUUUCUGGCUGCAAAGCCAGAUUGCCCUGUUGUGA .(((((((((..(((.((((((((....)))))))).......---.((((((((........))))...)))).........)))..(((((((....))))))).))))))))).... ( -50.30) >consensus AGGCAGGGCACGUACAGAGACCCUAGAAAGGGUCUUCGGUUCU___CCAGCCAGCAAAUCCCCGCUGCGAGCUGUUAUUUAUCGUGUUUCUGGCUACAAAGCCAGAUUGCCCUGUUGUGA .(((((((((((..(.((((((((....)))))))).).........((((((((........))))...))))........)))...(((((((....)))))))..)))))))).... (-46.70 = -46.66 + -0.04)

| Location | 19,319,585 – 19,319,705 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.16 |
| Mean single sequence MFE | -48.26 |
| Consensus MFE | -44.24 |
| Energy contribution | -44.80 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.03 |
| Mean z-score | -3.37 |
| Structure conservation index | 0.92 |
| SVM decision value | 5.03 |
| SVM RNA-class probability | 0.999970 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
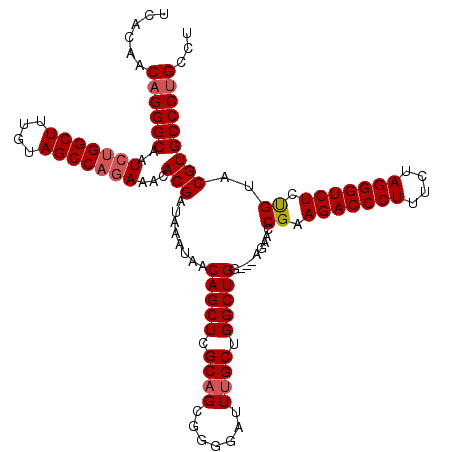
>2L_DroMel_CAF1 19319585 120 - 22407834 UCACAACAGGGCAAUCUGGCUUUGUAGCCAGAAACACGAUAAAUAACAGCUCGCAGCGGGGAUUUGCUGGCUGGAGGAGAACCGAAGACCCUUUCUAGGGUCUCUGUACGUGCCCUGCCU ......(((((((.(((((((....))))))).............(((((((.((((.((......)).))))))).........(((((((....)))))))))))...)))))))... ( -49.80) >DroSec_CAF1 12168 116 - 1 UCACAACAGGGCAAUCUGGCUUUGUAGCCAGA-ACACGAUAAAUAACAGCUCGCAGCGGGGAUUUGCUGGCUGG---AGAACCGAAGACCCUUUCUAGGGUCUCCGUACGUGCCCUGCCU ......((((((..(((((((....)))))))-..(((...........(((.((((.((......)).)))))---))...((.(((((((....))))))).))..)))))))))... ( -50.30) >DroSim_CAF1 13997 117 - 1 UCACAACAGGGCAAUCUGGCUUUGUAGCCAGAAACACGAUAAAUAACAGCUCGCAGCGGGGAUUUGCUGGCUGG---AGAACCGAAGACCCUUUCUAGGGUCUCCGUACGUGCCCCGCCU ........((((..(((((((....)))))))..((((...........(((.((((.((......)).)))))---))...((.(((((((....))))))).))..))))....)))) ( -46.60) >DroEre_CAF1 11303 116 - 1 UCACAACAGGGCAAUCUGGCUUUGCAGCCCAAAACACGAUAAAUAACAGCUCGCAGCGGGGAUUGGCUGGCUGG---GGAUCCGCAGACCCU-UCUAGGGUCUCUGUACGUGCCCUGCCU ......(((((((....((((....))))................((((......(((((..(..(....)..)---...)))))((((((.-....))))))))))...)))))))... ( -40.60) >DroYak_CAF1 12449 117 - 1 UCACAACAGGGCAAUCUGGCUUUGCAGCCAGAAACACGAUAAAUAACAGCUCGCAGCGGGGAUUUGCUGGCUGG---AGAUUCGGAGACCCUUUCUAGGGUCUCUGUACGUGCCCUGCCU ......((((((..(((((((....)))))))...(((...........(((.((((.((......)).)))))---))...((((((((((....))))))))))..)))))))))... ( -54.00) >consensus UCACAACAGGGCAAUCUGGCUUUGUAGCCAGAAACACGAUAAAUAACAGCUCGCAGCGGGGAUUUGCUGGCUGG___AGAACCGAAGACCCUUUCUAGGGUCUCUGUACGUGCCCUGCCU ......((((((..(((((((....)))))))...(((........(((((.((((.......)))).))))).........((.(((((((....))))))).))..)))))))))... (-44.24 = -44.80 + 0.56)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:23:48 2006