
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 19,273,253 – 19,273,401 |
| Length | 148 |
| Max. P | 0.756847 |

| Location | 19,273,253 – 19,273,369 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 89.89 |
| Mean single sequence MFE | -43.77 |
| Consensus MFE | -33.74 |
| Energy contribution | -33.42 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.20 |
| Mean z-score | -2.52 |
| Structure conservation index | 0.77 |
| SVM decision value | -0.06 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 19273253 116 - 22407834 AAAGUGGGUGGCAGUGGGAGGU---GGUGCGUGUGCCAAAGCAAUUAUUUCCCGCUGGCAGCAUUCUAAAUUGAGGCUGCUAAUAGAGCCAGAGAUGGCCG-GGAGCAAAGCUCAGUUAG .((.(((((.((((((((((((---(((((..........)).)))))))))))))((((((.(((......)))))))))....(.((((....))))).-...))...))))).)).. ( -44.50) >DroSec_CAF1 14499 116 - 1 AAAGUGGGUGGCAGUGGGAGGU---GGCGCGUGUGCCAAAGCAAUUAUUUCCCGCUGGCAGCAUUCUAAAUUGAGGCUGCUAAUAGAGCCAGAGAUGGCCG-GGAGCAAAGCUCAGUUAG .((.(((((.((((((((((((---((((....)))).((....))))))))))))((((((.(((......)))))))))....(.((((....))))).-...))...))))).)).. ( -44.10) >DroSim_CAF1 21380 116 - 1 AAAGUGGGUGGCAGUGGGAGGU---GGCGCGUGUGCCAAAGCAAUUAUUUCCCGCUGGCAGCAUUCUAAAUUGAGGCUGCUAAUAGAGCCAGAGAUGGCCG-GGAGCAAAGCUCAGUUAG .((.(((((.((((((((((((---((((....)))).((....))))))))))))((((((.(((......)))))))))....(.((((....))))).-...))...))))).)).. ( -44.10) >DroEre_CAF1 21200 116 - 1 UGGGCGGUGGGCGGUGGGUGGU---GGUGUGUGUGCCAAAGCAAUUAUUUCCCGCUGGCAGCAUUCUAAAUUGAGGCUGCUAAUAGAGCCAGAGAUGGCCG-GGAGCAGAGCUCAGUUAG ..(.(((((((..(((.((..(---(((......))))..))...)))..))))))).).........(((((((.(((((....(.((((....))))).-..)))))..))))))).. ( -44.50) >DroYak_CAF1 20586 116 - 1 AAAGUGGGUGGCGGUGGGUGGU---GGCGUGUGUGCCAAAGCAAUUAUUUCCCGCUGGCAGCAUUCUAAAUUGAGGCUGCUAAUAGAGCCAUAGAUGGCCG-GGAGCAAAGCUCAGUUAG .((.(((((.((.....((..(---((((....)))))..)).......(((((.(((((((.(((......)))))))))).....((((....))))))-)))))...))))).)).. ( -43.10) >DroAna_CAF1 36821 120 - 1 UGGAGGGGCCGGCGUGGGUGGUUGAGGCGCGUGUGCCAAAGCAAUUAUUUCUCGCUGGCAGCAUUCUAAAUUGAAGCUGCUAAUAGAGCCCGAAAAGCCCGAGGACCCAAGCUCAGUUGC ..((((((((....(((((..(((.((((((..(((....))).........)))(((((((.(((......)))))))))).....))))))...))))).)).)))...)))...... ( -42.30) >consensus AAAGUGGGUGGCAGUGGGAGGU___GGCGCGUGUGCCAAAGCAAUUAUUUCCCGCUGGCAGCAUUCUAAAUUGAGGCUGCUAAUAGAGCCAGAGAUGGCCG_GGAGCAAAGCUCAGUUAG .((.(((((.((.(((((.......((((....)))).((....))....)))))(((((((.(((......))))))))))...(.((((....))))).....))...))))).)).. (-33.74 = -33.42 + -0.33)

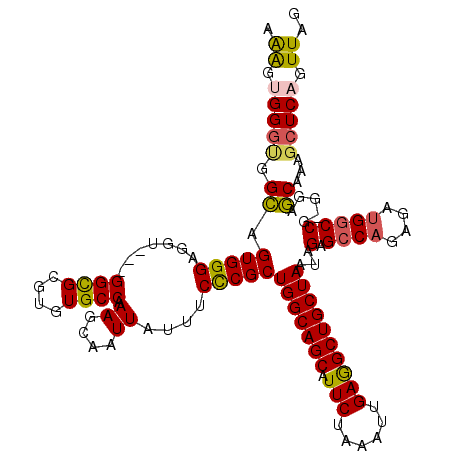
| Location | 19,273,292 – 19,273,401 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.25 |
| Mean single sequence MFE | -38.68 |
| Consensus MFE | -23.67 |
| Energy contribution | -23.78 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.27 |
| Mean z-score | -1.54 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.49 |
| SVM RNA-class probability | 0.756847 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 19273292 109 - 22407834 UUGGCAGU--------GGCAGUUGGCAGUUGGGUGGCCUGAAAGUGGGUGGCAGUGGGAGGU---GGUGCGUGUGCCAAAGCAAUUAUUUCCCGCUGGCAGCAUUCUAAAUUGAGGCUGC ...(((((--------..(((((.......(((((((((......))))(.(((((((((((---(((((..........)).)))))))))))))).)..)))))..)))))..))))) ( -41.10) >DroSec_CAF1 14538 117 - 1 UUGGCAGUUGGCAGUUGGCAGUUGGCAGUUGGGUGGCCUGAAAGUGGGUGGCAGUGGGAGGU---GGCGCGUGUGCCAAAGCAAUUAUUUCCCGCUGGCAGCAUUCUAAAUUGAGGCUGC (..((.((..((........))..)).))..)(..((((.((..((((((.(((((((((((---((((....)))).((....))))))))))))).))....))))..)).))))..) ( -45.10) >DroSim_CAF1 21419 117 - 1 UUGGCAGUUGGCAGUUGGCAGUUGGCAGUUGGGUGGCCUGAAAGUGGGUGGCAGUGGGAGGU---GGCGCGUGUGCCAAAGCAAUUAUUUCCCGCUGGCAGCAUUCUAAAUUGAGGCUGC (..((.((..((........))..)).))..)(..((((.((..((((((.(((((((((((---((((....)))).((....))))))))))))).))....))))..)).))))..) ( -45.10) >DroEre_CAF1 21239 102 - 1 UUGG------GGCGUUGGGAGAU-----GUGGGUGG----UGGGCGGUGGGCGGUGGGUGGU---GGUGUGUGUGCCAAAGCAAUUAUUUCCCGCUGGCAGCAUUCUAAAUUGAGGCUGC ..(.------(((.(..(.((((-----((.....(----(.(((((..((..((..((..(---(((......))))..)).))..))..))))).)).))).)))...)..).))).) ( -33.60) >DroYak_CAF1 20625 111 - 1 UGGA------AGCGUUGUGAGAUCGCAGAUGGGUGGCUCGAAAGUGGGUGGCGGUGGGUGGU---GGCGUGUGUGCCAAAGCAAUUAUUUCCCGCUGGCAGCAUUCUAAAUUGAGGCUGC ....------.......((((.((((......))))))))..((((((..(..((..((..(---((((....)))))..)).))..)..)))))).(((((.(((......)))))))) ( -35.90) >DroAna_CAF1 36861 95 - 1 UGGAUG-------------------------GAUGGAUUUUGGAGGGGCCGGCGUGGGUGGUUGAGGCGCGUGUGCCAAAGCAAUUAUUUCUCGCUGGCAGCAUUCUAAAUUGAAGCUGC .....(-------------------------(......(((((((..(((((((.(((((((((.((((....))))....)))))))))..)))))))....)))))))......)).. ( -31.30) >consensus UUGGCA____G__GUUGGCAGUUGGCAGUUGGGUGGCCUGAAAGUGGGUGGCAGUGGGAGGU___GGCGCGUGUGCCAAAGCAAUUAUUUCCCGCUGGCAGCAUUCUAAAUUGAGGCUGC ................................((.((((......)))).))((((((.......((((....)))).((....))....)))))).(((((.(((......)))))))) (-23.67 = -23.78 + 0.12)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:22:43 2006