
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 18,715,938 – 18,716,093 |
| Length | 155 |
| Max. P | 0.984609 |

| Location | 18,715,938 – 18,716,053 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.92 |
| Mean single sequence MFE | -39.18 |
| Consensus MFE | -32.38 |
| Energy contribution | -33.06 |
| Covariance contribution | 0.68 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.23 |
| Structure conservation index | 0.83 |
| SVM decision value | 1.98 |
| SVM RNA-class probability | 0.984609 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
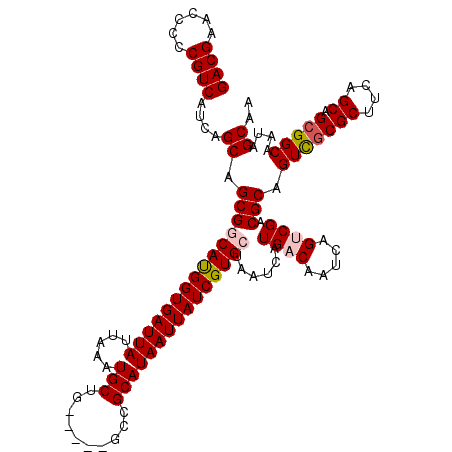
>2L_DroMel_CAF1 18715938 115 + 22407834 GACGAACCUUCGUCAUCAGCAGCGGCAUGGUGAUUAUUAAAUGCUG-----GACGCAUAAUUAUCGUGCUAAUCAUGACAAUCAGUCGACGCAGUCGCGCUUCAGCAGCGGCAAUAGCAA (((((....)))))....((.(((((((((((((((.....(((..-----...)))))))))))))))......((((.....)))).))).(((((((....)).)))))....)).. ( -39.90) >DroSec_CAF1 8787 120 + 1 GACGAACCCCCGUCAUCAGCAGCGGCAUGGUGAUUAUUAAAUGCUGGCCUGGCCGCAUAAUUAUCGUGCUAAUCAUGACAAUCAGUCGACGCAGUCGCGCUUCAGCAGCGGCAAUAGCAA ((((......))))....((.(((((((((((((((.....(((.(((...))))))))))))))))))......((((.....)))).))).(((((((....)).)))))....)).. ( -42.70) >DroSim_CAF1 7151 120 + 1 GACGAACCCCCGUCAUCAGCAGCGGCAUGGUGAUUAUUAAAUGCUGGCCUGGCCGCAUAAUUAUCGUGCUAAUCAUGACAAUCAGUCGACGCAGUCGCGCUUCAGCAGCGGCAAUAGCAA ((((......))))....((.(((((((((((((((.....(((.(((...))))))))))))))))))......((((.....)))).))).(((((((....)).)))))....)).. ( -42.70) >DroEre_CAF1 8792 115 + 1 GACGAACCUCCGUCAUCAGCAGCGGCAUGGUGAUUAUUAAAUGCUG-----GCCGCAUAAUUAUCGUGAUAAUCAUGACAAUCAGGCGACGCAGUCGCGCUUCAGCAGCGGCAAUAGCAA ((((......))))....((..((((((............))))))-----(((((.......(((((.....))))).......(((((...)))))((....)).)))))....)).. ( -36.80) >DroYak_CAF1 8764 115 + 1 GACGAACCCCCGUCAUCAGCAGCGCCACGGUGAUUAUUAAAUGCUG-----GCCGCAUAAUUAUCGUGAUAAUCAUGACAAUCAGGCGACGCAGUUUCGCUUCAGCAGCGGCAAUAGCAA ((((......))))..(((((.(((....))).........)))))-----(((((.......(((((.....))))).....((((((.......)))))).....)))))........ ( -33.80) >consensus GACGAACCCCCGUCAUCAGCAGCGGCAUGGUGAUUAUUAAAUGCUG_____GCCGCAUAAUUAUCGUGCUAAUCAUGACAAUCAGUCGACGCAGUCGCGCUUCAGCAGCGGCAAUAGCAA ((((......))))....((.(((((((((((((((.....(((..........)))))))))))))))......((((.....)))).))).(((((((....)).)))))....)).. (-32.38 = -33.06 + 0.68)

| Location | 18,715,978 – 18,716,093 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.92 |
| Mean single sequence MFE | -39.38 |
| Consensus MFE | -30.82 |
| Energy contribution | -31.46 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.15 |
| Structure conservation index | 0.78 |
| SVM decision value | 1.02 |
| SVM RNA-class probability | 0.901486 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18715978 115 - 22407834 CUGGUUGUGGUUUUGUUGCGCUUGAUUAUGCUUUUGGCUCUUGCUAUUGCCGCUGCUGAAGCGCGACUGCGUCGACUGAUUGUCAUGAUUAGCACGAUAAUUAUGCGUC-----CAGCAU ((((.((((...((((((.(((.((((((((..(..(.((.(((..((((.(((.....)))))))..)))..)))..)..).)))))))))).))))))...)))).)-----)))... ( -37.00) >DroSec_CAF1 8827 120 - 1 CUGGUUGUGGUUUUGUUGCGCUUGAUUAUGCUUUUGGCUCUUGCUAUUGCCGCUGCUGAAGCGCGACUGCGUCGACUGAUUGUCAUGAUUAGCACGAUAAUUAUGCGGCCAGGCCAGCAU (((((((((...((((((.(((.((((((((..(..(.((.(((..((((.(((.....)))))))..)))..)))..)..).)))))))))).))))))...)))))))))........ ( -42.00) >DroSim_CAF1 7191 120 - 1 CUGGUUGUGGUUUUGUUGCGCUUGAUUAUGCUUUUGGCUCUUGCUAUUGCCGCUGCUGAAGCGCGACUGCGUCGACUGAUUGUCAUGAUUAGCACGAUAAUUAUGCGGCCAGGCCAGCAU (((((((((...((((((.(((.((((((((..(..(.((.(((..((((.(((.....)))))))..)))..)))..)..).)))))))))).))))))...)))))))))........ ( -42.00) >DroEre_CAF1 8832 115 - 1 UUGGUUGUGGUUUAGUUGCGCUUGAUUAUGCUUUUCGCUCUUGCUAUUGCCGCUGCUGAAGCGCGACUGCGUCGCCUGAUUGUCAUGAUUAUCACGAUAAUUAUGCGGC-----CAGCAU ..(((((((.((((((.((((........)).....((....)).......)).)))))).)))))))(((((((.((((((((.((.....)).)))))))).)))))-----..)).. ( -40.00) >DroYak_CAF1 8804 115 - 1 UUGGUUGUGGUUUAGUUGCGCUUGAUUAUGCUUUUCGCUCUUGCUAUUGCCGCUGCUGAAGCGAAACUGCGUCGCCUGAUUGUCAUGAUUAUCACGAUAAUUAUGCGGC-----CAGCAU ......(((((.((((.(((...((......))..)))....))))..)))))(((((..(((....)))(((((.((((((((.((.....)).)))))))).)))))-----))))). ( -35.90) >consensus CUGGUUGUGGUUUUGUUGCGCUUGAUUAUGCUUUUGGCUCUUGCUAUUGCCGCUGCUGAAGCGCGACUGCGUCGACUGAUUGUCAUGAUUAGCACGAUAAUUAUGCGGC_____CAGCAU ...((((..((...(((((((((.(.((.((...((((....)))).....)))).).))))))))).))((((..((((((((.((.....)).))))))))..)))).....)))).. (-30.82 = -31.46 + 0.64)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:17:08 2006