
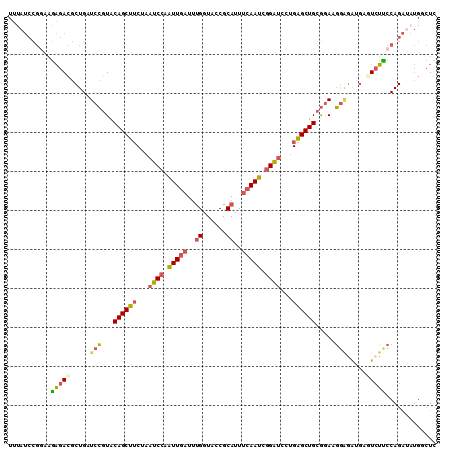
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 18,669,913 – 18,670,023 |
| Length | 110 |
| Max. P | 0.971485 |

| Location | 18,669,913 – 18,670,023 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 78.79 |
| Mean single sequence MFE | -35.45 |
| Consensus MFE | -21.23 |
| Energy contribution | -21.10 |
| Covariance contribution | -0.13 |
| Combinations/Pair | 1.36 |
| Mean z-score | -2.19 |
| Structure conservation index | 0.60 |
| SVM decision value | 1.08 |
| SVM RNA-class probability | 0.912746 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18669913 110 + 22407834 UUUAUCCGAUAGAGACGCUGAUCCGUACAGCUUCUAAUCCAGUUGAUUUGGUACCGCAUUUCAAUCGGAUCCUGGGCUGCGGACGGAGGUGAGUAUCCCAGAUAUGGCUC ...(((((((.((((.((....(((..(((((........)))))...)))....)).)))).))))))).(((((.(((..((....))..))).)))))......... ( -33.00) >DroSec_CAF1 22630 110 + 1 UUUAUCCGGUAGAGACGCUGAUCCGUACAGCUUCUAAUCCAGUUGAUUUGGUACCGCAUUUCAAUCGGAUCCUGAGCUGCGGACGGAGGUUAGUCUUCCAGAUAUGCCUC ..((((.((..(((((.....(((((.((((((...((((.(((((..((......))..))))).))))...))))))...))))).....))))))).))))...... ( -37.30) >DroSim_CAF1 23912 110 + 1 UUUAUCCGGUAGAGACGCUGAUCCGUACAGCUUCUAAUCCAGUUGAUUUGGUACCGCAUUUCAAUCGGAUCCUGAGCUGCGGACGGAGGUGAGUCUUCCAGAUAUGGCUC ..((((.((.(((..((((..(((((.((((((...((((.(((((..((......))..))))).))))...))))))...)))))))))..))).)).))))...... ( -37.80) >DroEre_CAF1 22925 110 + 1 UUUAUCCGGAAGAGACGCUGAUCCGUGCAGCUUCUAAUCCAAUUGAUUUGAUACCGCAUUUCAAUCGGAUCCUGAGCUGCGGAAGGGGAUGAAUCUUCCAGACUUGACUU ....((.((((((....(...(((.((((((((...((((.(((((..((......))..))))).))))...))))))))...)))...)..)))))).))........ ( -35.50) >DroYak_CAF1 22841 110 + 1 UUUAUGCGGAAGAGACGCUGAUCCGUACAGCUUCUAAUCCAAUUGAUUUGGUAUCGCAUUUCAAUCGGAUCCUGGGCUGCGGAAGGAGAUGAAUCUUCCAGAUAUGUCUU .......((((((..((((..((((..((((((...((((.(((((..((......))..))))).))))...))))))))))...)).))..))))))........... ( -32.50) >DroMoj_CAF1 66717 108 + 1 UGCAACAACAUUUGAGCCUGAUCACUACAGCUGCGCAGCCAAUUACUGCGAUAGCGCAUCUCAAUAAGUGAUAGAGCUGCAAAAUA--AAGACUCAAUCAGGCUCGGAUC .........(((((((((((((.......(((((((((.......))))).))))(((.(((.((.....)).))).)))......--........))))))))))))). ( -36.60) >consensus UUUAUCCGGAAGAGACGCUGAUCCGUACAGCUUCUAAUCCAAUUGAUUUGGUACCGCAUUUCAAUCGGAUCCUGAGCUGCGGAAGGAGAUGAGUCUUCCAGAUAUGGCUC ...........(((((.....(((...((((((...((((.(((((..((......))..))))).))))...)))))).....))).....)))))............. (-21.23 = -21.10 + -0.13)


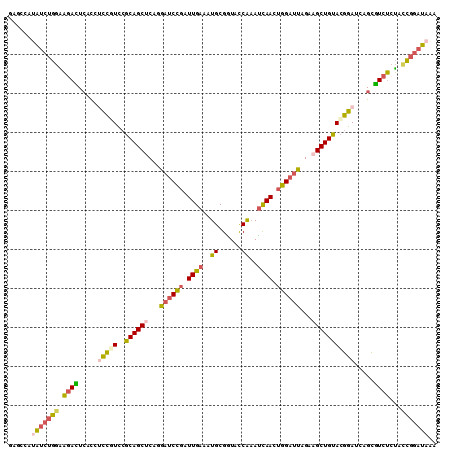
| Location | 18,669,913 – 18,670,023 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 78.79 |
| Mean single sequence MFE | -35.33 |
| Consensus MFE | -25.49 |
| Energy contribution | -25.72 |
| Covariance contribution | 0.23 |
| Combinations/Pair | 1.41 |
| Mean z-score | -2.59 |
| Structure conservation index | 0.72 |
| SVM decision value | 1.68 |
| SVM RNA-class probability | 0.971485 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18669913 110 - 22407834 GAGCCAUAUCUGGGAUACUCACCUCCGUCCGCAGCCCAGGAUCCGAUUGAAAUGCGGUACCAAAUCAACUGGAUUAGAAGCUGUACGGAUCAGCGUCUCUAUCGGAUAAA ......(((((((((.((.(...(((((..(((((....((((((.((((..((......))..)))).))))))....))))))))))...).)).)))..)))))).. ( -30.40) >DroSec_CAF1 22630 110 - 1 GAGGCAUAUCUGGAAGACUAACCUCCGUCCGCAGCUCAGGAUCCGAUUGAAAUGCGGUACCAAAUCAACUGGAUUAGAAGCUGUACGGAUCAGCGUCUCUACCGGAUAAA ......(((((((.((((.....(((((..((((((...((((((.((((..((......))..)))).))))))...))))))))))).....))))...))))))).. ( -36.50) >DroSim_CAF1 23912 110 - 1 GAGCCAUAUCUGGAAGACUCACCUCCGUCCGCAGCUCAGGAUCCGAUUGAAAUGCGGUACCAAAUCAACUGGAUUAGAAGCUGUACGGAUCAGCGUCUCUACCGGAUAAA ......(((((((.((((.(...(((((..((((((...((((((.((((..((......))..)))).))))))...)))))))))))...).))))...))))))).. ( -37.00) >DroEre_CAF1 22925 110 - 1 AAGUCAAGUCUGGAAGAUUCAUCCCCUUCCGCAGCUCAGGAUCCGAUUGAAAUGCGGUAUCAAAUCAAUUGGAUUAGAAGCUGCACGGAUCAGCGUCUCUUCCGGAUAAA .......((((((((((..(.......(((((((((...(((((((((((..((......))..)))))))))))...))))))..))).....)..))))))))))... ( -39.80) >DroYak_CAF1 22841 110 - 1 AAGACAUAUCUGGAAGAUUCAUCUCCUUCCGCAGCCCAGGAUCCGAUUGAAAUGCGAUACCAAAUCAAUUGGAUUAGAAGCUGUACGGAUCAGCGUCUCUUCCGCAUAAA ......(((.(((((((..(..((...((((((((....(((((((((((..((......))..)))))))))))....))))..))))..)).)..))))))).))).. ( -32.80) >DroMoj_CAF1 66717 108 - 1 GAUCCGAGCCUGAUUGAGUCUU--UAUUUUGCAGCUCUAUCACUUAUUGAGAUGCGCUAUCGCAGUAAUUGGCUGCGCAGCUGUAGUGAUCAGGCUCAAAUGUUGUUGCA .....((((((((((.......--......(((.(((...........))).)))(((..((((((.....)))))).)))......))))))))))............. ( -35.50) >consensus GAGCCAUAUCUGGAAGACUCACCUCCGUCCGCAGCUCAGGAUCCGAUUGAAAUGCGGUACCAAAUCAACUGGAUUAGAAGCUGUACGGAUCAGCGUCUCUACCGGAUAAA ......(((((((.((((.....(((((..((((((...((((((.((((..((......))..)))).))))))...))))))))))).....))))...))))))).. (-25.49 = -25.72 + 0.23)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:16:34 2006