
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 18,668,564 – 18,668,724 |
| Length | 160 |
| Max. P | 0.999954 |

| Location | 18,668,564 – 18,668,684 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.67 |
| Mean single sequence MFE | -47.70 |
| Consensus MFE | -42.84 |
| Energy contribution | -42.40 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.11 |
| Mean z-score | -3.17 |
| Structure conservation index | 0.90 |
| SVM decision value | 4.71 |
| SVM RNA-class probability | 0.999942 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18668564 120 + 22407834 CGAUCUCUUUCAGAUCAGUUCGGAACUUUACGCAGUCGAGAUCCUCGGAACUCCGUUCGCGAUGGAUCACGUCCAGGUACAACUGGUUAUGUUCCAGUCGUGCCAGGAAGUGAUCCAGCG .(((((.....)))))..............(((.((((.((....(((....))).)).))))(((((((.(((.(((((.(((((.......))))).))))).))).))))))).))) ( -48.80) >DroSec_CAF1 21245 120 + 1 CGAUCUCAUUCAGAUCAGUGCGGAACUUUACGCAGUCGAGAUCCUCGGCGGUCCGUUCGCGAUGGAUCACGUCCAGGUACAACUGGUUAUGUUCCAGUCGUGCCAGGAAGUGAUCCAACG .(((((.....))))).(.(((((..........((((((...))))))..))))).).((.((((((((.(((.(((((.(((((.......))))).))))).))).)))))))).)) ( -50.70) >DroSim_CAF1 22522 120 + 1 CGAUCUCAUUCAGAUCAGUGCGGAACUUUACGCAGUCGAGAUCCUCGGAGGUCCGUUCGCGAUGGAUCACGUCCAGGUACAACUGGUUAUGUUCCAGUCGUGCCAGGAAGUGAUCCAACG .(((((.....)))))..((((........))))((((.((....(((....))).)).))))(((((((.(((.(((((.(((((.......))))).))))).))).))))))).... ( -50.40) >DroEre_CAF1 21510 120 + 1 CGAUUUCACUCAGAUCAGUUCGGAACUUUACGCAAUCGAGAUCCUCGGAGCUCCGUUCCCGGUGGAUCACGUCCAGGUACAAUUGGCUAUGCUCCAGUCGUGCCAGGAAGUGAUCCAACG .(((((.....))))).....(((((.....((..(((((...))))).))...)))))((.((((((((.(((.(((((.(((((.......))))).))))).))).)))))))).)) ( -42.00) >DroYak_CAF1 21420 120 + 1 CGAUCUCACCCAGAUCAGUUCGAAACUUGACGCAGUUGAGAUCCUCGACGCUCCGUUCCCGGUGGAUCACGUCCAGGUACAACUGGUUAUGUUCCAGUCGUGCCAGGAAGUGAUCCAACG .(((((.....))))).....(((....((.((.((((((...))))))))))..))).((.((((((((.(((.(((((.(((((.......))))).))))).))).)))))))).)) ( -46.60) >consensus CGAUCUCAUUCAGAUCAGUUCGGAACUUUACGCAGUCGAGAUCCUCGGAGCUCCGUUCGCGAUGGAUCACGUCCAGGUACAACUGGUUAUGUUCCAGUCGUGCCAGGAAGUGAUCCAACG .(((((.....))))).(..((((...........(((((...)))))...))))..).((.((((((((.(((.(((((.(((((.......))))).))))).))).)))))))).)) (-42.84 = -42.40 + -0.44)

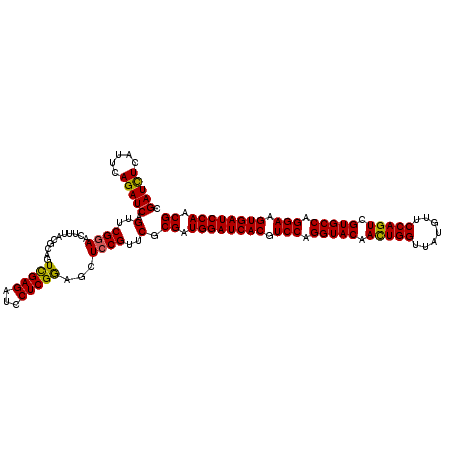
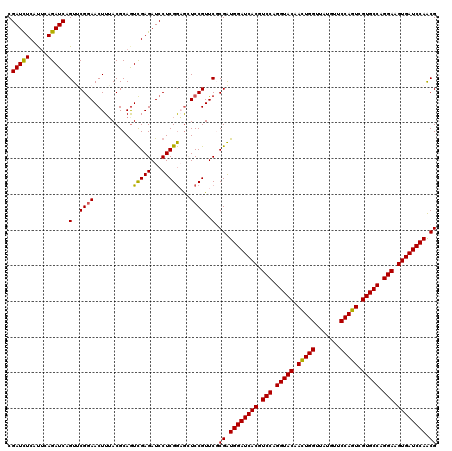
| Location | 18,668,564 – 18,668,684 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.67 |
| Mean single sequence MFE | -48.44 |
| Consensus MFE | -43.62 |
| Energy contribution | -43.54 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.08 |
| Mean z-score | -3.28 |
| Structure conservation index | 0.90 |
| SVM decision value | 4.83 |
| SVM RNA-class probability | 0.999954 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18668564 120 - 22407834 CGCUGGAUCACUUCCUGGCACGACUGGAACAUAACCAGUUGUACCUGGACGUGAUCCAUCGCGAACGGAGUUCCGAGGAUCUCGACUGCGUAAAGUUCCGAACUGAUCUGAAAGAGAUCG (((((((((((.(((.((.((((((((.......)))))))).)).))).))))))))..)))..((((....((((...))))(((......)))))))....(((((.....))))). ( -48.70) >DroSec_CAF1 21245 120 - 1 CGUUGGAUCACUUCCUGGCACGACUGGAACAUAACCAGUUGUACCUGGACGUGAUCCAUCGCGAACGGACCGCCGAGGAUCUCGACUGCGUAAAGUUCCGCACUGAUCUGAAUGAGAUCG .(.((((((((.(((.((.((((((((.......)))))))).)).))).)))))))).)((((((....(((((((...))))...)))....))).)))...(((((.....))))). ( -49.70) >DroSim_CAF1 22522 120 - 1 CGUUGGAUCACUUCCUGGCACGACUGGAACAUAACCAGUUGUACCUGGACGUGAUCCAUCGCGAACGGACCUCCGAGGAUCUCGACUGCGUAAAGUUCCGCACUGAUCUGAAUGAGAUCG (((((((((((.(((.((.((((((((.......)))))))).)).))).))))))))..)))..(((....)))..(((((((..((((........))))(......)..))))))). ( -48.90) >DroEre_CAF1 21510 120 - 1 CGUUGGAUCACUUCCUGGCACGACUGGAGCAUAGCCAAUUGUACCUGGACGUGAUCCACCGGGAACGGAGCUCCGAGGAUCUCGAUUGCGUAAAGUUCCGAACUGAUCUGAGUGAAAUCG ...((((((((.(((.((.((((.(((.......))).)))).)).))).))))))))(((....))).((((...((((((((...((.....))..)))...)))))))))....... ( -41.50) >DroYak_CAF1 21420 120 - 1 CGUUGGAUCACUUCCUGGCACGACUGGAACAUAACCAGUUGUACCUGGACGUGAUCCACCGGGAACGGAGCGUCGAGGAUCUCAACUGCGUCAAGUUUCGAACUGAUCUGGGUGAGAUCG (((((((((((.(((.((.((((((((.......)))))))).)).))).))))))).(((....))))))).....(((((((.(((.((((.((.....)))))).))).))))))). ( -53.40) >consensus CGUUGGAUCACUUCCUGGCACGACUGGAACAUAACCAGUUGUACCUGGACGUGAUCCAUCGCGAACGGAGCUCCGAGGAUCUCGACUGCGUAAAGUUCCGAACUGAUCUGAAUGAGAUCG ((.((((((((.(((.((.((((((((.......)))))))).)).))).)))))))).))....(((....)))..((((((((((......)))).....(......)...)))))). (-43.62 = -43.54 + -0.08)

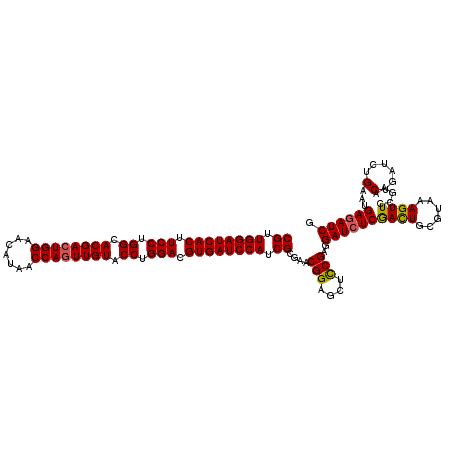

| Location | 18,668,604 – 18,668,724 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.17 |
| Mean single sequence MFE | -47.34 |
| Consensus MFE | -40.56 |
| Energy contribution | -40.08 |
| Covariance contribution | -0.48 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.22 |
| Structure conservation index | 0.86 |
| SVM decision value | 3.92 |
| SVM RNA-class probability | 0.999705 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18668604 120 + 22407834 AUCCUCGGAACUCCGUUCGCGAUGGAUCACGUCCAGGUACAACUGGUUAUGUUCCAGUCGUGCCAGGAAGUGAUCCAGCGACCACUUGAACAGAUUGUAGGGAAUCUCGUCGAUCUUUCC .....(((....))).((((..((((((((.(((.(((((.(((((.......))))).))))).))).))))))))))))......(((.((((((..(((...)))..))))))))). ( -46.80) >DroSec_CAF1 21285 120 + 1 AUCCUCGGCGGUCCGUUCGCGAUGGAUCACGUCCAGGUACAACUGGUUAUGUUCCAGUCGUGCCAGGAAGUGAUCCAACGGCCACUUGAAUAGAUUGUAGGGAAUCUCGUCGAUCUUACC ....(((((((.((((......((((((((.(((.(((((.(((((.......))))).))))).))).))))))))))))))........(((((......))))).)))))....... ( -47.70) >DroSim_CAF1 22562 120 + 1 AUCCUCGGAGGUCCGUUCGCGAUGGAUCACGUCCAGGUACAACUGGUUAUGUUCCAGUCGUGCCAGGAAGUGAUCCAACGACCACUUGAACAGAUUGUAGGGAAUGUCGUCGAUCUUUCC ......(((((((((....)).((((((((.(((.(((((.(((((.......))))).))))).))).))))))))..))))........((((((.(.((....)).))))))).))) ( -45.50) >DroEre_CAF1 21550 120 + 1 AUCCUCGGAGCUCCGUUCCCGGUGGAUCACGUCCAGGUACAAUUGGCUAUGCUCCAGUCGUGCCAGGAAGUGAUCCAACGACCACUUGAACAGAUUGUAGGGGAUGUCGUCGAUCUGUCC ......(((((...)))))((.((((((((.(((.(((((.(((((.......))))).))))).))).)))))))).)).........((((((((.(.((....)).))))))))).. ( -44.20) >DroYak_CAF1 21460 120 + 1 AUCCUCGACGCUCCGUUCCCGGUGGAUCACGUCCAGGUACAACUGGUUAUGUUCCAGUCGUGCCAGGAAGUGAUCCAACGACCACUUGAACAGGUUAUAGGGAAUCUCGUCGAUCUUUCC ....((((((....(((((((.((((((((.(((.(((((.(((((.......))))).))))).))).)))))))).)((((.........))))...))))))..))))))....... ( -52.50) >consensus AUCCUCGGAGCUCCGUUCGCGAUGGAUCACGUCCAGGUACAACUGGUUAUGUUCCAGUCGUGCCAGGAAGUGAUCCAACGACCACUUGAACAGAUUGUAGGGAAUCUCGUCGAUCUUUCC (((..(((...(((.....((.((((((((.(((.(((((.(((((.......))))).))))).))).)))))))).))....((.....)).......)))...)))..)))...... (-40.56 = -40.08 + -0.48)

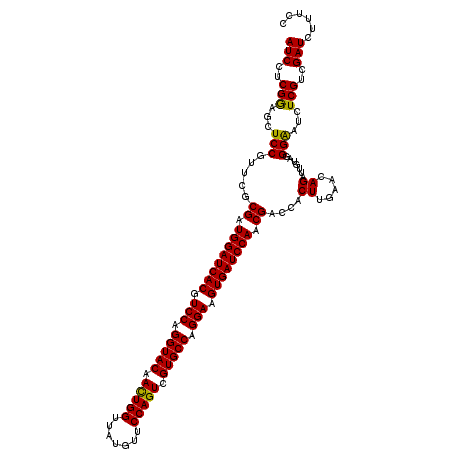
| Location | 18,668,604 – 18,668,724 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.17 |
| Mean single sequence MFE | -46.07 |
| Consensus MFE | -41.76 |
| Energy contribution | -42.52 |
| Covariance contribution | 0.76 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.98 |
| Structure conservation index | 0.91 |
| SVM decision value | 4.68 |
| SVM RNA-class probability | 0.999937 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18668604 120 - 22407834 GGAAAGAUCGACGAGAUUCCCUACAAUCUGUUCAAGUGGUCGCUGGAUCACUUCCUGGCACGACUGGAACAUAACCAGUUGUACCUGGACGUGAUCCAUCGCGAACGGAGUUCCGAGGAU ......(((..((..(((((((((...........))))((((((((((((.(((.((.((((((((.......)))))))).)).))).))))))))..))))..)))))..))..))) ( -48.20) >DroSec_CAF1 21285 120 - 1 GGUAAGAUCGACGAGAUUCCCUACAAUCUAUUCAAGUGGCCGUUGGAUCACUUCCUGGCACGACUGGAACAUAACCAGUUGUACCUGGACGUGAUCCAUCGCGAACGGACCGCCGAGGAU .......(((...(((((......)))))......((((((((((((((((.(((.((.((((((((.......)))))))).)).))).))))))))......)))).))))))).... ( -46.10) >DroSim_CAF1 22562 120 - 1 GGAAAGAUCGACGACAUUCCCUACAAUCUGUUCAAGUGGUCGUUGGAUCACUUCCUGGCACGACUGGAACAUAACCAGUUGUACCUGGACGUGAUCCAUCGCGAACGGACCUCCGAGGAU ((((.(........).))))......(((((((..((((....((((((((.(((.((.((((((((.......)))))))).)).))).)))))))))))))))))))((.....)).. ( -44.30) >DroEre_CAF1 21550 120 - 1 GGACAGAUCGACGACAUCCCCUACAAUCUGUUCAAGUGGUCGUUGGAUCACUUCCUGGCACGACUGGAGCAUAGCCAAUUGUACCUGGACGUGAUCCACCGGGAACGGAGCUCCGAGGAU ((((.....((((((.......((.....)).......))))))(((((((.(((.((.((((.(((.......))).)))).)).))).))))))).(((....))).).)))...... ( -41.54) >DroYak_CAF1 21460 120 - 1 GGAAAGAUCGACGAGAUUCCCUAUAACCUGUUCAAGUGGUCGUUGGAUCACUUCCUGGCACGACUGGAACAUAACCAGUUGUACCUGGACGUGAUCCACCGGGAACGGAGCGUCGAGGAU .......((((((.(.(((((....(((.........))).(.((((((((.(((.((.((((((((.......)))))))).)).))).)))))))).)))))))....)))))).... ( -50.20) >consensus GGAAAGAUCGACGAGAUUCCCUACAAUCUGUUCAAGUGGUCGUUGGAUCACUUCCUGGCACGACUGGAACAUAACCAGUUGUACCUGGACGUGAUCCAUCGCGAACGGAGCUCCGAGGAU ...................(((....(((((((..((((....((((((((.(((.((.((((((((.......)))))))).)).))).)))))))))))))))))))......))).. (-41.76 = -42.52 + 0.76)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:16:28 2006