
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 18,539,473 – 18,539,590 |
| Length | 117 |
| Max. P | 0.997348 |

| Location | 18,539,473 – 18,539,590 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 71.93 |
| Mean single sequence MFE | -54.53 |
| Consensus MFE | -29.07 |
| Energy contribution | -29.82 |
| Covariance contribution | 0.75 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.64 |
| Structure conservation index | 0.53 |
| SVM decision value | 2.70 |
| SVM RNA-class probability | 0.996484 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18539473 117 + 22407834 GCUGGGCGAGGGGCAGGGGGACACCGGCGUGGCGUGUGGCGAUGAGCAGCGAGUGCUCGCUGGAUCCGUAGCCGCCGUACCCCGUGCCCCCCUGCUCCGUGUUCAUCCUGCCAGUGG ((((((((.(((((((((((.(((.((.(((.((.(((((...((.((((((....))))))..))....)))))))))))).))).))))))))))).)))........))))).. ( -63.40) >DroPse_CAF1 29260 102 + 1 GUGCGGCUGGUGGCAGGGGGACACCGGU---GCGUGUGGCAAGGAGGAGCGAGUGCUCGCCGUAGCCAUAGC---CGUACCCUGUGCCCCCCUGCUCCGCAUUCAUUC--------- ((((((.....(((((((((.(((.(((---(((((((((..((..((((....)))).))...))))))..---))))))..))).)))))))))))))))......--------- ( -56.30) >DroGri_CAF1 18524 102 + 1 GCUGUGAUAGCGGCAGGGGGACACCGGUGCG---UGUGGCAAUGAGCAGACUAUGCUCACU-----CAUA----CCGUACCCUGUGCCCCCCUGCUCCGCAGGCAUUCGAG---CUG ((((((...(((((((((((.(((.((((((---(((((...((((((.....)))))).)-----))))----.))))))..))).))))))))..)))...)))...))---).. ( -49.90) >DroWil_CAF1 20638 105 + 1 GCUGUGUUAGCGGCAGGGGGACACCCGA---GCGUGUGGCAAGUAGCAGACUGUGCUCACUCA---------CACCGUACCCUGUGCCCCCCUGCUCCGCGGGCAUCCUGGCAGCUA (((((....(.(((((((((.(((..(.---(((.(((...(((((((.....)))).)))..---------)))))).)...))).))))))))).).((((...))))))))).. ( -44.90) >DroMoj_CAF1 20455 105 + 1 GCUGUGAUAGCGGCAGGGGGACACCGGUGCGGUGUGUGGCAAUGAGCAGGCUAUGCUCACU-----CAUA----CCGUACCCUGUGCCCCCCUGCUCCGCGGGCAUACGUG---UCG ((((((...(((((((((((.(((.(((((((((((.(....((((((.....))))))).-----))))----)))))))..))).))))))))..)))...)))).)).---... ( -56.40) >DroPer_CAF1 27335 102 + 1 GUGCGGCUGGUGGCAGGGGGACACCGGU---GCGUGUGGCAAGGAGGAGCGAGUGCUCGCCGUAGCCAUAGC---CGUACCCUGUGCCCCCCUGCUCCGCAUUCAUUC--------- ((((((.....(((((((((.(((.(((---(((((((((..((..((((....)))).))...))))))..---))))))..))).)))))))))))))))......--------- ( -56.30) >consensus GCUGGGCUAGCGGCAGGGGGACACCGGU___GCGUGUGGCAAGGAGCAGCCAGUGCUCACUG____CAUA____CCGUACCCUGUGCCCCCCUGCUCCGCAGGCAUUC__G_____G .........(((((((((((.(((.((....((.....))...(((((.....)))))......................)).))).))))))))..)))................. (-29.07 = -29.82 + 0.75)

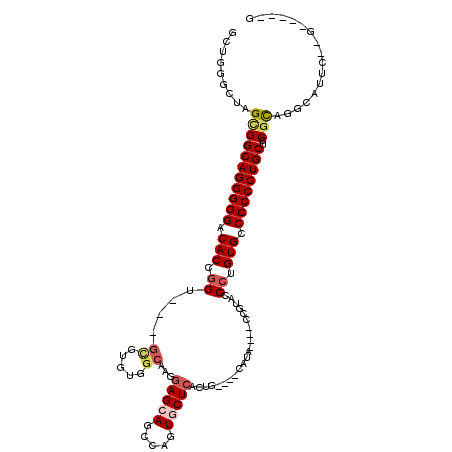
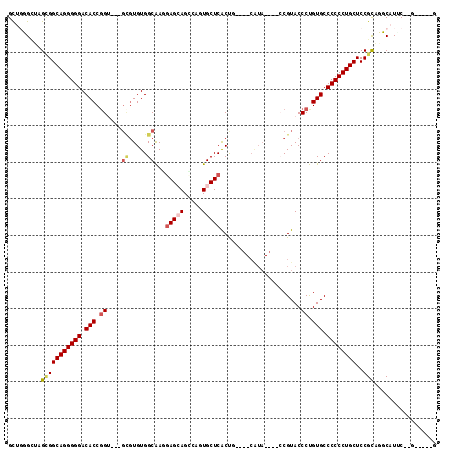
| Location | 18,539,473 – 18,539,590 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 71.93 |
| Mean single sequence MFE | -51.75 |
| Consensus MFE | -30.47 |
| Energy contribution | -31.42 |
| Covariance contribution | 0.95 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.78 |
| Structure conservation index | 0.59 |
| SVM decision value | 2.84 |
| SVM RNA-class probability | 0.997348 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18539473 117 - 22407834 CCACUGGCAGGAUGAACACGGAGCAGGGGGGCACGGGGUACGGCGGCUACGGAUCCAGCGAGCACUCGCUGCUCAUCGCCACACGCCACGCCGGUGUCCCCCUGCCCCUCGCCCAGC ...(((((.(......)..((.((((((((((((.((((.(((.(((....((..((((((....)))))).))...))).).))..)).)).)))))))))))).))..).)))). ( -56.90) >DroPse_CAF1 29260 102 - 1 ---------GAAUGAAUGCGGAGCAGGGGGGCACAGGGUACG---GCUAUGGCUACGGCGAGCACUCGCUCCUCCUUGCCACACGC---ACCGGUGUCCCCCUGCCACCAGCCGCAC ---------.......(((((.((((((((((((..(((.((---....((((...((.((((....))))..))..))))..)).---))).))))))))))))......))))). ( -52.50) >DroGri_CAF1 18524 102 - 1 CAG---CUCGAAUGCCUGCGGAGCAGGGGGGCACAGGGUACGG----UAUG-----AGUGAGCAUAGUCUGCUCAUUGCCACA---CGCACCGGUGUCCCCCUGCCGCUAUCACAGC ..(---(......))..(((..((((((((((((..(((.(((----(...-----((((((((.....))))))))...)).---)).))).)))))))))))))))......... ( -47.40) >DroWil_CAF1 20638 105 - 1 UAGCUGCCAGGAUGCCCGCGGAGCAGGGGGGCACAGGGUACGGUG---------UGAGUGAGCACAGUCUGCUACUUGCCACACGC---UCGGGUGUCCCCCUGCCGCUAACACAGC ..((((...((....))(((..((((((((((((.((((...(((---------..(((.((((.....)))))))..).))..))---))..))))))))))))))).....)))) ( -51.00) >DroMoj_CAF1 20455 105 - 1 CGA---CACGUAUGCCCGCGGAGCAGGGGGGCACAGGGUACGG----UAUG-----AGUGAGCAUAGCCUGCUCAUUGCCACACACCGCACCGGUGUCCCCCUGCCGCUAUCACAGC ...---...........(((..((((((((((((..(((.(((----(.((-----((((((((.....))))))))....)).)))).))).)))))))))))))))......... ( -50.20) >DroPer_CAF1 27335 102 - 1 ---------GAAUGAAUGCGGAGCAGGGGGGCACAGGGUACG---GCUAUGGCUACGGCGAGCACUCGCUCCUCCUUGCCACACGC---ACCGGUGUCCCCCUGCCACCAGCCGCAC ---------.......(((((.((((((((((((..(((.((---....((((...((.((((....))))..))..))))..)).---))).))))))))))))......))))). ( -52.50) >consensus C_____C__GAAUGAACGCGGAGCAGGGGGGCACAGGGUACGG____UAUG____CAGCGAGCACACGCUGCUCAUUGCCACACGC___ACCGGUGUCCCCCUGCCGCUAGCACAGC ...................((.((((((((((((.((......................(((((.....)))))...((.....))....)).))))))))))))..))........ (-30.47 = -31.42 + 0.95)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:15:22 2006