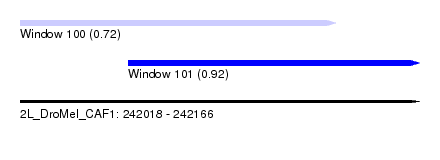
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 242,018 – 242,166 |
| Length | 148 |
| Max. P | 0.920717 |

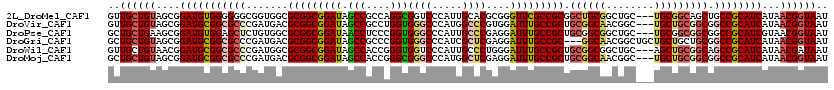
| Location | 242,018 – 242,135 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.58 |
| Mean single sequence MFE | -61.72 |
| Consensus MFE | -47.56 |
| Energy contribution | -48.40 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.22 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.39 |
| SVM RNA-class probability | 0.718528 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 242018 117 + 22407834 GUUGCUGUAGCGGAUGUGGGGGGCGGUGGCGCGGCGGAUAGCCGCCAGGCGGUCCCAUUGCACGCGGGUUCGCCGCGGCUGCGGCUGC---UGCGGCAGCUGCCGCAUCAUAACGGUAAU (((((((((((((.((..((((((.((.(.(((((.....))))))..)).)))))......(((((.....))))).)..)).))))---)))))))))(((((........))))).. ( -64.80) >DroVir_CAF1 39785 117 + 1 GUUGCUGUAGCGGAUGCGGCGCCCGAUGACGCGGCGGAUAGCCGCCUGGUGGGCCCAUGGCCCGUGGAUUUGCCGCUGCGGCAACGGC---UGCUGCGGCGGCCGCAUCAUAACGGUAAU ((((((((....((((((((((((.((.(.(((((.....))))).).))))))(((((...)))))...((((((.(((((....))---))).))))))))))))))...)))))))) ( -66.70) >DroPse_CAF1 40964 117 + 1 GCUGCUGAAGCGGAUGUGGAGCUCUGUGGCGCGGCGGAUAACCUCCCGGUGGGCCCAUUGCCCGAGGAUUUGCCGCUGCGGCGGCUGC---UGCGGCGGCGGCCGCAUCGUAACGGUAAU ..(((((..(((.((((((.((..((.(((.((.(((........))).)).)))))..))))..((...((((((((((((....))---)))))))))).)))))))))..))))).. ( -57.50) >DroGri_CAF1 40768 117 + 1 GCUGCUGUAGCGGAUGCGGCGCCCGAUGACGCGGCGGAUAGCCGCCCGGUGGGCCCAUCGCUCGAGGAUUUGCCGC---GGCAACGGCUGCUGCUGCUGCGGCCGCAUCAUAACGGUAAU ((.(((((((((...((((((.(((.((.((((((((((...((..(((((....)))))..))...)))))))))---).)).))).))))))))))))))).)).............. ( -58.90) >DroWil_CAF1 42686 117 + 1 GUUGCUGUAACGGAUGCGGCGCCCGAUGGCGCGGCGGAUAGCCACCGGGUGGUCCCAUUGCCCUGGGAUUUGCCGCUGCGGCGGCUGC---AGCUGCGGCAGCCGCAUCAUAACGAUAAU ((((.(((....(((((((((((....)))(((((((((..(((..(((..((...))..)))))).)))))))))(((.(((((...---.))))).)))))))))))...))).)))) ( -59.50) >DroMoj_CAF1 38325 117 + 1 GCUGCUGUAGCGGAUGCGGCGCCCGAUGACGCGGCGGAUAGCCACCGGGCGGGCCCAUGGCUCGAGGAUUUGCCGCUGCGGCAACGGC---UGCUGCGGCGGCCGCAUCAUAACGGUAAU ..((((((....(((((((((((((.....(.(((.....))).))))))(((((...))))).......((((((.(((((....))---))).))))))))))))))...)))))).. ( -62.90) >consensus GCUGCUGUAGCGGAUGCGGCGCCCGAUGACGCGGCGGAUAGCCGCCCGGUGGGCCCAUUGCCCGAGGAUUUGCCGCUGCGGCAACGGC___UGCUGCGGCGGCCGCAUCAUAACGGUAAU ..((((((....(((((((((((.......(((((((((.(((....)))((((.....))))....))))))))).((((((........))))))))).))))))))...)))))).. (-47.56 = -48.40 + 0.84)
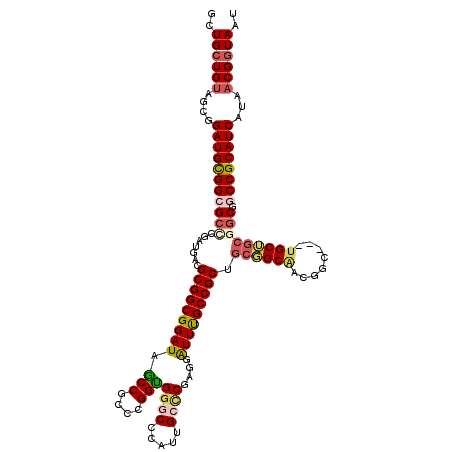
| Location | 242,058 – 242,166 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 79.89 |
| Mean single sequence MFE | -44.94 |
| Consensus MFE | -32.12 |
| Energy contribution | -32.77 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.71 |
| SVM decision value | 1.14 |
| SVM RNA-class probability | 0.920717 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 242058 108 + 22407834 GCCGCCAGGCGGUCCCAUUGCACGCGGGUUCGCCGCGGCUGCGGCUGCUGCGGCAGCUGCCGCAUCAUAACGGUAAUAGGGAUCUCUGUGGAU-CAGAAAAA---GAAAACA (((....)))(((((((((((.(((((.....)))))((.((((((((....)))))))).)).........))))).))))))((((.....-))))....---....... ( -50.10) >DroVir_CAF1 39825 107 + 1 GCCGCCUGGUGGGCCCAUGGCCCGUGGAUUUGCCGCUGCGGCAACGGCUGCUGCGGCGGCCGCAUCAUAACGGUAAUAGGGAUCUCUGUGUGU-UA-AAAAG---GAAGAGA ((((..(((((((((...)))))((((...((((((.(((((....))))).)))))).))))))))...))))........(((((......-..-.....---..))))) ( -45.36) >DroPse_CAF1 41004 111 + 1 ACCUCCCGGUGGGCCCAUUGCCCGAGGAUUUGCCGCUGCGGCGGCUGCUGCGGCGGCGGCCGCAUCGUAACGGUAAUAGGGAUCUCUAUAAAG-AAGGAAAGACCGAAAAAA .((((((..(((((.....))))).....((((((.((((((((((((((...))))))))))..)))).))))))..))))(((......))-).)).............. ( -44.80) >DroWil_CAF1 42726 109 + 1 GCCACCGGGUGGUCCCAUUGCCCUGGGAUUUGCCGCUGCGGCGGCUGCAGCUGCGGCAGCCGCAUCAUAACGAUAAUAGGGAUCUCUAAAAUGGUAGAAGAA---AAAAAAA ((((...((.((((((((((...((..((.(((.(((((.(((((....))))).))))).)))..))..)).)))).)))))).))....)))).......---....... ( -40.70) >DroMoj_CAF1 38365 107 + 1 GCCACCGGGCGGGCCCAUGGCUCGAGGAUUUGCCGCUGCGGCAACGGCUGCUGCGGCGGCCGCAUCAUAACGGUAAUAGGGAUCUCUGUAAGC-GG-AUAAU---GAAAUUA ((..((...((((((...)))))).))....((((((((((((.....)))))))))))).)).((((..((.(.(((((....))))).).)-).-...))---))..... ( -43.90) >DroPer_CAF1 41418 111 + 1 ACCUCCCGGUGGGCCCAUUGCCCGAGGAUUUGCCGCUGCGGCGGCUGCUGCGGCGGCGGCCGCAUCGUAACGGUAAUAGGGAUCUCUAUAAAG-AAGGAAAGACCGAAAAAA .((((((..(((((.....))))).....((((((.((((((((((((((...))))))))))..)))).))))))..))))(((......))-).)).............. ( -44.80) >consensus GCCACCCGGUGGGCCCAUUGCCCGAGGAUUUGCCGCUGCGGCGGCUGCUGCGGCGGCGGCCGCAUCAUAACGGUAAUAGGGAUCUCUAUAAAG_AAGAAAAG___GAAAAAA (((....)))((.(((((((((....(((..((((((((((((.....))))))))))))...))).....)))))).))).))............................ (-32.12 = -32.77 + 0.64)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:24:52 2006