
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 18,508,767 – 18,508,927 |
| Length | 160 |
| Max. P | 0.993043 |

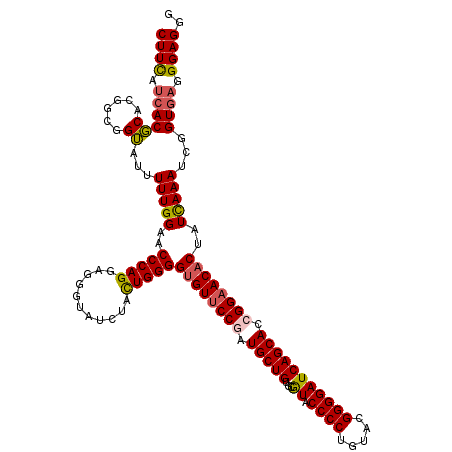
| Location | 18,508,767 – 18,508,887 |
|---|---|
| Length | 120 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.00 |
| Mean single sequence MFE | -45.52 |
| Consensus MFE | -38.47 |
| Energy contribution | -38.85 |
| Covariance contribution | 0.38 |
| Combinations/Pair | 1.13 |
| Mean z-score | -1.45 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.14 |
| SVM RNA-class probability | 0.604116 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18508767 120 + 22407834 CUUCAUCACGCACGGCGGUAUUUUUGGAACCCAGGAGGGUAUUUACUGGGGUGUUCCGAUGCUGUGCGUACCCCUGUACGGGGAUCAGCACCGGAACACUAUUAAAUCGGUGAGGGAGGG (((((((.(((...))).........((.(((((...........)))))((((((((.(((((......((((.....))))..))))).)))))))).......)))))))))..... ( -46.60) >DroPse_CAF1 34566 120 + 1 CUUUAUCACCCACGGAGGGAUCUUUGGCACCCAGGAGGGCAUCUAUUGGGGCGUGCCGAUGCUGUGCAUUCCCCUCUAUGGGGAUCAGCACAGGAACACGAUCAAAUCAGUGCGGGAGGG .....((.(((((.((..((((.(((((((((....))((..(....)..)))))))))(.((((((..(((((.....)))))...)))))).)....))))...)).))).))))... ( -46.90) >DroSec_CAF1 29167 120 + 1 CUUCAUCACGCACGGCGGUAUUUUUGGAACCCAGGAGGGUAUUUACUGGGGUGUUCCGAUGCUGUGCGUACCCCUAUACGGGGAUCAGCACCGGAACACUAUCAAAUCGGUGAGGGAGGG (((((((.(((...))).....(((((..(((((...........)))))((((((((.(((((......((((.....))))..))))).))))))))..)))))..)))))))..... ( -48.20) >DroYak_CAF1 29219 120 + 1 CUUCAUCACGCACGGCGGUAUUUUUGGAACCCAGGAGGGUAUCUACUGGGGUGUUCCCAUGCUGUGUGUACCCCUGUACGGGGAUCAGCACCGGAACACUAUCAAAUCGGUUAGGGAGGG ((((.((.(((...))).....(((((..(((((.((.....)).)))))(((((((..(((((......((((.....))))..)))))..)))))))..))))).......)))))). ( -40.40) >consensus CUUCAUCACGCACGGCGGUAUUUUUGGAACCCAGGAGGGUAUCUACUGGGGUGUUCCGAUGCUGUGCGUACCCCUGUACGGGGAUCAGCACCGGAACACUAUCAAAUCGGUGAGGGAGGG ((((.((((((......))...(((((..(((((...........)))))((((((((.(((((...((.((((.....))))))))))).))))))))..)))))...)))).)))).. (-38.47 = -38.85 + 0.38)


| Location | 18,508,767 – 18,508,887 |
|---|---|
| Length | 120 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.00 |
| Mean single sequence MFE | -42.08 |
| Consensus MFE | -36.89 |
| Energy contribution | -37.08 |
| Covariance contribution | 0.19 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.30 |
| Structure conservation index | 0.88 |
| SVM decision value | 1.72 |
| SVM RNA-class probability | 0.973811 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18508767 120 - 22407834 CCCUCCCUCACCGAUUUAAUAGUGUUCCGGUGCUGAUCCCCGUACAGGGGUACGCACAGCAUCGGAACACCCCAGUAAAUACCCUCCUGGGUUCCAAAAAUACCGCCGUGCGUGAUGAAG .....(.((((((........((((((((((((((..((((.....))))......))))))))))))))(((((...........))))).................)).)))).)... ( -41.90) >DroPse_CAF1 34566 120 - 1 CCCUCCCGCACUGAUUUGAUCGUGUUCCUGUGCUGAUCCCCAUAGAGGGGAAUGCACAGCAUCGGCACGCCCCAAUAGAUGCCCUCCUGGGUGCCAAAGAUCCCUCCGUGGGUGAUAAAG ..(.(((((...((...(((((((((.((((((...(((((.....)))))..))))))....)))))((((((..((.....))..)))).))....))))..)).))))).)...... ( -43.60) >DroSec_CAF1 29167 120 - 1 CCCUCCCUCACCGAUUUGAUAGUGUUCCGGUGCUGAUCCCCGUAUAGGGGUACGCACAGCAUCGGAACACCCCAGUAAAUACCCUCCUGGGUUCCAAAAAUACCGCCGUGCGUGAUGAAG .....(.((((...((((...((((((((((((((..((((.....))))......))))))))))))))(((((...........)))))...))))......((...)))))).)... ( -42.60) >DroYak_CAF1 29219 120 - 1 CCCUCCCUAACCGAUUUGAUAGUGUUCCGGUGCUGAUCCCCGUACAGGGGUACACACAGCAUGGGAACACCCCAGUAGAUACCCUCCUGGGUUCCAAAAAUACCGCCGUGCGUGAUGAAG ..............((((...(((((((.((((((..((((.....))))......)))))).)))))))(((((.((.....)).)))))...))))....((((...))).)...... ( -40.20) >consensus CCCUCCCUCACCGAUUUGAUAGUGUUCCGGUGCUGAUCCCCGUACAGGGGUACGCACAGCAUCGGAACACCCCAGUAAAUACCCUCCUGGGUUCCAAAAAUACCGCCGUGCGUGAUGAAG ...((.(.(((..(((((...((((((((((((((..((((.....))))......)))))))))))))).....)))))((((....))))...............))).).))..... (-36.89 = -37.08 + 0.19)


| Location | 18,508,807 – 18,508,927 |
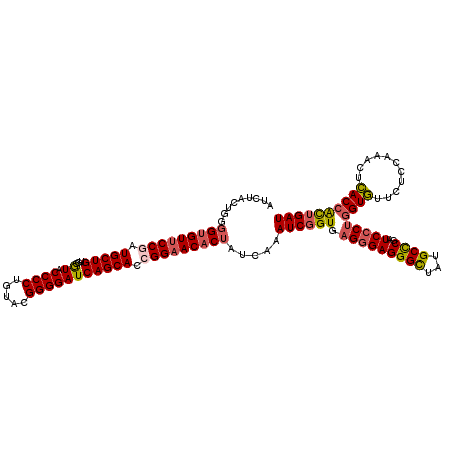
|---|---|
| Length | 120 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 89.17 |
| Mean single sequence MFE | -47.33 |
| Consensus MFE | -38.86 |
| Energy contribution | -39.80 |
| Covariance contribution | 0.94 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.75 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.84 |
| SVM RNA-class probability | 0.979586 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18508807 120 + 22407834 AUUUACUGGGGUGUUCCGAUGCUGUGCGUACCCCUGUACGGGGAUCAGCACCGGAACACUAUUAAAUCGGUGAGGGAGGGCUAUGCCCGAUCGCUGGUGUUCUCCAAACUCACCACUGAU .........(((((((((.(((((......((((.....))))..))))).)))))))))........(((((((((((((...((......))....)))))))...))))))...... ( -49.70) >DroPse_CAF1 34606 120 + 1 AUCUAUUGGGGCGUGCCGAUGCUGUGCAUUCCCCUCUAUGGGGAUCAGCACAGGAACACGAUCAAAUCAGUGCGGGAGGGUUAUGCUCGAUCCCUGGUCUUCUCCAAACUGACAGUCGAU (((.(((((..((((....(.((((((..(((((.....)))))...)))))).).))))..)...(((((..(((((((.....)))..))))(((......))).))))))))).))) ( -42.10) >DroSec_CAF1 29207 120 + 1 AUUUACUGGGGUGUUCCGAUGCUGUGCGUACCCCUAUACGGGGAUCAGCACCGGAACACUAUCAAAUCGGUGAGGGAGGGCUAUGCCCGAUCCCUGGUGUUCUCCAAACUCACCACUGAU ......((((((((((((.(((((......((((.....))))..))))).))))))))).))).((((((.(((((((((...))))..)))))((((...........)))))))))) ( -50.80) >DroYak_CAF1 29259 120 + 1 AUCUACUGGGGUGUUCCCAUGCUGUGUGUACCCCUGUACGGGGAUCAGCACCGGAACACUAUCAAAUCGGUUAGGGAGGGCUAUGCCCGAUCCCUGGUGUUCUCCAAACUCACCACUGAU ......(((((((((((..(((((......((((.....))))..)))))..)))))))).))).((((((.(((((((((...))))..)))))((((...........)))))))))) ( -46.70) >consensus AUCUACUGGGGUGUUCCGAUGCUGUGCGUACCCCUGUACGGGGAUCAGCACCGGAACACUAUCAAAUCGGUGAGGGAGGGCUAUGCCCGAUCCCUGGUGUUCUCCAAACUCACCACUGAU .........(((((((((.(((((...((.((((.....))))))))))).))))))))).....((((((.(((((((((...))))..)))))((((...........)))))))))) (-38.86 = -39.80 + 0.94)


| Location | 18,508,807 – 18,508,927 |
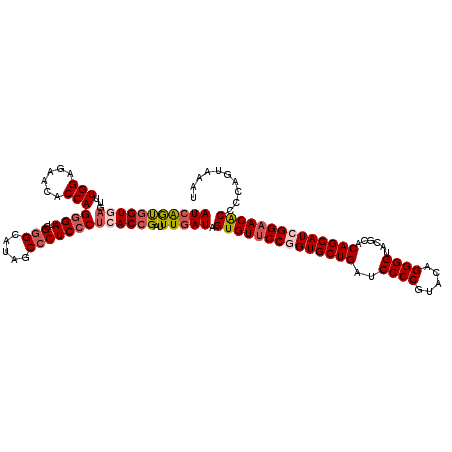
|---|---|
| Length | 120 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 89.17 |
| Mean single sequence MFE | -47.68 |
| Consensus MFE | -40.65 |
| Energy contribution | -42.40 |
| Covariance contribution | 1.75 |
| Combinations/Pair | 1.10 |
| Mean z-score | -3.14 |
| Structure conservation index | 0.85 |
| SVM decision value | 2.37 |
| SVM RNA-class probability | 0.993043 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18508807 120 - 22407834 AUCAGUGGUGAGUUUGGAGAACACCAGCGAUCGGGCAUAGCCCUCCCUCACCGAUUUAAUAGUGUUCCGGUGCUGAUCCCCGUACAGGGGUACGCACAGCAUCGGAACACCCCAGUAAAU .....(((((((.((((......)))).....((((...))))...)))))))(((((...((((((((((((((..((((.....))))......)))))))))))))).....))))) ( -50.20) >DroPse_CAF1 34606 120 - 1 AUCGACUGUCAGUUUGGAGAAGACCAGGGAUCGAGCAUAACCCUCCCGCACUGAUUUGAUCGUGUUCCUGUGCUGAUCCCCAUAGAGGGGAAUGCACAGCAUCGGCACGCCCCAAUAGAU (((((..((((((.(((......)))((((.............))))..)))))))))))((((((.((((((...(((((.....)))))..))))))....))))))........... ( -37.52) >DroSec_CAF1 29207 120 - 1 AUCAGUGGUGAGUUUGGAGAACACCAGGGAUCGGGCAUAGCCCUCCCUCACCGAUUUGAUAGUGUUCCGGUGCUGAUCCCCGUAUAGGGGUACGCACAGCAUCGGAACACCCCAGUAAAU ((((.((((((...(((......)))((((..((((...))))))))))))))...)))).((((((((((((((..((((.....))))......)))))))))))))).......... ( -55.00) >DroYak_CAF1 29259 120 - 1 AUCAGUGGUGAGUUUGGAGAACACCAGGGAUCGGGCAUAGCCCUCCCUAACCGAUUUGAUAGUGUUCCGGUGCUGAUCCCCGUACAGGGGUACACACAGCAUGGGAACACCCCAGUAGAU ((((.((((......((......))(((((..((((...))))))))).))))...)))).(((((((.((((((..((((.....))))......)))))).))))))).......... ( -48.00) >consensus AUCAGUGGUGAGUUUGGAGAACACCAGGGAUCGGGCAUAGCCCUCCCUCACCGAUUUGAUAGUGUUCCGGUGCUGAUCCCCGUACAGGGGUACGCACAGCAUCGGAACACCCCAGUAAAU (((((((((((...(((......)))((((..(((.....)))))))))))))..))))).((((((((((((((..((((.....))))......)))))))))))))).......... (-40.65 = -42.40 + 1.75)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:14:47 2006