
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 18,336,706 – 18,336,831 |
| Length | 125 |
| Max. P | 0.998493 |

| Location | 18,336,706 – 18,336,804 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.05 |
| Mean single sequence MFE | -36.00 |
| Consensus MFE | -17.51 |
| Energy contribution | -18.57 |
| Covariance contribution | 1.06 |
| Combinations/Pair | 1.14 |
| Mean z-score | -4.08 |
| Structure conservation index | 0.49 |
| SVM decision value | 3.12 |
| SVM RNA-class probability | 0.998493 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
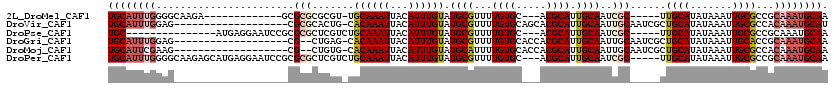
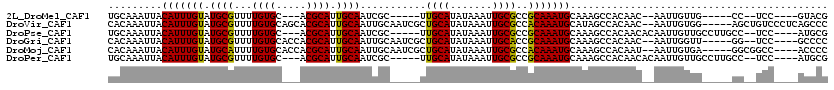
>2L_DroMel_CAF1 18336706 98 + 22407834 UGCAUUUGGGGCAAGA-------------GCGCGCGCGU-UGCAAAUUACAUUUGUAUGCGUUUUGUGC---ACGCAUUGCAAUCGC-----UUGCAUAUAAAUUGCGCCGCAAAUGCAA ((((((((.(((....-------------((((((((((-.((((((...))))))))))))...))))---..(((.(((((....-----))))).......)))))).)))))))). ( -39.20) >DroVir_CAF1 273527 100 + 1 UGCAUUUGGAG-------------------CGCGCACUG-CACAAAUUACAUUUGUAUGCGUUUUGUGCAGCACGCAUUGCAAUUGCAAUCGCUGCAUAUAAAUUGCGCCACAAAUGCAU ((((((((..(-------------------(((....((-((((((...(((....)))...))))))))(((.(((((((....))))).))))).........))))..)))))))). ( -41.30) >DroPse_CAF1 213074 97 + 1 UGC---------------AUGAGGAAUCCGCGCGCUCGUCUGCAAAUUACAUUUGUAUGCGUUUUGUGC---ACGCAUUGCAAUCGC-----UUGCAUAUAAAUUGCGCCGCAAAUGCAA (((---------------((..((.....(((((..(((.(((((((...))))))).)))...)))))---.((((.(((((....-----))))).......))))))....))))). ( -28.10) >DroGri_CAF1 222899 98 + 1 UGCAUUUGGAG-------------------CG--CUGAG-CACAAAUUACAUUUGUAUGCGUUUUGUGCACCACGCAUUGCAAUUGCAAUCGCUGCAUAUAAAUUGCACCGCAAAUGCAA ((((((((..(-------------------((--..(.(-((((((...(((....)))...))))))).)..)))(((((....)))))((.((((.......)))).)))))))))). ( -31.70) >DroMoj_CAF1 293756 98 + 1 UGCAUUCGAAG-------------------CG--CUGUG-CACAAAUUACAUUUGUAUGCAUUUUGUGCACCACGCAUUGCAAUUGCAAUCGCUGCAUAUAAAUUGCGCCACAAAUGCAA ((((((.(..(-------------------((--(.(((-((((((...(((....)))...)))))))))...(((((((....))))).))............))))..).)))))). ( -33.30) >DroPer_CAF1 215411 112 + 1 UGCAUUUGGGGCAAGAGCAUGAGGAAUCCGCGCGCUCGUCUGCAAAUUACAUUUGUAUGCGUUUUGUGC---ACGCAUUGCAAUCGC-----UUGCAUAUAAAUUGCGCCGCAAAUGCAA ((((((((.(((..((((.((.((...)).)).))))....(((......(((((((((((.....(((---(.....)))).....-----.))))))))))))))))).)))))))). ( -42.40) >consensus UGCAUUUGGAG___________________CGCGCUCGG_CACAAAUUACAUUUGUAUGCGUUUUGUGC___ACGCAUUGCAAUCGC_____CUGCAUAUAAAUUGCGCCGCAAAUGCAA ((((((((.......................(((.......((((((...)))))).((((...((((.....)))).))))..)))......((((.......))))...)))))))). (-17.51 = -18.57 + 1.06)
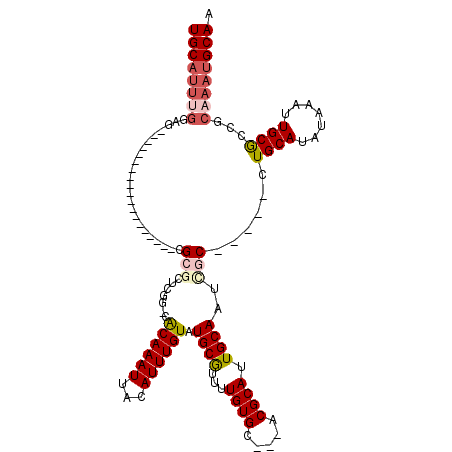
| Location | 18,336,706 – 18,336,804 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.05 |
| Mean single sequence MFE | -39.59 |
| Consensus MFE | -20.33 |
| Energy contribution | -20.58 |
| Covariance contribution | 0.25 |
| Combinations/Pair | 1.18 |
| Mean z-score | -5.09 |
| Structure conservation index | 0.51 |
| SVM decision value | 2.68 |
| SVM RNA-class probability | 0.996334 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18336706 98 - 22407834 UUGCAUUUGCGGCGCAAUUUAUAUGCAA-----GCGAUUGCAAUGCGU---GCACAAAACGCAUACAAAUGUAAUUUGCA-ACGCGCGCGC-------------UCUUGCCCCAAAUGCA .((((((((.(((((((.((((((((((-----....)))).((((((---.......))))))....)))))).)))).-..((....))-------------....))).)))))))) ( -36.50) >DroVir_CAF1 273527 100 - 1 AUGCAUUUGUGGCGCAAUUUAUAUGCAGCGAUUGCAAUUGCAAUGCGUGCUGCACAAAACGCAUACAAAUGUAAUUUGUG-CAGUGCGCG-------------------CUCCAAAUGCA .((((((((..((((............((.(((((....)))))))(..(((((((((..((((....))))..))))))-)))..))))-------------------)..)))))))) ( -48.40) >DroPse_CAF1 213074 97 - 1 UUGCAUUUGCGGCGCAAUUUAUAUGCAA-----GCGAUUGCAAUGCGU---GCACAAAACGCAUACAAAUGUAAUUUGCAGACGAGCGCGCGGAUUCCUCAU---------------GCA .((((((((((((((((.((((((((((-----....)))).((((((---.......))))))....)))))).))))......)).)))))).......)---------------))) ( -26.81) >DroGri_CAF1 222899 98 - 1 UUGCAUUUGCGGUGCAAUUUAUAUGCAGCGAUUGCAAUUGCAAUGCGUGGUGCACAAAACGCAUACAAAUGUAAUUUGUG-CUCAG--CG-------------------CUCCAAAUGCA .((((((((.(((((......(((((((((((....)))))..))))))(.(((((((..((((....))))..))))))-).).)--))-------------------)).)))))))) ( -37.80) >DroMoj_CAF1 293756 98 - 1 UUGCAUUUGUGGCGCAAUUUAUAUGCAGCGAUUGCAAUUGCAAUGCGUGGUGCACAAAAUGCAUACAAAUGUAAUUUGUG-CACAG--CG-------------------CUUCGAAUGCA .((((((((.(((((......(((((((((((....)))))..))))))(((((((((.(((((....))))).))))))-))).)--))-------------------)).)))))))) ( -45.50) >DroPer_CAF1 215411 112 - 1 UUGCAUUUGCGGCGCAAUUUAUAUGCAA-----GCGAUUGCAAUGCGU---GCACAAAACGCAUACAAAUGUAAUUUGCAGACGAGCGCGCGGAUUCCUCAUGCUCUUGCCCCAAAUGCA .((((((((.(((((((.((((((((((-----....)))).((((((---.......))))))....)))))).))))....(((((.(.((...)).).)))))..))).)))))))) ( -42.50) >consensus UUGCAUUUGCGGCGCAAUUUAUAUGCAA_____GCAAUUGCAAUGCGU___GCACAAAACGCAUACAAAUGUAAUUUGCA_ACGAGCGCG___________________CUCCAAAUGCA .((((((((.((.(((.......))).......(((((((((((((((..........)))))).....))))).))))..............................)).)))))))) (-20.33 = -20.58 + 0.25)

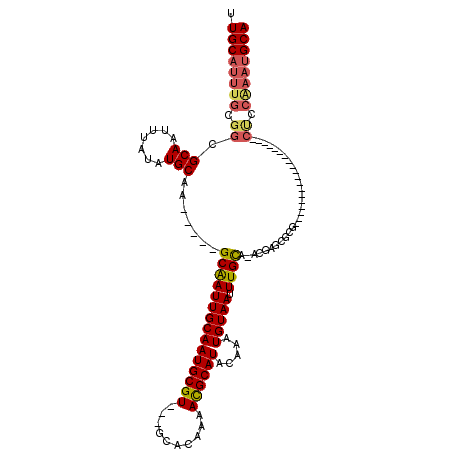
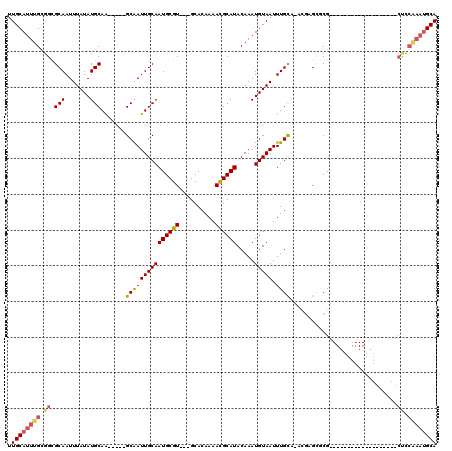
| Location | 18,336,732 – 18,336,831 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.68 |
| Mean single sequence MFE | -27.87 |
| Consensus MFE | -15.58 |
| Energy contribution | -15.08 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.16 |
| Mean z-score | -2.25 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.46 |
| SVM RNA-class probability | 0.743651 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18336732 99 + 22407834 UGCAAAUUACAUUUGUAUGCGUUUUGUGC---ACGCAUUGCAAUCGC-----UUGCAUAUAAAUUGCGCCGCAAAUGCAAAGCCACAAC--AAUUGUUG-----CC--UCC----GUACG (((......(((((((..((((..(((((---(.((.........))-----.))))))......)))).)))))))........((((--....))))-----..--...----))).. ( -22.10) >DroVir_CAF1 273547 113 + 1 CACAAAUUACAUUUGUAUGCGUUUUGUGCAGCACGCAUUGCAAUUGCAAUCGCUGCAUAUAAAUUGCGCCACAAAUGCAUAGCCACAAC--AAUUGUGG-----AGCUGUCCCUCAGCCC .........(((((((..((((..((((((((....(((((....))))).))))))))......)))).))))))).....(((((..--...)))))-----.((((.....)))).. ( -36.60) >DroPse_CAF1 213099 106 + 1 UGCAAAUUACAUUUGUAUGCGUUUUGUGC---ACGCAUUGCAAUCGC-----UUGCAUAUAAAUUGCGCCGCAAAUGCAAAGCCACAACACAAUUGUUGCCUUGCC--UCC----AUGCG .(((......((((((..((((..(((((---(.((.........))-----.))))))......)))).))))))((((.((.((((.....)))).)).)))).--...----.))). ( -27.70) >DroGri_CAF1 222917 107 + 1 CACAAAUUACAUUUGUAUGCGUUUUGUGCACCACGCAUUGCAAUUGCAAUCGCUGCAUAUAAAUUGCACCGCAAAUGCAAAGCCACAAC--AAUUGGUU-----GG--UCC----GCCCC .........(((((((.((((...((((((...((.(((((....))))))).)))))).....))))..)))))))...(((((....--...)))))-----((--...----.)).. ( -26.20) >DroMoj_CAF1 293774 109 + 1 CACAAAUUACAUUUGUAUGCAUUUUGUGCACCACGCAUUGCAAUUGCAAUCGCUGCAUAUAAAUUGCGCCACAAAUGCAAAGCCACAAU--AAUUGUGA-----GGCGGCC----ACCCC ..........(((((((((((....(((...)))(((((((....))))).)))))))))))))..((((......((...))((((..--...)))).-----))))...----..... ( -26.90) >DroPer_CAF1 215451 106 + 1 UGCAAAUUACAUUUGUAUGCGUUUUGUGC---ACGCAUUGCAAUCGC-----UUGCAUAUAAAUUGCGCCGCAAAUGCAAAGCCACAACACAAUUGUUGCCUUGCC--UCC----AUGCG .(((......((((((..((((..(((((---(.((.........))-----.))))))......)))).))))))((((.((.((((.....)))).)).)))).--...----.))). ( -27.70) >consensus CACAAAUUACAUUUGUAUGCGUUUUGUGC___ACGCAUUGCAAUCGC_____CUGCAUAUAAAUUGCGCCGCAAAUGCAAAGCCACAAC__AAUUGUUG_____CC__UCC____AUCCC .........(((((((.((((...((((.....)))).))))...........((((.......))))..)))))))........................................... (-15.58 = -15.08 + -0.50)
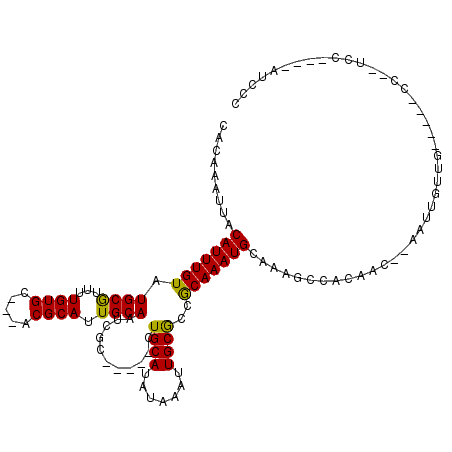
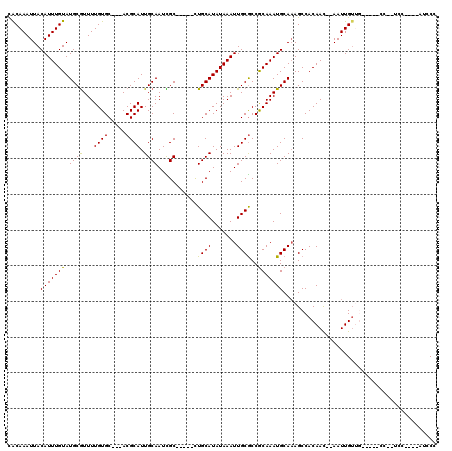
| Location | 18,336,732 – 18,336,831 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.68 |
| Mean single sequence MFE | -34.82 |
| Consensus MFE | -21.19 |
| Energy contribution | -20.75 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.63 |
| Structure conservation index | 0.61 |
| SVM decision value | 1.06 |
| SVM RNA-class probability | 0.908236 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18336732 99 - 22407834 CGUAC----GGA--GG-----CAACAAUU--GUUGUGGCUUUGCAUUUGCGGCGCAAUUUAUAUGCAA-----GCGAUUGCAAUGCGU---GCACAAAACGCAUACAAAUGUAAUUUGCA .(((.----((.--.(-----((((....--)))))..)).))).........((((.((((((((((-----....)))).((((((---.......))))))....)))))).)))). ( -29.30) >DroVir_CAF1 273547 113 - 1 GGGCUGAGGGACAGCU-----CCACAAUU--GUUGUGGCUAUGCAUUUGUGGCGCAAUUUAUAUGCAGCGAUUGCAAUUGCAAUGCGUGCUGCACAAAACGCAUACAAAUGUAAUUUGUG ((((((.....)))))-----)((((...--..))))....(((((((((((((.........((((((((((((....)))))...))))))).....))).))))))))))....... ( -37.14) >DroPse_CAF1 213099 106 - 1 CGCAU----GGA--GGCAAGGCAACAAUUGUGUUGUGGCUUUGCAUUUGCGGCGCAAUUUAUAUGCAA-----GCGAUUGCAAUGCGU---GCACAAAACGCAUACAAAUGUAAUUUGCA ((((.----...--.(((((((.(((((...))))).)))))))...))))..((((.((((((((((-----....)))).((((((---.......))))))....)))))).)))). ( -38.40) >DroGri_CAF1 222917 107 - 1 GGGGC----GGA--CC-----AACCAAUU--GUUGUGGCUUUGCAUUUGCGGUGCAAUUUAUAUGCAGCGAUUGCAAUUGCAAUGCGUGGUGCACAAAACGCAUACAAAUGUAAUUUGUG ((...----...--))-----.((((...--((((..(..(((((.((((..((((.......)))))))).))))))..))))...)))).((((((..((((....))))..)))))) ( -30.60) >DroMoj_CAF1 293774 109 - 1 GGGGU----GGCCGCC-----UCACAAUU--AUUGUGGCUUUGCAUUUGUGGCGCAAUUUAUAUGCAGCGAUUGCAAUUGCAAUGCGUGGUGCACAAAAUGCAUACAAAUGUAAUUUGUG .....----.(((((.-----.((((...--..))))((.(((((.(((..((((............))).)..))).))))).))))))).((((((.(((((....))))).)))))) ( -35.10) >DroPer_CAF1 215451 106 - 1 CGCAU----GGA--GGCAAGGCAACAAUUGUGUUGUGGCUUUGCAUUUGCGGCGCAAUUUAUAUGCAA-----GCGAUUGCAAUGCGU---GCACAAAACGCAUACAAAUGUAAUUUGCA ((((.----...--.(((((((.(((((...))))).)))))))...))))..((((.((((((((((-----....)))).((((((---.......))))))....)))))).)))). ( -38.40) >consensus CGGAU____GGA__CG_____CAACAAUU__GUUGUGGCUUUGCAUUUGCGGCGCAAUUUAUAUGCAA_____GCAAUUGCAAUGCGU___GCACAAAACGCAUACAAAUGUAAUUUGCA .......................(((((...))))).((.(((((((((....(((((.....(((.......)))))))).((((((..........)))))).)))))))))...)). (-21.19 = -20.75 + -0.44)

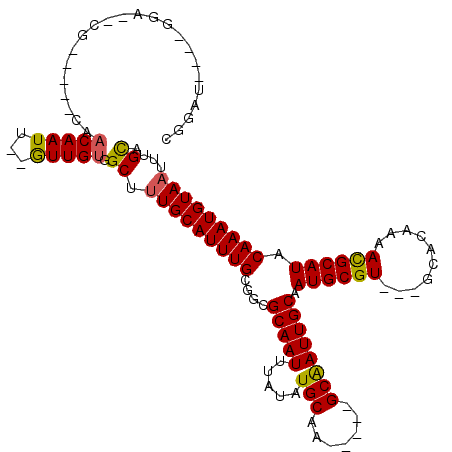
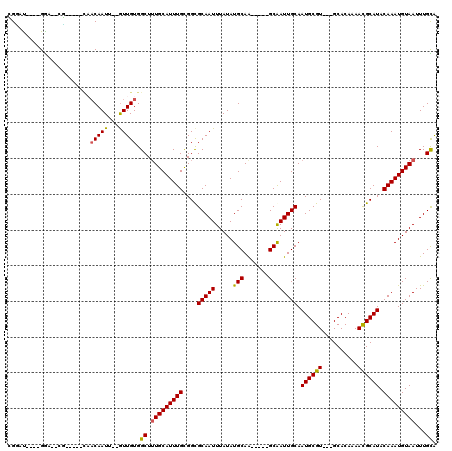
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:13:08 2006