
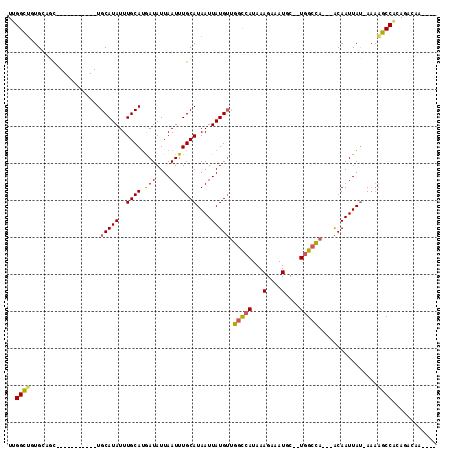
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 18,212,155 – 18,212,250 |
| Length | 95 |
| Max. P | 0.973842 |

| Location | 18,212,155 – 18,212,250 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 79.01 |
| Mean single sequence MFE | -26.77 |
| Consensus MFE | -14.37 |
| Energy contribution | -14.12 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.98 |
| Structure conservation index | 0.54 |
| SVM decision value | 1.72 |
| SVM RNA-class probability | 0.973842 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18212155 95 + 22407834 UUGGCUAUGGAAU-----------UGCAUAUUUGCAUGAUAUUAAUUUGCAUAAUUAUGUUGGUCAUAAAGAAAUGC--UGGCUA---ACAAUUAU-AAUGGCCACAGACAA---- .(((((((.....-----------((((....))))..............(((((..(((((((((...(....)..--))))))---))))))))-.))))))).......---- ( -22.90) >DroPse_CAF1 62607 110 + 1 UUGGCUGUGCUGCUGCUAUGCUGUUGCAUAUUUGCAUAAUAUUAAUUUGCAUAAUUAUGUUGGCCAUAAAGAAAUCCUCUGGCCAACAACAAUUAUGAAAAGCCGCA--CAA---- .....(((((.((((((((((....)))))...))..............(((((((.(((((((((...((.....)).)))))))))..)))))))...))).)))--)).---- ( -35.20) >DroEre_CAF1 55542 95 + 1 UUGGCUGUGGAGC-----------UGCAUAUUUGCAUGAUAUUAAUUUGCAUAAUUAUGUUGGCCAUAAAGAAAUGC--UAGCUA---ACAAUUAU-AAAGGCCACAGACAA---- .((.((((((..(-----------((((....)))(((((..(((((.....)))))((((((((((......))).--..))))---))))))))-..)).)))))).)).---- ( -21.90) >DroYak_CAF1 55369 95 + 1 UUGGCUCUGAAGC-----------UGCAUAUUUGCAUGAUAUUAAUUUGCAUAAUUAUGUUGGCCAUAAAGAAAUGC--UGGCUA---ACAAUUAU-AAAGGCCACAGACAA---- .((((.((.....-----------((((....))))..............(((((..(((((((((...(....)..--))))))---))))))))-..)))))).......---- ( -22.90) >DroAna_CAF1 51127 99 + 1 UCGGUACUGUCUC-----------UGCAUAUUUGCAGGAUAUUAAUUUGCAUAAUUAUGGCGACCAUAAAGAAAUGC--UGGCCA---GCAAUUAC-ACAAGCCACAAGCAAACAA ..(((..((((.(-----------((((....))))))))).....(((..(((((.((((.(.(((......))).--).))))---..))))).-.))))))............ ( -24.40) >DroPer_CAF1 63712 110 + 1 UUGGCUGUGCUGCUGCUAUGCUGUUGCAUAUUUGCAUAAUAUUAAUUUGCAUAAUUAUGUUGGCCAUAAAGAAAUCCUCUGGCCAAUAACAAUUAUGAAAAGCCGCA--CAA---- .....(((((.((((((((((....)))))...))..............(((((((.(((((((((...((.....)).)))))))))..)))))))...))).)))--)).---- ( -33.30) >consensus UUGGCUGUGCAGC___________UGCAUAUUUGCAUGAUAUUAAUUUGCAUAAUUAUGUUGGCCAUAAAGAAAUGC__UGGCCA___ACAAUUAU_AAAAGCCACAGACAA____ ..((((...................(((((..((((.(((....)))))))....))))).(((((.............)))))................))))............ (-14.37 = -14.12 + -0.25)


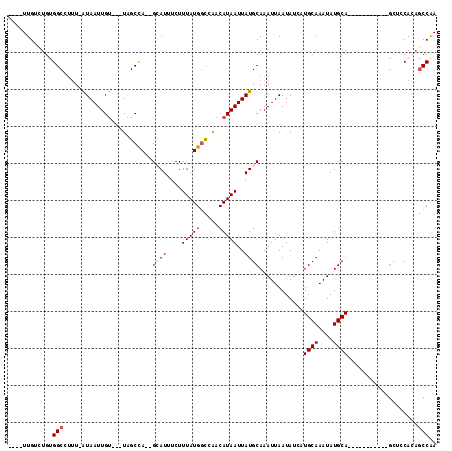
| Location | 18,212,155 – 18,212,250 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 79.01 |
| Mean single sequence MFE | -26.88 |
| Consensus MFE | -11.65 |
| Energy contribution | -12.15 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.24 |
| Structure conservation index | 0.43 |
| SVM decision value | 1.56 |
| SVM RNA-class probability | 0.964096 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18212155 95 - 22407834 ----UUGUCUGUGGCCAUU-AUAAUUGU---UAGCCA--GCAUUUCUUUAUGACCAACAUAAUUAUGCAAAUUAAUAUCAUGCAAAUAUGCA-----------AUUCCAUAGCCAA ----.((.((((((.....-.....(((---(((...--((((....(((((.....)))))..))))...))))))...((((....))))-----------...)))))).)). ( -18.20) >DroPse_CAF1 62607 110 - 1 ----UUG--UGCGGCUUUUCAUAAUUGUUGUUGGCCAGAGGAUUUCUUUAUGGCCAACAUAAUUAUGCAAAUUAAUAUUAUGCAAAUAUGCAACAGCAUAGCAGCAGCACAGCCAA ----(((--(((.(((((.(((((((((.((((((((((((....)))).))))))))))))))))).))).........(((...(((((....)))))))))).)))))).... ( -40.00) >DroEre_CAF1 55542 95 - 1 ----UUGUCUGUGGCCUUU-AUAAUUGU---UAGCUA--GCAUUUCUUUAUGGCCAACAUAAUUAUGCAAAUUAAUAUCAUGCAAAUAUGCA-----------GCUCCACAGCCAA ----.((.((((((.....-.....(((---(((...--((((....(((((.....)))))..))))...))))))...((((....))))-----------...)))))).)). ( -21.40) >DroYak_CAF1 55369 95 - 1 ----UUGUCUGUGGCCUUU-AUAAUUGU---UAGCCA--GCAUUUCUUUAUGGCCAACAUAAUUAUGCAAAUUAAUAUCAUGCAAAUAUGCA-----------GCUUCAGAGCCAA ----...((((.(((...(-((((((((---(.((((--...........)))).))))..)))))).............((((....))))-----------))).))))..... ( -20.00) >DroAna_CAF1 51127 99 - 1 UUGUUUGCUUGUGGCUUGU-GUAAUUGC---UGGCCA--GCAUUUCUUUAUGGUCGCCAUAAUUAUGCAAAUUAAUAUCCUGCAAAUAUGCA-----------GAGACAGUACCGA ((((((((.....))((((-(((((((.---((((.(--.(((......))).).))))))))))))))).........(((((....))))-----------)))))))...... ( -26.20) >DroPer_CAF1 63712 110 - 1 ----UUG--UGCGGCUUUUCAUAAUUGUUAUUGGCCAGAGGAUUUCUUUAUGGCCAACAUAAUUAUGCAAAUUAAUAUUAUGCAAAUAUGCAACAGCAUAGCAGCAGCACAGCCAA ----(((--(((.(((((.(((((((((..(((((((((((....)))).))))))).))))))))).))).........(((...(((((....)))))))))).)))))).... ( -35.50) >consensus ____UUGUCUGUGGCCUUU_AUAAUUGU___UAGCCA__GCAUUUCUUUAUGGCCAACAUAAUUAUGCAAAUUAAUAUCAUGCAAAUAUGCA___________GCUCCACAGCCAA ............(((........................((((....(((((.....)))))..))))............((((....))))...................))).. (-11.65 = -12.15 + 0.50)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:12:19 2006